尽管人们的口腔卫生在近些年提高了,但是龋齿和牙周病仍然是最常见的牙齿疾病,几乎每个成年人都有。口腔细菌是引起龋齿的关键因素。在人的口腔中大约有1 000种细菌。口腔细菌和黏附在牙齿表面的唾液大分子如糖蛋白、淀粉酶 (Amylase)、凝集蛋白 (Agglutinin) 等相互作用在牙齿表面形成早期牙菌斑生物膜 (Dental plaque Biofilm),其中85%的早期细菌是绿色链球菌属 (Viridans streptococcus) 的4种细菌 (Streptococcus sanguinis, S.gordonii, S.oralis, S.mitis),5%是放线菌 (Actinomyces)[1-3]。早期牙菌斑中的细菌再与其他细菌 (比如Fusobacterium, Actinomyces, Veillonella) 等相互黏附凝结增强了生物膜中细菌的多样性,同时密切相关的细菌在牙齿表面大量生长最终导致生物膜的成熟[4-8]。牙菌斑生物膜中的细菌能够发酵唾液中的糖产生有机酸从而在牙齿表面形成一个酸性环境,导致牙釉质分解引起龋齿和牙周炎等疾病。
在不加外源葡萄糖时,链球菌属和放线菌属的细菌不能在唾液中单独生长[9]。但是,一个例外是S.gordoniiDL1能够在25%的唾液中生长到107 CFU/mL[10]。此外,还发现几个口腔细菌能利用1%的猪胃粘蛋白 (Mucin) 和0.5%的酸性糖蛋白 (α1-Acid glycoprotein) 生长[11-12]。细菌S.intermedius表面的多个糖苷酶和磷酸转移酶在粘蛋白MUCIN5B生长中发挥重要作用[13];S.oralis表面的唾液酸糖苷酶移走酸性糖蛋白N-糖链的唾液酸使细菌生长[14];此外,不同口腔细菌能够相互合作利用唾液粘蛋白共生[15]。这些发现表明在缺少外源葡萄糖的情况下,细菌的生长可能是由于细菌捕食唾液中糖链的结果,且与细菌的糖苷酶相关。但是很少知道细菌如何捕食特定的唾液大分子糖链,不同细菌如何相互喂养共生并影响牙菌斑生物膜的形成。
本综述总结了笔者对口腔细菌的研究,揭示细菌的糖苷酶能依次水解唾液糖蛋白的糖链,释放的单糖或糖链被细菌代谢,提供碳源和能源供细菌生长。不同细菌有不同的糖苷酶,可能会帮助它们在唾液中相互喂养得以共生。
1 唾液蛋白唾液蛋白的分泌是由神经系统控制,依赖于刺激物的刺激。首先唾液腺体中的基因转录翻译成唾液蛋白,然后进行翻译后修饰包括蛋白糖基化、磷酸化和蛋白裂解。因此唾液蛋白往往是一些结构密切相关的蛋白家族。此外,一旦唾液被分泌到并非无菌的口腔环境中,唾液蛋白将被进一步修饰,导致蛋白裂解、部分去糖基化及蛋白复合物的形成。唾液蛋白的修饰发生在一个唾液被分泌和吞咽的动态环境中,因此唾液蛋白组是高度变化的[16]。绝大多数唾液蛋白都是糖蛋白如淀粉酶,粘蛋白,凝集子,富含脯氨酸的唾液蛋白 (PRP) 和分泌型免疫球蛋白A (sIgA)。唾液中非糖基化的蛋白一般较小,如半胱氨酸蛋白酶抑制剂 (cystatin),富酪蛋白 (statherin) 和富组蛋白 (histatin)。唾液蛋白的糖基化即有N-糖基化也有O-糖基化[17-19]。比如淀粉酶上有Lewis x的糖链结构,除了催化淀粉水解之外,淀粉酶能结合口腔细菌S.gordonii、S.mitis和S.oralis,将它们黏附到牙齿表面形成早期的微生物菌膜[1, 20]。而富含脯氨酸的唾液蛋白 (PRB) 是腮腺分泌最多的糖蛋白,分为碱性PRB和酸性PRB,它们的分子量从39 kDa到89 kDa[20-21]。PRB富含糖链因此被称为富含脯氨酸的糖蛋白 (PRG),几乎50%的PRG重量来自糖链,而且绝大多数是N-糖链[22]。通过NMR和质谱解析了PRG的27种N-糖链结构,都富含岩藻糖,也有一些含有唾液酸单糖[23]。PRG有Lewis x和Lewis y的糖链结构,能结合各种细菌包括F.nucleatum, S.mitis和S.sanguini[23-26]。而粘液糖蛋白 (MG1/MG2) 由颌下腺和舌下腺大量分泌。MG2大约68%的重量来自糖链,且O-糖链众多,已经鉴定有41个不同的糖链结构。MG2能表达多种糖链结构如唾液酸标记的T-抗原、Lewis x (sialy-Lewis x) 和Lewis x。这些结构是细菌识别MG2的特异配体,导致细菌粘在一起并被清除,因此粘液糖蛋白被认为是先天的口腔免疫防卫系统[20, 27]。粘液糖蛋白中的MG1是糖基化最多的唾液蛋白,78%的重量是糖链,而且这些糖链中29%都含有唾液酸,19%被硫酸化[28]。MG1的大量O-糖链使其呈现出可溶性胶的状态,在早期牙菌斑菌膜中介于牙齿和细菌之间[29-30]。但是,尽管MG1含有大量糖链,它能结合的细菌要比MG2少得多[31]。另外,免疫球蛋白sIgA重链上的N-糖链含有各种Lewis糖链结构,因此sIgA能通过重链结合大量细菌并参与先天和后天免疫[32-33]。
2 口腔细菌的糖苷酶糖苷酶 (Glycoside hydrolase, GH) 能够水解单糖之间或糖与非糖基团之间的糖苷键。糖苷酶根据其底物特异性和催化的分子机制命名,而编码糖苷酶的基因在基因组上大量存在。根据氨基酸序列的相似性,糖苷酶分为不同的家族 (Glycoside hydrolase family),在CAZy (Carbohydrate-Active Enzymes database) 数据库中,GH有135个家族 (GH1到GH135)。因为蛋白的折叠比氨基酸序列更加保守,所以根据糖苷酶催化活性中心结构域的结构,又可将部分家族归纳为14个族 (Clan),从GH-A到GH-N。同一Clan中的不同GH家族水解的底物可能不同,但是它们的结构、催化的氨基酸和催化机制高度相似,亲缘关系更近。此外,有的糖苷酶含有不同GH家族的催化域,具有水解多种糖苷键的功能。糖苷酶又被分为内切糖苷酶 (endo-glycosidase) 和外切糖苷酶 (exo-glycosidase)。前者是从糖链的中间水解糖苷键,后者是从底物的末端非还原端水解糖苷键。
许多口腔细菌都有糖苷酶,有些糖苷酶的结构及其底物的特异性已经被鉴定。绿色链球菌属能以人血清的α1-酸性糖蛋白 (AGP) 作为唯一碳源生长,其中S.defective能产生唾液酸糖苷酶 (Sialidase) 水解AGP糖链末端的唾液酸单糖露出相邻的N-乙酰葡萄糖。而S.intermedius能产生β-半乳糖苷酶和β-N-乙酰葡萄糖苷酶,从N-糖链上切下N-乙酰葡萄糖和半乳糖[13, 34]。S.oralis能产生β-半乳糖苷酶、α-岩藻糖苷酶和甘露糖苷酶,它们水解N-糖链上的岩藻糖和甘露糖,最后只留下N-乙酰葡萄糖挂在AGP肽链的天冬氨酸上,释放的单糖除了岩藻糖外都被用于细菌的生长[12]。S.gordoniiV288产生岩藻糖苷酶,属于GH29,能水解α1-3/4-岩藻糖[35]。S.intermediusPC574是从人的牙菌斑中分离的兼性厌氧菌,与大脑内脓肿感染相关。从该菌中发现了一个新的分泌型糖苷酶MsgA,该酶被鉴定有多个糖苷酶的活性,其LacZ区域有β-半乳糖苷酶和β-岩藻糖苷酶的活性,GH20区域有β-N-乙酰葡萄糖苷酶和β-N-乙酰半乳糖苷酶的活性,纯化的MsgA能够水解人的α1-抗胰蛋白酶抗体上的糖链[36]。唾液酸糖苷酶是最关键的糖苷酶,位于糖链最末端的唾液酸对糖链起保护作用,只有释放出唾液酸后,内部的糖链结构才能被其他糖苷酶识别,或产生新的能被细菌识别的糖链结构。比如当用唾液酸糖苷酶处理黏蛋白后,它失去了与S.sangunis黏附的能力,但是仍然能与S.mutans黏附[37-38]。
链球菌 (Streptococcus) 和放线菌 (Actinomyc-es) 是黏附在牙齿表面的最早期的细菌,它们主要结合唾液粘蛋白和富含脯氨酸的唾液蛋白。通过人口腔微生物基因组数据库 (Human Oral Microbiome Database) 进行分析,并根据蛋白N-端信号肽和C-端分选结构域[39],我们对几个牙菌斑早期细菌表面的糖苷酶进行了总结 (表 1)。这些糖苷酶分布在9个GH家族中。GH2家族的糖苷酶具有β-半乳糖苷酶的活性;GH3家族的糖苷酶有的是β-葡萄糖苷酶,有的是β-N-乙酰半乳糖苷酶,也有的是α-L-阿拉伯糖水解酶;GH20家族有β-N-乙酰半乳糖苷酶或β-N-乙酰葡萄糖苷酶的活性;GH29家族都是α-L-岩藻糖苷酶;GH33家族多为唾液酸糖苷酶;GH84家族多为N-乙酰葡萄糖苷酶;GH85家族是内切的N-乙酰葡萄糖苷酶;GH95是另外一个α-L-岩藻糖苷酶家族;GH101是内切的N-乙酰半乳糖苷酶。细菌菌株S.oralis Uo5和S.mitis B6细胞表面有多个糖苷酶,模式菌株S.gordoniiDL1细胞表面有3个糖苷酶但是没有唾液酸糖苷酶。而链球菌S.sanguinisSK36和放线菌A.naeslundiiMG1细胞表面只有一个糖苷酶。
| 表 1 牙菌斑生物膜中早期细菌细胞表面的糖苷酶 Table 1 Glycoside hydrolase of early colonizer in dental plaque biofilm |
我们通过qPCR (real-time quantitative PCR) 研究比较了32种口腔细菌在唾液中的生长[40]。这些细菌都在牙菌斑早期生物膜中出现,分布在链球菌属 (Streptococcus),放线菌属 (Actinomyces),兼性双球菌属 (Gemella),颗粒链菌属 (Granulicatella),营养缺陷菌属 (Abiotrophia) 和罗思氏菌属 (Rothia) 等16个属。首先通过咀嚼封口膜来刺激人分泌唾液,收集唾液并用1.25 mmol/L二硫苏糖醇在冰上处理10 min,接着离心收集上清并在70℃作用30 min灭活唾液中的内源糖苷酶,然后将唾液转入透析袋中用缓冲液透析除去小分子物质,过滤除菌,最后获得用于培养细菌的唾液。细菌被接种到培养基中过夜培养后,用唾液重悬细菌并以1‰的接种量转接到2 mL新鲜的唾液中,在37℃,5% CO2中过夜培养,然后再以1‰的接种量转接到2 mL新鲜的唾液中培养24 h获得二次培养物,同时以没有接种细菌的唾液作为对照。取适量二次培养物离心收集细菌,用含有变溶菌素、溶菌酶和Triton-X100的缓冲液裂解细菌后提取基因组DNA,然后根据每个细菌的16S rRNA基因设计引物并用qPCR扩增目的片段,最后用10倍稀释的细菌基因组做标准曲线,根据标准曲线计算唾液中细菌量,结果如图 1所示。我们发现只有大约1/3的细菌能够单独在唾液中生长,其中形成牙菌斑生物膜的早期细菌S.oralis、S.gordonii和S.mitis在唾液中生长最好,在培养24 h后,细胞数都增加了100~1 000倍;细菌S.mutans、Granulicatella elegans和Genella haemolysans的生长增加了10~100倍;而其他细菌如Actinomycesspp.、Abiotrophia defective、Rothiaspp.、S.salivarius和S.sanguinis等几乎没有生长或不能生长。
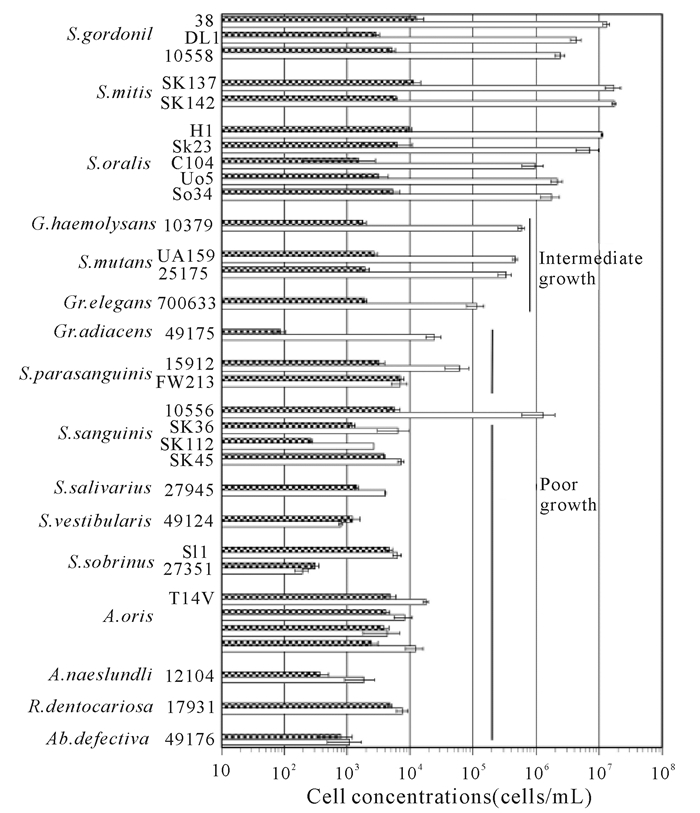
|
用qPCR检测细菌细胞浓度 (cell/mL),黑色柱子表示接种的细菌量,白色柱子表示相应细菌在唾液中生长24 h后的细菌量 Cell concentrations were measured by qPCR immediately after inoculation (hatched bar) and after 24 h in cubation at 37℃ (open bars).Error bars show the±standard deviation (n=3) 图 1 口腔细菌在唾液中的生长 Fig.1 Growth of oral bacteria in secondary cultures of whole saliva |
为了揭示其中的原因,我们检查了唾液蛋白的变化。将细菌生长后的唾液蛋白用SDS-PAGE胶分离后银染,发现生长最好的细菌培养液中,60~80 kDa的唾液蛋白迁移到30~50 kDa处。因为凝集素能识别糖链,不同的凝集素特异识别不同的糖链结构,所以我们将蛋白转移到硝酸纤维素膜上并与不同的凝集素杂交。结果发现这些唾液糖蛋白失去了糖链,而且生长得越好的细菌失去的糖链越多,因为它们同时不再与多个凝集素反应;而不能生长的细菌如放线菌属和链球菌属的某些细菌,其所在唾液的唾液蛋白和所有凝集素都反应,表明蛋白糖链没有任何改变。表 1中揭示S.mitisB6、S.oralisUo5和S.gordoniiDL1有多个位于细胞表面的糖苷酶,而它们在唾液中生长良好,但是S.sanguinisSK36和A.naeslundiiMG1只有1个糖苷酶,它们在唾液中几乎不能生长,因此细菌表面的糖苷酶与细菌捕食唾液糖链密切相关。另一方面,放线菌菌株A.naeslundii T14V细胞表面没有糖苷酶,该菌单独在唾液中也不能生长,但是当与链球菌菌株S.oralis34共同培养时,它们在25%的唾液中共同生长形成生物膜[10]。目前,细菌表面的糖苷酶如何利用唾液糖蛋白帮助细菌生长还不清楚,为此我们做了深入研究。
4 细菌捕食唾液糖蛋白的糖链S.gordonii DL1是牙菌斑生物膜中的优势细菌,在唾液中生长良好。我们以S.gordoniiDL1为研究材料,揭示细菌表面的糖苷酶如何捕食唾液糖链[41]。通过CAZy的注释,我们发现这个细菌有31个推测的糖苷酶 (GH),分布在16个GH家族。基于每个家族的酶的特殊活性,我们推测至少有17个糖苷酶可能参与细菌对唾液糖链的捕食。有3个糖苷酶位于细胞表面 (表 1),而且它们分别是肺炎链球菌β-半乳糖苷酶 (BgaA)[42-43]、N-乙酰葡萄糖苷酶 (StrH)[44-45]和内切己糖糖苷酶 (EndoD)[46]的同源蛋白。BgaA和StrH在肺炎链球菌中能够作用于α1-酸性糖蛋白的糖链,启动肺炎链球菌在人血清中的生长[47-49]。因此假设S.gordoniiDL1表面的糖苷酶BgaA、StrH和Endo对于捕食唾液大分子的糖链是关键的,敲除每个糖苷酶基因,同时也获得双基因敲除和3个基因敲除的突变株,然后将每个糖苷酶的催化功能域进行表达、纯化并耦联血蓝蛋白后和Freund佐剂一起注射兔子获得抗血清。通过抗血清鉴定,发现野生菌株表面有3个糖苷酶表达,而各突变菌株失去了相应糖苷酶的表达。酶活性鉴定发现,敲除BgaA的突变株对合成底物2-pNp-β-Gal的水解减少,敲除StrH的突变株对2-pNp-β-GlcNAc的水解减少,表明BgaA是β-半乳糖苷酶基因而StrH是N-乙酰葡萄糖苷酶基因。敲除这些糖苷酶基因后并没有影响该突变株在含有葡萄糖的THB培养基上的生长,但是导致该突变株在唾液中的生长量大幅减少 (图 2a)。3个糖苷酶基因都被敲除后,突变菌株在唾液中的生长量比野生菌株减少了约95%,单个糖苷酶基因或两个基因被敲除的突变株的生长量也减少了40%~70%。而且突变菌株在流动的唾液中形成的生物膜量也比野生菌株减少了75%。这些结果表明,细胞表面的糖苷酶是S.gordoniiDL1在唾液中达到最大量生长所必需的。

|
Sc是不接种细菌的唾液作为对照,将70 kDa的蛋白带切下来用Nano LC-MS/MS鉴定。所有菌株糖苷酶的基因型被列出,+表示有相应的糖苷酶,—表示没有相应的糖苷酶 Filtered saliva from un-inoculated control cultures (Sc), the 70 kDa band was excised (white box) from un-inoculated control saliva for Nano LC-MS/MS, GH genotypes of all strains are indicated at the bottom of panel 图 2 野生株和各个突变株在唾液中的生长 (a) 及通过SDS-PAGE分离蛋白并银染后比较唾液蛋白的特征 (b) Fig.2 Growth of S.gordonii DL1 wild type and △GH mutant strains measured as genomes/ml by qPCR of saliva cultures (a) and cultures of S.gordonii DL1 wild type or △GH mutant strains are compared by SDS PAGE followed by silver staining (b) |
分析唾液总蛋白,发现在野生菌株生长的唾液中,位于70 kDa处的蛋白消失并迁移到40~65 kDa处,在单个糖苷酶被敲除的突变菌株生长的唾液中也向下迁移,但是在失去多个糖苷酶的突变菌株生长的唾液中没有改变 (图 2b),这个结果进一步被野生菌株和3个糖苷酶被敲除的突变株的唾液总蛋白的二维电泳证实,这个蛋白位于等电点8~10处,是一个碱性蛋白。用Nano LC-MS/MS鉴定,揭示它是富含脯氨酸的唾液蛋白PRB3或PRB4[50]。
PRB蛋白含有很多的N-糖链和少量的O-糖链[51-52],其中PRB3有27个结构不同的N-糖链,绝大多数含有岩藻糖不含有唾液酸[23]。为了检测PRG上糖链的改变,唾液蛋白被转移到硝酸纤维素膜上并与不同凝集素杂交 (图 3)。
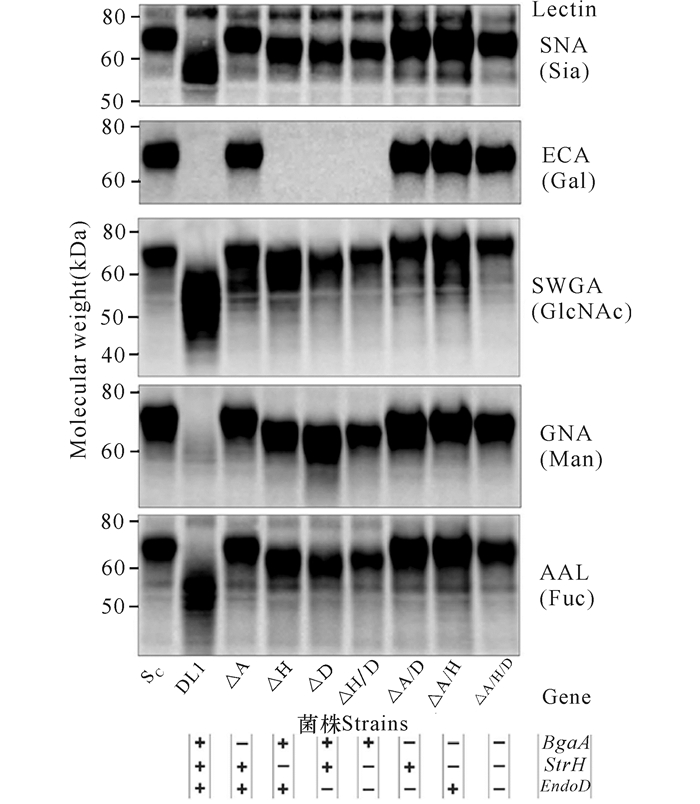
|
图 3 细菌唾液培养物中蛋白质与不同凝集素的反应,不接种细菌的唾液作为对照 (Sc) Fig.3 Lectin blots of saliva from un-inoculated control cultures (Sc), monocultures of S.gordonii DL1 and monocultures of △GH mutant strains.GH genotypes of all strains are indicated at the bottom of each panel.Lectin probes are listed on the right |
结果发现在野生菌株生长的唾液中,迁移到40~60 kDa处的PRG与凝集素SNA、SWGA和AAL反应,但是不能与凝集素ECA和GNA反应;表达BgaA的突变株PRG除了不能与ECA识别外,和其他凝集素都反应;而在所有失去BgaA的突变株生长的唾液中,PRG没有改变并和所有凝集素都反应;只有当3个糖苷酶都表达时,如野生株生长的唾液,PRG与GNA的反应才能消失。这个结果表明3个糖苷酶对唾液糖蛋白PRG上的N-糖链是依次水解 (图 4)。
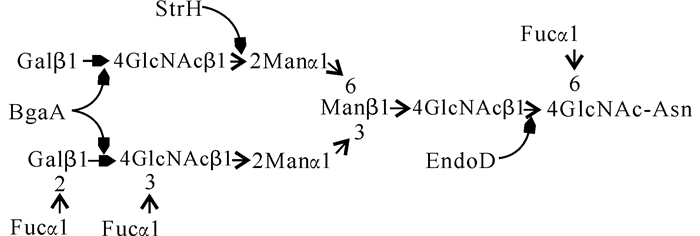
|
图 4 S.gordonii DL1的糖苷酶BgaA、StrH和EndoD对PRB3上N-糖链的顺序水解 Fig.4 Proposed sequential degradation of the major N-linked glycan of PRB3 by BgaA, StrH and EndoD of S.gordonii DL1 |
凝集素ECA能识别Galβ1-4GlcNAc和Fucα1-2Galβ1-4GlcNAc糖链结构[53-54],在表达BgaA的菌株中,β-半乳糖苷酶从末端水解掉Gal或者水解掉Fucα1-2Gal暴露出GlcNAcβ1-2Manα或者Fucα1-3GlcNAcβ1-2Manα,因此它们的唾液糖蛋白PRG都失去了与ECA的反应。而寡糖结构GlcNAcβ1-2Manα是StrH作用的底物[44],只有它暴露出来,StrH才能水解掉GlcNAc从而失去与特异识别GlcNAc的凝集素SWGA[55]的反应。EndoD是一个内切的糖苷酶,作用于N-糖链与氨基酸相连的GlcNAcβ1-4GlcNAc,但是EndoD对N-糖链的核心结构有严格的特异性[56]。BgaA和StrH从N-糖链上先后水解下Gal和GlcNAc暴露出甘露糖 (Man),而末端的α1-3Man是EndoD识别底物的关键[57],之后EndoD水解GlcNAcβ1-4GlcNAc的β1-4糖苷键,将高甘露糖核心结构释放出来,从而失去与能特异识别甘露糖的GNA的结合。相反,当细菌失去BgaA后,PRG的N-糖链最末端的半乳糖 (Gal) 不被水解下来,内部的糖链结构不能暴露出来,StrH和EndoD不能水解N-糖链,所以唾液蛋白PRG能被凝集素SWGA和GNA识别。尽管PRG蛋白绝大多数N-糖链都没有唾液酸 (Sia),但是在PRB的个别寡糖链末端有α2-6 Sia[23],因此解释了唾液PRG蛋白能与凝集素SNA反应。实际上,S.gordoniiDL1在唾液中生长并不影响SNA的识别,这表明末端的唾液酸能保护PRG的N-糖链阻止糖苷酶对糖链的水解。
我们的研究清楚地表明,S.gordonii DL1在唾液中的生长依赖于细胞表面的3个糖苷酶对唾液糖蛋白PRG的N-糖链依次水解,继而捕食释放的糖供细菌生长,这与肺炎链球菌中相应酶的底物特异性是一致的[47]。
5 展望唾液糖蛋白PRG也与凝集素AAL反应。AAL能识别α1-3和α1-6连接的岩藻糖[58],因此BgaA和EndoD的产物可能包括含有岩藻糖 (Fuc) 的寡糖。值得注意的是,S.gordoniiDL1不能在L-岩藻糖上生长[40],但是它的基因组上有两个推测的L-岩藻糖苷酶基因 (Sgo_0150和Sgo_1771)。我们表达了这两个蛋白,并且鉴定它们分别为α1-2-L-岩藻糖苷酶和α1-3/4-L-岩藻糖苷酶 (未发表)。我们推测蛋白酶Sgo_0150可能作用于从PRG上由BgaA水解下来的Fucα1-2Gal,而蛋白酶Sgo_1771和N-乙酰葡萄糖苷酶[59]及3个甘露糖苷酶位于一个gom操纵子[35]中,它们的功能可能是水解由EndoD释放的含有高甘露糖的寡糖链。实际上,当gom操纵子中的一些基因失去活性后,S.gordonii在α1-6-D-甘露二糖 (mannobiose) 和双糖GlcNAcβ1-2Man上的生长都减少[35]。我们推测,细菌表面糖苷酶水解下来的糖链可能会被细胞吸收,并由细胞内的其他糖苷酶进一步水解,释放的单糖参与细胞内的代谢,作为碳源和能源供细菌生长, 其中的机制还有待进一步深入研究。
我们的研究表明,糖苷酶对底物有严格的特异性,PRG糖链只能先被BgaA作用,释放出半乳糖后才能被StrH作用,暴露出EndoD识别底物后才能被EndoD从内部水解N-糖链的核心结构。有的细菌不能单独在唾液中生长,可能不是因为它的细胞表面没有糖苷酶,而是没有水解糖链末端糖的糖苷酶,比如有些糖链的末端有唾液酸单糖,能够封闭糖链,只有当唾液酸糖苷酶去除这个唾液酸后,糖链才能被其他糖苷酶进一步水解。不同的细菌表面有不同的糖苷酶,当不同的细菌被共同培养时,能够相互合作更好地水解唾液糖蛋白上的糖链,从而在唾液中相互喂养并在牙齿表面共生。细菌如何相互喂养,有哪些唾液蛋白参与以及如何影响牙菌斑生物膜的形成等问题还需要进一步探索。
| [1] |
SCANNAPIECO F A. Saliva-bacterium interactions in oral microbial ecology[J]. Crit Rev Oral Biol Med, 1994, 5(3/4): 203-248. |
| [2] |
YAO Y, BERG E A, COSTELLO C E, et al. Identification of protein components in human acquired enamel pellicle and whole saliva using novel proteomics approaches[J]. The Journal of Biological Chemistry, 2003, 278(7): 5300-5308. DOI:10.1074/jbc.M206333200 |
| [3] |
KOLENBRANDER P E, LONDON J. Adhere today, here tomorrow:Oral bacterial adherence[J]. J Bacteriol, 1993, 175(11): 3247-3252. DOI:10.1128/jb.175.11.3247-3252.1993 |
| [4] |
BRECX M, THEILADE J, ATTSTRÖM R. An ultrastr-uctural quantitative study of the significance of microbial multiplication during early dental plaque growth[J]. Journal of Periodontal Research, 1983, 18(2): 177-186. DOI:10.1111/jre.1983.18.issue-2 |
| [5] |
JAKUBOVICS N S. Saliva as the sole nutritional source in the development of multispecies communities in dental plaque[J]. Microbiol Spectr, 2015, 3(3): 1-11. DOI:10.1128/microbiolspec.MBP-0013-2014 |
| [6] |
DIAZ P I, CHALMERS N I, RICKARD A H, et al. Molecular characterization of subject-specific oral microflora during initial colonization of enamel[J]. Appl Environ Microbiol, 2006, 72(4): 2837-2848. DOI:10.1128/AEM.72.4.2837-2848.2006 |
| [7] |
DIGE I, NILSSON H, KILIAN M, et al. In situ identification of streptococci and other bacteria in initial dental biofilm by confocal laser scanning microscopy and fluorescence in situ hybridization[J]. Eur J Oral Sci, 2007, 115(6): 459-467. DOI:10.1111/eos.2007.115.issue-6 |
| [8] |
NYVAD B, KILIAN M. Microbiology of the early colonization of human enamel and root surfaces in vivo[J]. Eur J Oral Sci, 1987, 95(5): 369-380. DOI:10.1111/eos.1987.95.issue-5 |
| [9] |
DE JONG M H, VAN DER HOEVEN J S, VAN OS J H, et al. Growth of oral streptococcus species and actinomyces viscosus in human saliva[J]. Appl Environ Microbiol, 1984, 47(5): 901-904. |
| [10] |
PALMER R J JR, KAZMERZAK K, HANSEN M C, et al. Mutualism versus independence:Strategies of mixed-species oral biofilms in vitro using saliva as the sole nutrient source[J]. Infect Immun, 2001, 69(9): 5794-5804. DOI:10.1128/IAI.69.9.5794-5804.2001 |
| [11] |
WICKSTRÖM C, HAMILTON I R, SVENSÄTER G. Differential metabolic activity by dental plaque bacteria in association with two preparations of MUC5B mucins in solution and in biofilms[J]. Microbiology, 2009, 155(1): 53-60. DOI:10.1099/mic.0.022111-0 |
| [12] |
BYERS H L, TARELLI E, HOMER K A, et al. Grow-th of Viridans streptococci on human serum α1-acid glycoprotein[J]. J Dent Res, 1999, 78(7): 1370-1380. DOI:10.1177/00220345990780071201 |
| [13] |
HOMER K A, WHILEY R A, BEIGHTON D. Produ-ction of specific glycosidase activities by Streptococcus intermedius strain UNS35 grown in the presence of mucin[J]. J Med Microbiol, 1994, 41(3): 184-190. DOI:10.1099/00222615-41-3-184 |
| [14] |
BYERS H L, TARELLI E, HOMER K A, et al. Seque-ntial deglycosylation and utilization of the N-linked, complex-type glycans of human alpha1-acid glycoprotein mediates growth of Streptococcus oralis[J]. Glycobiology, 1999, 9(5): 469-479. DOI:10.1093/glycob/9.5.469 |
| [15] |
BRADSHAW D J, HOMER K A, MARSH P D, et al. Metabolic cooperation in oral microbial communities during growth on mucin[J]. Microbiology, 1994, 140(12): 3407-3412. DOI:10.1099/13500872-140-12-3407 |
| [16] |
HELMERHORST E J, OPPENHEIM F G. Saliva:A dynamic proteome[J]. J Dent Res, 2007, 86(8): 680-693. DOI:10.1177/154405910708600802 |
| [17] |
KORNFELD R, KORNFELD S. Assembly of asparagine-linked oligosaccharides[J]. Annu Rev Biochem, 1985, 54(1): 631-664. DOI:10.1146/annurev.bi.54.070185.003215 |
| [18] |
WEERAPANA E, IMPERIALI B. Asparagine-linked protein glycosylation:From eukaryotic to prokaryotic systems[J]. Glycobiology, 2006, 16(6): 91R-101R. DOI:10.1093/glycob/cwj099 |
| [19] |
TEN HAGEN K G, FRITZ T A, TABAK L A. All in the family:The UDP-GalNAc:Polypeptide N-acetylgalactosaminyltransferases[J]. Glycobiology, 2003, 13(1): 1R-16R. DOI:10.1093/glycob/cwg007 |
| [20] |
MURRAY P A, PRAKOBPHOL A, LEE T, et al. Adherence of oral streptococci to salivary glycoproteins[J]. Infect Immun, 1992, 60(1): 31-38. |
| [21] |
MAEDA N. Inheritance of the human salivary proline-rich proteins:A reinterpretation in terms of six loci forming two subfamilies[J]. Biochem Genet, 1985, 23(5/6): 455-464. |
| [22] |
REDDY M S, LEVINE M J, TABAK L A. Structure of the carbohydrate chains of the proline-rich glycoprotein from human parotid saliva[J]. Biochem Biophys Res Commun, 1982, 104(3): 882-888. DOI:10.1016/0006-291X(82)91331-6 |
| [23] |
GILLECE-CASTRO B L, PRAKOBPHOL A, BURLI-NGAME A L, et al. Structure and bacterial receptor activity of a human salivary proline-rich glycoprotein[J]. J Biol Chem, 1991, 266(26): 17358-17368. |
| [24] |
NAGATA K, NAKAO M, SHIBATA S, et al. Purification and characterization of galactosephilic component present on the cell surfaces of Streptococcus sanguis ATCC 10557[J]. J Periodontol, 1983, 54(3): 163-172. DOI:10.1902/jop.1983.54.3.163 |
| [25] |
LEVINE M J, REDDY M S, TABAK L A, et al. Structural aspects of salivary glycoproteins[J]. J Den Res, 1987, 66(2): 436-441. DOI:10.1177/00220345870660020901 |
| [26] |
RUHL S, SANDBERG A L, CISAR J O. Salivary receptors for the proline-rich protein-binding and lectin-like adhesins of oral actinomyces and streptococci[J]. J Den Res, 2004, 83(6): 505-510. DOI:10.1177/154405910408300614 |
| [27] |
PRAKOBPHOL A, THOMSSON K A, HANSSON G C, et al. Human low-molecular-weight salivary mucin expresses the sialyl Lewis determinant and has L-selectin ligand activity[J]. Biochemistry, 1998, 37(14): 4916-4927. DOI:10.1021/bi972612a |
| [28] |
LOOMIS R E, PRAKOBPHOL A, LEVINE M J, et al. Biochemical and biophysical comparison of two mucins from human submandibular-sublingual saliva[J]. Arch Biochem Biophys, 1987, 258(2): 452-464. DOI:10.1016/0003-9861(87)90366-3 |
| [29] |
OFFNER G D, TROXLER R F. Heterogeneity of high-molecular-weight human salivary mucins[J]. Adv Dent Res, 2000, 14(1): 69-75. DOI:10.1177/08959374000140011101 |
| [30] |
TABAK L A, LEVINE M J, JAIN N K, et al. Adsorption of human salivary mucins to hydroxyapatite[J]. Arch Oral Biol, 1985, 30(5): 423-427. DOI:10.1016/0003-9969(85)90070-6 |
| [31] |
PRAKOBPHOL A, BORÉN T, MA W G, et al. Highly glycosylated human salivary molecules present oligosaccharides that mediate adhesion of leukocytes and Helicobacter pylori[J]. Biochemistry, 2005, 44(6): 2216-2224. DOI:10.1021/bi0480180 |
| [32] |
FALK P, ROTH K A, BORÉN T, et al. An in vitro adherence assay reveals that Helicobacter pylori exhibits cell lineage-specific tropism in the human gastric epithelium[J]. P Natl Acad Sci USA, 1993, 90(5): 2035-2039. DOI:10.1073/pnas.90.5.2035 |
| [33] |
ROYLE L, ROOS A, HARVEY D J, et al. Secretory IgA N-and O-glycans provide a link between the innate and adaptive immune systems[J]. J Boil Chem, 2003, 278(22): 20140-20153. DOI:10.1074/jbc.M301436200 |
| [34] |
HOMER K A, ROBERTS G, BYERS H L, et al. Mannosidase production by viridans group streptococci[J]. J Clin Microbiol, 2001, 39(3): 995-1001. DOI:10.1128/JCM.39.3.995-1001.2001 |
| [35] |
KILIÇ A O, TAO L, ZHANG Y S, et al. Involvement of Streptococcus gordonii beta-glucoside metabolism systems in adhesion, biofilm formation, and in vivo gene expression[J]. J Bacteriol, 2004, 186(13): 4246-4253. DOI:10.1128/JB.186.13.4246-4253.2004 |
| [36] |
IMAKI H, TOMOYASU T, YAMAMOTO N, et al. Identification and characterization of a novel secreted glycosidase with multiple glycosidase activities in Streptococcus intermedius[J]. J Bacteriol, 2014, 196(15): 2817-2826. DOI:10.1128/JB.01727-14 |
| [37] |
MCBRIDE B C, GISSLOW M T. Role of sialic acid in saliva-induced aggregation of Streptococcus sangunis[J]. Infect Immun, 1977, 18(1): 35-40. |
| [38] |
LEVINE M J, HERZBERG M C, LEVINE M S, et al. Specificity of salivary-bacterial interactions:Role of terminal sialic acid residues in the interaction of salivary glycoproteins with Streptococcus sanguis and Streptococcus mutans[J]. Infect Immun, 1978, 19(1): 107-115. |
| [39] |
TON-THAT H, MARRAFFINI L A, SCHNEEWIND O. Protein sorting to the cell wall envelope of Gram-positive bacteria[J]. Biochim Biophys Acta, 2004, 1694(1/2/3): 269-278. |
| [40] |
ZHOU Y, YANG J H, ZHANG L X, et al. Differential utilization of basic proline-rich glycoproteins during growth of oral bacteria in saliva[J]. Appl Environ Microbiol, 2016, 82(17): 5249-5258. DOI:10.1128/AEM.01111-16 |
| [41] |
YANG J H, ZHOU Y, ZHANG L X, et al. Cell surface glycoside hydrolases of Streptococcus gordonii promote growth in saliva[J]. Appl Environ Microbiol, 2016, 82(17): 5278-5286. DOI:10.1128/AEM.01291-16 |
| [42] |
SINGH A K, PLUVINAGE B, HIGGINS M A, et al. Unravelling the multiple functions of the architecturally intricate Streptococcus pneumoniae β-galactosidase, BgaA[J]. PLoS Pathog, 2014, 10(9): e1004364. DOI:10.1371/journal.ppat.1004364 |
| [43] |
ZÄHNER D, HAKENBECK R. The Streptococcus pneumoniae beta-galactosidase is a surface protein[J]. J Bacteriol, 2000, 182(20): 5919-5921. DOI:10.1128/JB.182.20.5919-5921.2000 |
| [44] |
CLARKE V A, PLATT N, BUTTERS T D. Cloning and expression of the β-N-acetylglucosaminidase gene from Streptococcus pneumoniae:Generation of truncated enzymes with modified aglycon specificity[J]. J Biol Chem, 1995, 270(15): 8805-8814. DOI:10.1074/jbc.270.15.8805 |
| [45] |
JIANG Y L, YU W L, ZHANG J W, et al. Structural basis for the substrate specificity of a novel β-N-acetylhexosaminidase StrH protein from Streptococcus pneumoniae R6[J]. J Biol Chem, 2011, 286(50): 43004-43012. DOI:10.1074/jbc.M111.256578 |
| [46] |
ABBOTT D W, MACAULEY M S, VOCADLO D J, et al. Streptococcus pneumoniae endohexosaminidase D, structural and mechanistic insight into substrate-assisted catalysis in family 85 glycoside hydrolases[J]. J Biol Chem, 2009, 284(17): 11676-11689. DOI:10.1074/jbc.M809663200 |
| [47] |
KING S J. Pneumococcal modification of host sugars:A major contributor to colonization of the human airway[J]. Mol Oral Microbiol, 2010, 25(1): 15-24. DOI:10.1111/omi.2010.25.issue-1 |
| [48] |
KING S J, HIPPE K R, WEISER J N. Deglycosylation of human glycoconjugates by the sequential activities of exoglycosidases expressed by Streptococcus pneumoniae[J]. Mol Microbiol, 2006, 59(3): 961-974. DOI:10.1111/mmi.2006.59.issue-3 |
| [49] |
BURNAUGH A M, FRANTZ L J, KING S J. Growth of Streptococcus pneumoniae on human glycoconjugates is dependent upon the sequential activity of bacterial exoglycosidases[J]. J Bacteriol, 2008, 190(1): 221-230. DOI:10.1128/JB.01251-07 |
| [50] |
LYONS K M, AZEN E A, GOODMAN P A, et al. Many protein products from a few loci:Assignment of human salivary proline-rich proteins to specific loci[J]. Genetics, 1988, 120(1): 255-265. |
| [51] |
CARPENTER G H, PROCTOR G B. O-linked glycosylation occurs on basic parotid salivary proline-rich proteins[J]. Oral Microbiol Immunol, 1999, 14(5): 309-315. DOI:10.1034/j.1399-302X.1999.140507.x |
| [52] |
OHO T, RAHEMTULLA F, MÅNSSON-RAHEMT-ULLA B, et al. Purification and characterization of a glycosylated proline-rich protein from human parotid saliva[J]. Int J Biochem, 1992, 24(7): 1159-1168. DOI:10.1016/0020-711X(92)90387-G |
| [53] |
BLIXT O, HEAD S, MONDALA T, et al. Printed covalent glycan array for ligand profiling of diverse glycan binding proteins[J]. P Natl Acad Sci USA, 2004, 101(49): 17033-17038. DOI:10.1073/pnas.0407902101 |
| [54] |
TENEBERG S, ANGSTRÖM J, JOVALL P A, et al. Characterization of binding of Gal beta 4GlcNAc-specific lectins from Erythrina cristagalli and Erythrina corallodendron to glycosphinogolipids.Detection, isolation, and characterization of a novel glycosphinglipid of bovine buttermilk[J]. J Biol Chem, 1994, 269(11): 8554-8563. |
| [55] |
MONSIGNY M, ROCHE A C, SENE C, et al. Sugar-lectin interactions:How does wheat-germ agglutinin bind sialoglycoconjugates[J]. Eur J Biochem, 1980, 104(1): 147-153. DOI:10.1111/ejb.1980.104.issue-1 |
| [56] |
KOIDE N, MURAMATSU T. Endo-β-N-acetylglucosaminidase acting on carbohydrate moieties of glycoproteins purification and properties of the enzyme from Diplococcus pneumoniae[J]. J Biol Chem, 1974, 249(15): 4897-4904. |
| [57] |
TAI T, YAMASHITA K, OGATA-ARAKAWA M, et al. Structural studies of two ovalbumin glycopeptides in relation to the endo-beta-N-acetylglucosaminidase specificity[J]. J Biol Chem, 1975, 250(21): 8569-8575. |
| [58] |
GOLDSTEIN I J, HAMMARSTRÖM S, SUNDBLAD G. Precipitation and carbohydrate-binding specificity studies on wheat germ agglutinin[J]. Biochim Biophys Acta, 1975, 405(1): 53-61. DOI:10.1016/0005-2795(75)90313-X |
| [59] |
LANGLEY D B, HARTY D W S, JACQUES N A, et al. Structure of N-acetyl-β-D-glucosaminidase (GcnA) from the endocarditis pathogen Streptococcus gordonii and its complex with the mechanism-based inhibitor NAG-thiazoline[J]. J Mol Biol, 2008, 377(1): 104-116. DOI:10.1016/j.jmb.2007.09.028 |
 2017, Vol. 24
2017, Vol. 24 


