2. 广西科学院,国家非粮生物质能源工程技术研究中心,非粮生物质酶解国家重点实验室,广西生物炼制重点实验室,广西南宁 530007
2. National Engineering Research Center for Non-food Biorefinery, State Key Laboratory of Non-food Biomass and Enzyme Technology, Guangxi Key Laboratory of Biorefinery, Guangxi Academy of Sciences, Nanning, Guangxi, 530007, China
【研究意义】α-淀粉酶 (α-1, 4-糖苷-D-糖苷水解酶,EC 3.2.1.1) 是一类对淀粉和其他多聚糖内部的α-1, 4-糖苷键进行随机切割产生不同长度寡糖的酶类[1]。淀粉是地球上重要的能源和碳源物质,淀粉酶是仅次于蛋白酶的应用最广泛的酶类之一,占整个酶制剂市场的30%[2]。淀粉酶可以从植物、动物和微生物等多样的生物体中获得。微生物来源的淀粉酶,特别是芽孢杆菌来源的淀粉酶,往往因具有较高的热稳定性而被广泛应用在工业生产上[3]。热稳定性高的酶,由于可以耐受较高的反应温度,一方面可以减少反应过程中微生物的污染,另一方面通过降低反应底物的粘度,可增加底物和产物的溶解度、提高底物产物的扩散系数,进而提高反应速率,增加产物得率等[4]。此外,耐热酶有助于人们认识蛋白质对高热环境的适应特征和耐热机理,在蛋白质工程中具有重要作用。【前人研究进展】嗜热环境中的微生物,具有与高温生境相适应的细胞组分和代谢产物,是耐热酶的主要天然来源[5]。耐热α-淀粉酶的产生菌主要有Geobacillus stearothermophilus,Bacillus subtilis,Bacillus licheniformis,Anoxybacillus,Bacillus amyloliquefaciens,Bacillus cereus,Bacillus globisporus和Bacillus alvei等[6]。Anoxybacillussp.主要分布于地热火山温泉、堆肥等嗜热生境中,适宜生长温度为45~65℃,pH值为6~10,属于中等嗜热碱性菌[7],最早由Heinen等[8]在新西兰北部的温泉中发现,命名为Bacillus flavothermus,2000年时Anoxybacillus首次作为新的种属报道[9]。近年来,Anoxybacillussp.作为耐热葡萄糖异构酶、耐热核酮糖激酶、耐热木糖酶等的产生菌被陆续发现和报道[10-12]。【本研究切入点】相对于其它已进行了大量研究的α-淀粉酶,来源于Anoxybacillus sp.的α-淀粉酶的研究仍很有限。【拟解决的关键问题】从云南腾冲火山温泉土壤中分离筛选产耐热α-淀粉酶的Anoxybacillus sp.菌株,并从中克隆耐热α-淀粉酶基因,然后在大肠杆菌中进行异源表达,最后对该重组酶进行酶学性质分析。
1 材料与方法 1.1 材料 1.1.1 土样、菌株及质粒土样:采集自云南腾冲火山温泉土壤。
菌株及质粒:大肠杆菌DH5α购自Invitrogen公司,大肠杆菌M15、质粒pQE30购自Qiagen公司。
1.1.2 培养基及碘液配方富集培养基:蛋白胨 (1%)、淀粉 (0.1%)、Na2HPO4(25 mmol/L)、KH2PO4(25 mmol/L)、NH4Cl (25 mmol/L)、MgSO4(2 mmol/L)。
淀粉酶筛选培养基:M9培养基加上可溶性淀粉 (0.1%)。
鲁格氏碘液 (300 mL):1 g I2,2 g KI。
1.1.3 酶、试剂及主要仪器设备实验所用到的各种限制性内切酶、T4 DNA连接酶、ExTaq酶、LA酶、pMD18T-Vector克隆试剂盒、基因组步移试剂盒等购自Takara公司,基因组DNA提取试剂盒、质粒提取试剂盒、DNA纯化回收试剂盒等购自Tiangen公司,蛋白纯化用Ni-NTA购自Qiagen公司,脱盐柱HiTrap Desalting G-25、阴离子柱Hiload 16/10 Q Sepharose High Performance、分子筛柱SuperdexTM 75 10/300GL等购自GE公司,引物由上海生工生物工程公司合成,其它试剂均为化学分析纯。梯度PCR仪为Biometra公司的TProfessional Thermocycler,凝胶成像仪为Bio-Rad公司的Gel DocTM XR+,蛋白层析仪为GE公司的ÄKTA purifier,核酸蛋白分析仪为ThermoFisher公司的NanoDrop 2000c Spectrophotometer。
1.2 耐热α-淀粉酶产生菌的分离和鉴定称取自然风干土样1 g加入100 mL富集培养基,37℃、220 r/min培养24 h,取1 mL培养液,以无菌生理盐水为稀释液,进行10-1、10-2、10-3、10-4、10-5的连续梯度稀释,涂布于淀粉酶筛选固体平板,置于恒温培养箱60℃培养48~72 h,待平板长出1 mm左右大小的菌落后,在平板表面滴加鲁格氏碘液,挑选水解圈明显的菌株进行菌种鉴定。菌株基因组DNA的提取按试剂盒说明书操作,选取原核生物16S rDNA的通用引物27F:5′-AGAGTTTGATCCTGGCTCA-3′,1492R:5′-GGTTACCTTGTTACGACTT-3′,用ExTaq酶进行PCR扩增,扩增产物经凝胶回收并纯化,将纯化产物与pMD-18T载体连接,转化DH5α感受态细胞,挑取转化子进行菌体电泳后,挑选验证正确的转化子送上海生工生物工程公司测序。根据测序结果从NCBI数据库中选取近缘菌株的16S rDNA基因序列,采用Clustal Omega (http://www.ebi.ac.uk/Tools/msa/clustalo)[13]进行多序列比对分析和系统进化树构建,以确定该菌株的分类地位。
1.3 耐热α-淀粉酶基因的克隆 1.3.1 基因序列比较和蛋白质分析使用NCBI的BLAST (http://blast.ncbi.nlm.nih.gov/Blast.cgi) 进行基因序列比对,ORF Finder (http://www.ncbi.nlm.nih.gov/gorf/gorf.html) 进行基因开放阅读框预测,BPROM程序 (http://linux1.softberry.com/berry.phtml)[14]用于启动子区预测,CAZY数据库 (http://www.cazy.org)[15]进行糖苷水解酶分类分析和检索,SMART软件 (http://smart.embl-heidelberg.de/)[16]进行蛋白质结构组件分析,SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)[17]用于信号肽预测,使用Primer premier 5.0软件进行引物设计。
1.3.2 α-淀粉酶基因克隆以Anoxybacillus flavithermus WK1的基因座位“Aflv_2188”的Amylase序列为参照,对淀粉酶氨基酸序列BLAST分析,选取保守区域进行引物设计,正向引物Amy-F:5′-ATGGTCGACCGKTTYAA-3′,反向引物Amy-R:5′-AAAATCRCCGCGMCGMARVGA-3′(K=T/G, Y=C/T,R=A/G,M=A/C, V=A/G/C),按基因组步移试剂盒的说明进行基因步移扩增,获得淀粉酶完整编码基因序列。通过SignalP 4.1 Server,对该淀粉酶序列进行信号肽预测分析。设计引物AmyGX-F:5′-ATCGGATCCAAAACGGAGCGAGCGTGGC-3′和AmyGX-R:5′-AGCCTGCAGTCAGAGATTTGATGCTTTCG-3′,分别在上下游引物的5′端引入BamHⅠ和PstⅠ酶切位点 (下划线部分),扩增去除信号肽序列的结构域部分,凝胶回收目的PCR产物,并进行BamHⅠ和PstⅠ酶切后,与经相同限制性内切酶酶切的表达载体pQE30连接,连接产物转化到大肠杆菌DH5α感受态细胞中,重组质粒命名为pQE30-AmyGX,酶切鉴定重组质粒并测序验证。
1.4 α-淀粉酶的表达和纯化将测序验证正确的重组质粒转化大肠杆菌表达宿主M15,然后转接到含有100 μg/mL氨苄霉素和25 μg/mL卡那霉素的LB培养基中,置37℃、220 r/min摇床培养,当OD600达到0.5~0.6时,加入终浓度为0.5 mmol/L的IPTG进行诱导,在20℃下培养16 h,离心收集菌体,磷酸缓冲液洗涤菌体,以含10 mmol/L咪唑、1 mmol/L PMSF的磷酸缓冲液悬浮菌体后进行超声破碎。对表达的重组蛋白进行过Ni-NTA柱、阴离子柱Hiload 16/10 Q Sepharose High Performance、分子筛柱SuperdexTM 75 10/300GL的纯化和置换缓冲液,纯化后的蛋白保存在pH值为8.0的50 mmol/L Tris-HCl缓冲液中,SDS-PAGE检测其纯度。
1.5 α-淀粉酶的活性分析酶活力分析采用二硝基水杨酸 (DNS) 法[18],在相应的温度、pH值条件下,以490 μL 1%淀粉溶液为底物,加入10 μL适当稀释的淀粉酶液,反应10 min,加入500 μL DNS液终止反应,沸水浴中反应10 min,测定OD540吸收值,以麦芽糖的DNS显色OD540吸收值绘制标准曲线进行标定。所有分析进行3个平行实验测定。
酶活力单位定义:在最适反应pH值和最适反应温度条件下,每毫升反应体系中每分钟水解可溶性淀粉产生1 μmol麦芽糖所需的酶量为1活力单位 (U)。
1.6 α-淀粉酶的酶学性质研究 1.6.1 最适反应pH值和最适反应温度的测定配置pH值为4.5~10.5(梯度为0.5) 的一系列缓冲液,其中,pH值为4.5~5.5时采用乙酸-乙酸钠缓冲液 (0.1 mol/L)、pH值为6.0~7.0时采用磷酸二氢钠-磷酸氢二钠缓冲液 (0.1 mol/L)、pH值在7.5~8.5时采用Tris-HCl缓冲液 (0.1 mol/L)、pH值为9.0~10.5时采用甘氨酸-氢氧化钠缓冲液 (0.1 mol/L),在60℃反应10 min,以最高酶活力为100%计算相对酶活,绘制pH值-相对活力曲线,确定最适反应pH值。
在最适pH值条件下,进行40~75℃温度范围下的反应10 min,以最高酶活力为100%计算相对酶活,绘制温度-相对活力曲线,确定最适反应温度。
1.6.2 动力学常数测定在最适反应温度和最适反应pH值条件下,测定1 μg重组α-淀粉酶在一系列淀粉浓度 (1.00 mg/mL、1.25 mg/mL、2.00 mg/mL、2.50 mg/mL、4.00 mg/mL、6.00 mg/mL、8.00 mg/mL、10.0 mg/mL) 条件下的表观催化速率,绘制1/V-1/[S]双倒数曲线。
1.6.3 酶热稳定性的测定在梯度PCR仪上,取0.1 mg/mL的重组淀粉酶在一系列温度 (56.0℃、58.0℃、60.0℃、62.0℃、64.0℃、66.0℃、68.0℃、70.0℃) 上热激30 min,然后在最适反应温度和pH值条件下检测残余酶活力,分析重组淀粉酶的热半失活温度T5030值,以未经温度处理的酶的活力为100%。
1.6.4 金属离子对重组淀粉酶的影响在反应体系中,加入终浓度为5 mmol/L的金属离子Na+、K+、Mg2+、Ca2+、Zn2+、Co2+、Cu2+、Ba2+、Fe3+,在最适反应温度和pH值条件下,测定金属离子对重组淀粉酶AmyGX活性的影响,以不添加金属离子的酶的活力为100%。
2 结果与分析 2.1 产耐热α-淀粉酶菌株的鉴定以M9培养基加上可溶性淀粉为唯一碳源的筛选培养基,共分离获得6株具明显降解可溶性淀粉的菌株,挑选其中透明圈直径 (H) 与菌落直径 (C) 比值 (H/C) 大的菌株作进一步的分析。
提取该菌株的基因组DNA,PCR反应扩增得到约1.5 kb的DNA片段,测序后,经序列比对、系统进化树分析 (图 1),确定该菌株为厚壁菌门的厌氧芽胞杆菌属菌株,将其命名为Anoxybacillussp.GXS-3(GenBank Accession No.JF831203)。

|
图 1 菌株Anoxybacillussp.GXS-3的16S rDNA系统进化树 Fig.1 Phylogenetic dendrogram based on 16S rDNA sequences showing the position of strain Anoxybacillus sp.GXS-3 amongAnoxybacilluscluster.Bacillus subtilisis out-group.Bar indicates 0.01 nucleotide substitution per position |
以Anoxybacillussp.GXS-3的16S rDNA序列在NCBI数据库中进行搜索比对分析,发现与其序列一致性达99%的好热黄无氧芽胞杆菌Anoxybacillus flavithermus WK1已完成了全基因组测序 (GenBank Accession No.CP000922)[19],基因组注释显示Anoxybacillus flavithermus WK1编码一个α-淀粉酶,基因座位为“Aflv_2188”,以此序列为参照进行氨基酸序列的BLAST分析 (图 2)。根据Geobacillus kaustophilus HTA426(GenBank Accession No.BA000043),Geobacillussp.POT5 (GenBank Accession No.EF080863) 和Anoxybacillus flavithermusWK1的淀粉酶保守序列YFIMVDRFNN、LPSLRRGDF,设计简并引物对Anoxybacillussp.GXS-3的基因组DNA进行PCR扩增,得到的DNA片段约1.1 kb,测序分析。按照基因组步移试剂盒的说明,对目标序列进行两端步移扩增。对扩增后的基因序列进行启动子区、开放阅读框的分析,获得具有完整编码序列的α-淀粉酶基因 (GenBank Accession No.JF946749),该基因编码505个氨基酸,与Aflv_2188的一致性为92%,命名为AmyGX。使用CAZY数据库进行检索,AmyGX属于糖苷水解酶类的第13家族 (GH13);使用SMART工具分析蛋白质的结构组件,AmyGX的N端第13~361位氨基酸为α-淀粉酶家族结构域;使用SignalP 4.1分析信号肽序列,显示AmyGX在第23与24位氨基酸为信号肽酶切割位点。

|
Red:Identical; Orange:Strong similarity; Blue:Weak similarity; White:Non-matching 图 2 α-淀粉酶氨基酸序列的比对 Fig.2 Alignment of putative α-amylase sequences from the strain Anoxybacillus flavithermusWK1, Geobacillussp.POT5 and Geobacillus kaustophilus HTA426 |
经IPTG诱导表达,SDS-PAGE分析,含有重组质粒pQE30-AmyGX的大肠杆菌菌株M15与对照菌株相比,在约58 kDa处有明显蛋白质特征条带出现,蛋白分子量与由基因序列推测所得理论分子量一致;经Ni-NTA亲和层析、阴离子柱、分子筛柱分离纯化,获得纯度大于90%的重组蛋白AmyGX,用于酶学性质分析 (图 3)。
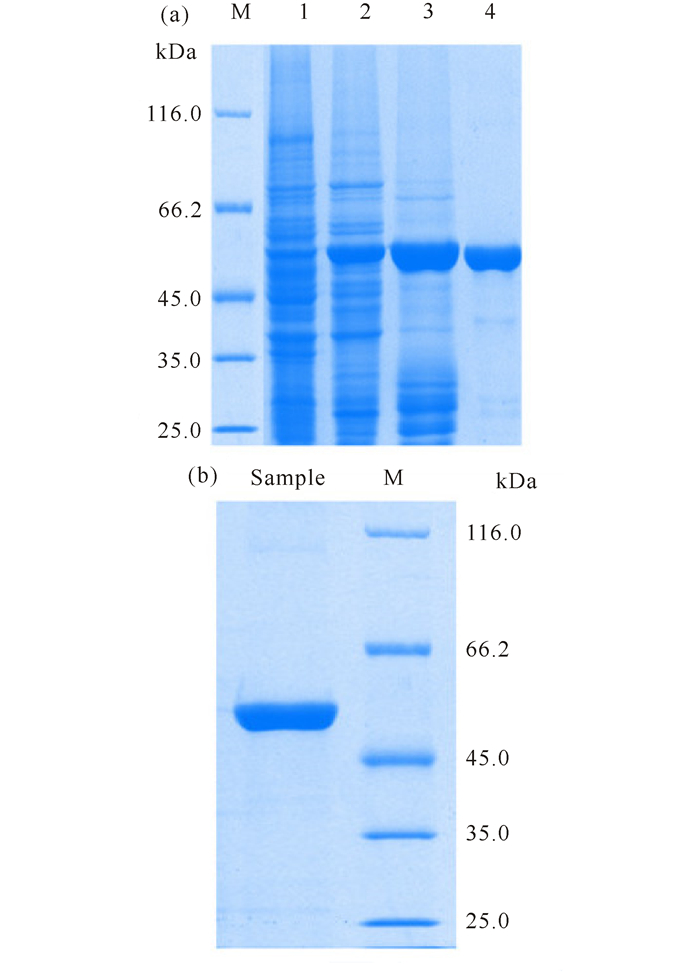
|
M:Molecular weight markers; 1:Crude extract of E.coliM15 cells before induction; 2:Crude extract of E.coli M15 cells, 16 h after IPTG induction; 3:Purified recombinant AmyGX after Ni2+-affinity chromatography; 4:Purified recombinant AmyGX after ion-exchange chromatography. Sample:Purified recombinant AmyGX after size-exclusion chromatography 图 3 重组α-淀粉酶AmyGX的纯化 Fig.3 SDS-PAGE analysis of recombinant AmyGX |
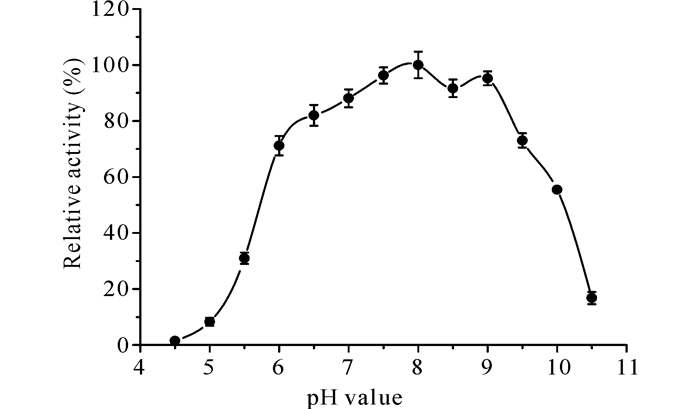
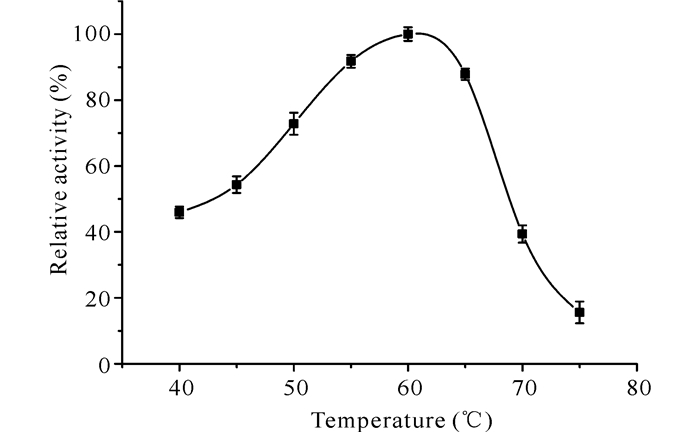
如图 4所示,重组酶AmyGX的最适反应pH值为8.0,当pH值为6.0~9.5时,酶活力在70%以上,表明重组酶AmyGX是一个偏碱性的α-淀粉酶。在最适反应pH值条件下,重组酶AmyGX的最适反应温度为60℃,当温度为55~65℃时,酶活力在85%以上 (图 5)。
|
图 4 pH值对重组酶AmyGX活性的影响 Fig.4 Effect of pH on recombinant AmyGX activity |
|
图 5 温度对重组酶AmyGX活性的影响 Fig.5 Effect of temperature on recombinant AmyGX activity |
如图 6所示,根据重组酶AmyGX在不同浓度底物下的双倒数曲线,通过米氏方程计算出重组酶AmyGX的Vmax、Km值分别为0.19 U/mg、3.14 mg/mL。

|
图 6 重组酶AmyGX的双倒数曲线 Fig.6 Lineweaver-Burk plot of recombinant AmyGX |
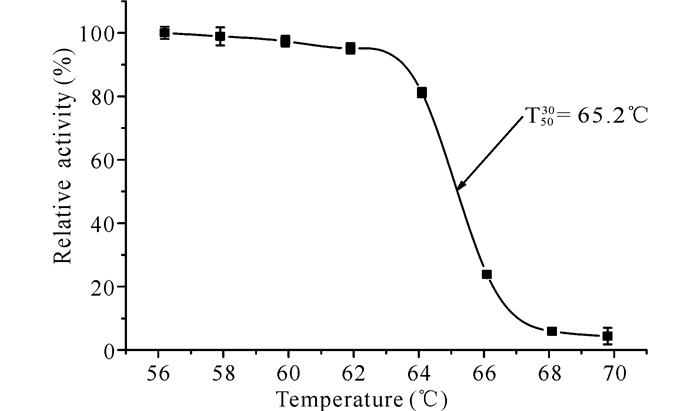
如图 7所示,重组酶AmyGX的T5030值为65.2℃。当温度为56~62℃时,其酶活力没有明显变化,属于中等耐温酶。
|
图 7 重组酶AmyGX的热稳定性分析 Fig.7 Thermostability of recombinant AmyGX |
如表 1所示,Zn2+完全抑制重组酶AmyGX的活性;Cu2+、Co2+、Fe3+对重组酶AmyGX具有强烈的抑制作用,相对酶活力分别下降到1.12%、27.59%、37.99%;Ba2+对重组酶AmyGX具有明显的抑制作用;Mg2+、Ca2+对重组酶AmyGX的活力影响不明显;Na+、K+则对重组淀粉酶AmyGX有激活作用,相对酶活力分别提高到129%、115%。
| 表 1 金属离子对重组淀粉酶AmyGX活性的影响 Table 1 Effect of metal ions (5 mmol/L) on the activity of recombinant AmyGX |
Anoxybacillus最初是被作为一种新的厌氧芽孢杆菌属报道[9],随后,兼性厌氧、需氧的Anoxybacillus菌株相继被发现[20],目前,属于Anoxybacillus的有22个种和2个亚种被命名 (http://www.bacterio.net)。我们从腾冲火山温泉的土壤中,经筛选培养基分离、16S rDNA鉴定,获得生长温度为60℃的产α-淀粉酶的中等耐热菌株Anoxybacillussp.GXS-3。从进化树的结果分析,与Anoxybacillussp.GXS-3进化关系最近的为Anoxybacillus gonensis NCIMB 13933T,其次为Anoxybacillus kamchatkensis DSM 22626T和Anoxybacillus salavatliensisDSM 14988T,与同是来源于腾冲温泉的Anoxybacillus tengchongensis KCTC 1372T、Anoxybacillus flavithermussubsp.yunnanensisDSM23293T尚有一定的进化距离。腾冲火山温泉中蕴藏着丰富的嗜热菌资源,尚待进一步的挖掘。
与来源于嗜热生境中的耐热α-淀粉酶往往需要Ca2+来维持其稳定的构象不同,Ca2+对重组淀粉酶AmyGX的酶活力影响不明显,这与Kikani等[21]对来源于Anoxybacillus beppuensis的α-淀粉酶的描述相一致。Zn2+能完全抑制重组酶AmyGX的活性,表明该酶的活性中心存在含巯基或羧基的关键氨基酸残基[22]。我们根据淀粉酶的两端保守序列YFIMVDRFNN、LPSLRRGDF,采用步移法从Anoxybacillus sp.GXS-3中克隆出α-淀粉酶基因AmyGX并在大肠杆菌中表达。该α-淀粉酶的氨基酸序列与来源于Geobacillus thermoleovorans的α-淀粉酶4E2O的一致性最大,为72%;其次与来源于Thermus sp.IM 6501的α-淀粉酶1SMA的一致性为31%;与目前工业上广泛应用的嗜热脂肪芽孢杆菌α-淀粉酶 (Bacillus stearothermophilus α-amylase,BAA)[23]和地衣芽孢杆菌α-淀粉酶 (Bacillus licheniformis α-amylase,BLA)[24]等的一致性低于20%。是一类结构新颖的淀粉酶,我们将对该酶开展结构与功能关系的后续相关研究。
4 结论α-淀粉酶基因AmyGX全长1 515 bp,编码505个氨基酸,属于糖苷水解酶13家族 (GH13),具有淀粉酶的7个保守氨基酸序列区域 (CSR)。重组酶AmyGX的最适反应温度为60℃,最适反应pH值为8.0,T5030值为约65.2℃,在pH值为6.0~9.5的范围内具有较稳定的酶活力,为中等耐温碱性酶,在造纸、纺织品、洗涤剂生产、有毒废弃物去除和环境金属离子监测等方面具有潜在的应用。
| [1] |
MACGREGOR E A. α-Amylase structure and activity[J]. Journal of Protein Chemistry, 1988, 7(4): 399-415. DOI:10.1007/BF01024888 |
| [2] |
AZAD M A K, BAE J H, KIM J S, et al. Isolation and characterization of a novel thermostable α-amylase from Korean pine seeds[J]. New Biotechnology, 2009, 26(3/4): 143-149. |
| [3] |
BURHAN A, NISA U, GÖKHAN C, et al. Enzymatic properties of a novel thermostable, thermophilic, alkaline and chelator resistant amylase from an alkaliphilic Bacillus sp.isolate ANT-6[J]. Process Biochemistry, 2003, 38(10): 1397-1403. DOI:10.1016/S0032-9592(03)00037-2 |
| [4] |
VAN DER MAAREL M, VAN DER VEEN B, UITDEHAAG J C M, et al. Properties and applications of starch-converting enzymes of the α-amylase family[J]. Journal of Biotechnology, 2002, 94(2): 137-155. DOI:10.1016/S0168-1656(01)00407-2 |
| [5] |
ZEIKUS J G, VIEILLE C, SAVCHENKO A. Thermoz-ymes:Biotechnology and structure-function relationships[J]. Extremophiles, 1998, 2(3): 179-183. DOI:10.1007/s007920050058 |
| [6] |
AGVLOGLU S, ENSARI N Y, UYAR F, et al. The effects of amino acids on production and transport of α-amylase through bacterial membranes[J]. Starch-Stärke, 2000, 52(8/9): 290-295. |
| [7] |
GOH K M, KAHAR U M, CHAI Y Y, et al. Recent discoveries and applications of Anoxybacillus[J]. Applied Microbiology and Biotechnology, 2013, 97(4): 1475-1488. DOI:10.1007/s00253-012-4663-2 |
| [8] |
HEINEN W, LAUWERS A M, MULDERS J W. Baci-llus flavothermus, a newly isolated facultative thermophile[J]. Antonie van Leeuwenhoek, 1982, 48(3): 265-272. DOI:10.1007/BF00400386 |
| [9] |
PIKUTA E, LYSENKO A, CHUVILSKAYA N, et al. Anoxybacillus pushchinensis gen.nov., sp nov., a novel anaerobic, alkaliphilic, moderately thermophilic bacterium from manure, and description of Anoxybacillus falvitherms comb.nov[J]. International Journal of Systematic and Evolutionary Microbiology, 2000, 50(6): 2109-2117. DOI:10.1099/00207713-50-6-2109 |
| [10] |
KARAOGLU H, YANMIS D, SAL F A, et al. Bioche-mical characterization of a novel glucose isomerase from Anoxybacillus gonensis G2T that displays a high level of activity and thermal stability[J]. Journal of Molecular Catalysis B:Enzymatic, 2013, 97: 215-224. DOI:10.1016/j.molcatb.2013.08.019 |
| [11] |
TOKGÖZ M, BEKTAS K I, BELDUZ A O, et al. Cloning, purification, and characterization of a thermophilic ribulokinase fromAnoxybacillus kestanbolensis AC26Sari[J]. Molecular Biology, 2014, 38(38): 633-639. |
| [12] |
XU J Y, REN F F, HUANG C H, et al. Functional and structural studies of pullulanase from Anoxybacillus sp.LM18-11[J]. Proteins:Structure Function and Bioinformatics, 2014, 82(9): 1685-1693. DOI:10.1002/prot.24498 |
| [13] |
MCWILLIAM H, LI W Z, ULUDAG M, et al. Analysis tool web services from the EMBL-EBI[J]. Nucleic Acids Research, 2013, 41(W1): W597-W600. DOI:10.1093/nar/gkt376 |
| [14] |
BUSBY S, EBRIGHT R H. Promoter structure, prom-oter recognition, and transcription activation in prokaryotes[J]. Cell, 1994, 79(5): 743-746. DOI:10.1016/0092-8674(94)90063-9 |
| [15] |
LOMBARD V, RAMULU H G, DRULA E, et al. The carbohydrate-active enzymes database (CAZy) in 2013[J]. Nucleic Acids Research, 2014, 42(D1): D490-D495. DOI:10.1093/nar/gkt1178 |
| [16] |
LETUNIC I, DOERKS T, BORK P. SMART:Recent updates, new developments and status in 2015[J]. Nucleic Acids Research, 2015, 43(D1): D257-D260. DOI:10.1093/nar/gku949 |
| [17] |
PETERSEN T N, BRUNAK S, VON HEIJNE G, et al. SignalP 4.0:Discriminating signal peptides from transmembrane regions[J]. Nature Methods, 2011, 8(10): 785-786. DOI:10.1038/nmeth.1701 |
| [18] |
MILLER G L. Use of dinitrosalicylic acid reagent for determination of reducing sugar[J]. Analytical Chemistry, 1959, 31(3): 426-428. DOI:10.1021/ac60147a030 |
| [19] |
SAW J H, MOUNTAIN B W, FENG L, et al. Encapsulated in silica:Genome, proteome and physiology of the thermophilic bacterium Anoxybacillus flavithermus WK1[J]. Genome Biology, 2008, 9: R161. DOI:10.1186/gb-2008-9-11-r161 |
| [20] |
PIKUTA E, CLELAND D, TANG J. Aerobic growth of Anoxybacillus pushchinoensis K1T:Emended descriptions of A.pushchinoensisand the genus Anoxybacillus[J]. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(5): 1561-1562. DOI:10.1099/ijs.0.02643-0 |
| [21] |
KIKANI B A, SINGH S P. The stability and thermodynamic parameters of a very thermostable and calcium-independent α-amylase from a newly isolated bacterium, Anoxybacillus beppuensis TSSC-1[J]. Process Biochemistry, 2012, 47(12): 1791-1798. DOI:10.1016/j.procbio.2012.06.005 |
| [22] |
CHAI Y Y, RAHMAN R N Z R A, ILLIAS R M, et al. Cloning and characterization of two new thermostable and alkali-tolerant α-amylases from the Anoxybacillus species that produce high levels of maltose[J]. Journal of Industrial Microbiology & Biotechnology, 2012, 39(5): 731-741. |
| [23] |
ALIKHAJEH J, KHAJEH K, RANJBAR B, et al. Structure ofBacillus amyloliquefaciens α-amylase at high resolution:Implications for thermal stability[J]. Acta Crystallographica F:Structural Biology and Crystallzation Communications, 2010, 66: 121-129. DOI:10.1107/S1744309109051938 |
| [24] |
DECLERCK N, MACHIUS M, CHAMBERT R, et al. Hyperthermostable mutants of Bacillus licheniformis α-amylase:Thermodynamic studies and structural interpretation[J]. Protein Engineering Design and Selection, 1997, 10(5): 541-549. DOI:10.1093/protein/10.5.541 |
 2017, Vol. 24
2017, Vol. 24 


