2. 广西大学,广西南宁 530004;
3. 美国加利福里亚大学生物化学和分子生物系,戴维斯 CA 95616;
4. 美国戈登生命科学研究所,波斯顿 MA 02478
2. Guangxi University, Nanning, Guangxi, 530004, China;
3. Department of Biochemistry and Molecular Medicine, University of California School of Medicine, Davis, CA 95616, USA;
4. Gordon Life Science Institute, Boston, MA 02478, USA
在哺乳动物细胞中由α-2, 8键连接成的多唾液酸聚糖 (polySA),能够对神经细胞黏附分子 (NCAM) 进行翻译后修饰,是一类和肿瘤细胞有联系的抗原。polySA在胚胎发育、突触可塑性形成、突触发生、神经干细胞增殖和分化及肿瘤细胞远端转移等过程中,对细胞间的相互作用的调节起到关键的作用[1-4]。NCAM是多聚唾液酸的主要载体蛋白[5-9],其上的polySA能在胚胎脑部大量表达。虽然在成人脑组织中大多数NCAM含有较低水平的polySA,但polySA能持续地在成人大脑的神经中枢的可塑性区域,包括与空间认知和记忆有关的海马体、下丘脑等区域[10-11]表达。此外,细胞迁移和神经轴索的收缩等现象也和神经细胞黏附分子的多聚唾液酸化有联系[12]。
PolySA是由ST8SiaⅡ(STX) 和ST8SiaⅣ(PST) 两种多聚唾液酸转移酶 (polySTs) 催化而成。在多聚唾液酸链延伸过程中,STX和PST把唾液酸残基从其高能活化前体胞苷-磷酸-乙酰神经氨酸 (CMP-Neu5Ac) 转移到神经细胞黏附分子或其他蛋白受体的非还原性末端,通过N-型和O-型共价键连接到受体糖复合物的寡聚糖链上[13-21]。
到目前为止,除了寡聚唾液酸转移酶ST8Sia Ⅲ的晶体结构之外,用X射线晶体学或核磁共振 (NMR) 光谱法还没有获得ST8Sia家族中其它成员的三维结构。这主要是由于这些唾液酸酶分子中含有大量的疏水性氨基酸残基,存在于细胞膜环境中,以及蛋白中有较多的非结构区域,这就不易获得理想的实验样品来进行X射线晶体学和NMR的结构生物学实验[22]。因此,至今所得到的有关唾液酸转移酶的结构信息还是很有限的。
然而,近来我们用Phyre2软件[23]构建成的ST8Sia Ⅳ多聚唾液酸酶分子结构是一个相当精确的三维结构模型[22],这个预测的模型支持了在前面的研究中所提出的推测,即在ST8Sia Ⅳ中存在着识别NCAM并催化NCAM多聚唾液酸化的特定结构区域。通过对这个ST8Sia Ⅳ模型及其突变体模型之间的比较和分析,表明在该多聚唾液酸转移酶中的聚碱基多聚唾液酸转移酶结构域 (PSTD) 和聚碱的氨基酸区域 (PBR) 对NCAM的多聚唾液酸化起了关键作用[24]。在ST8Sia家族成员结构研究中另一个重要的进展是,寡聚唾液酸转移酶ST8Sia Ⅲ的X-射线晶体结构已经被解析出来[25]。该晶体结构的确定,不仅有助于我们对NCAM多聚唾液酸化分子机制有更清楚的理解,而且还为构建ST8Sia家族中所有其它成员的三维结构提供了更为精确的结构模板。
ST8SiaⅡ是肿瘤细胞多聚唾液酸化的主要驱动源[26]。因为多聚唾液酸在癌细胞中的再表达促使肿瘤发生转移[6],于是ST8SiaⅡ多聚唾液酸酶是一个引起科学家们注意和重视的抗肿瘤靶。为了研究肿瘤细胞的转移、新陈代谢和NCAM的多聚唾液酸化等的分子机制,就必须先确定ST8SiaⅡ多聚唾液酸转移酶的三维结构,并找出其结构与催化功能之间的内在联系,这将对抑制肿瘤细胞转移的抑制物研究和设计,以及开发有效的药物提供更有价值的信息。
1 ST8SiaⅡ的三维模型及其特征正如以上分析,Phyre2服务器对蛋白结构特征进行预测已被证明是一个切实可行的方法,用该方法能可靠地检测到与目标蛋白很遥远的同源序列。即使序列之间的一致性低到15%[23],也能够构建出一个精确的蛋白模型。我们先前所构建的ST8Sia Ⅳ的结构模型,采用来源于哺乳动物细胞中单唾液酸酶和半乳糖-β-1, 3-N-乙酰半乳糖胺-正-硝基酚的复合物的晶体结构作为模板 (编码为c2wnfa)[23],所获得的预测模型的最大置信值为100%,氨基酸序列一致性为26%。然而当我们采用最近发表的ST8Sia Ⅲ的晶体结构[25]作为模板再预测ST8Sia Ⅳ的三维结构时,置信值仍为100%,但序列一致性已升至40%。这表明最新的ST8Sia Ⅳ三维结构模型比原来的更为精准。因此,在本研究中,我们用ST8Sia Ⅲ的晶体结构作为模板 (编码为c5bo6b) 来构建ST8SiaⅡ的三维模型 (图 1)。

|
多聚唾液酸转移酶结构域 (PSTD) 和多聚碱性氨基酸区域 (PBR) 分别用黄色和蓝色表征;在这两个区域中所有的碱性氨基酸标记为红色;两对二硫键的Cys原子用球形表征 All basic amino acid residues on the PSTB and PBR are shown in red, and other residues in PSTB and PBR are shown in yellow and blue, respectively; The Cys atoms formed disulfide bonds are shown as spheres 图 1 ST8SiaⅡ分子主链的三维骨架构象 Fig.1 The backbone conformation of the 3D structure of ST8Sia Ⅱ |
ST8SiaⅡ的结构中含有一对反平行β-折叠,对应的氨基酸序列是从G120到I123,以及从M133到V135,靠近该酶的N端,位于蛋白的表面,该反平行β-折叠的功能目前还不清楚。在ST8SiaⅡ的结构中,每个β-折叠和其邻近β-折叠的距离为4.5~5.0 Å,所有8个平行的β-折叠都包埋在蛋白分子内,并处于分子模型的核心区,那里富含疏水性氨基酸。另外,与ST8SiaⅣ结构相似,ST8SiaⅡ(共375氨基酸残基) 也含有两对二硫键:C157:C307和C171:C371。由于这4个残基构成的每个二硫键中都有一个半胱氨酸残基非常接近蛋白分子的C端,而大多数C端氨基酸残基包括C末端的C371被折叠到蛋白分子的中心或核心区,于是C371不是处在C末端的无规则卷曲区。这种折叠方式合理地说明了C371也是ST8SiaⅡ酶活性所需要的残基。以上这些特征提出了ST8Sia Ⅱ的催化区域可能是从残基85到371的范围内。
这个预测的ST8SiaⅡ三维模型,表明了从N端的前85个氨基酸残基中,包含有1个被认为是跨膜区的、长的α-螺旋 (Leu3~Glu30)[25]。从第31到第84个残基中的大多数氨基酸属于无规则卷曲区,在该区域不存在稳定的构象[27]。此外,与ST8Sia Ⅳ的模型相似的是,同它们的C末端部分相比,ST8SiaⅡ的N端区域远离酶分子模型的核心区。因此,这些区域可能不存在影响酶的活性和对多聚唾液酸化有催化作用的氨基酸残基。
2 影响NCAM多聚唾液酸化的功能域及关键氨基酸残基 2.1 PSTD功能域ST8SiaⅡ(STX) 和ST8Sia Ⅳ(PST) 的氨基酸序列的一致性约为59%,它们的催化区位于高尔基复合体的内腔[28-32]。Troy和他的同事发现,在ST8SiaⅡ和ST8Sia Ⅳ中都含有一个由32个氨基酸残基组成的多碱性氨基酸区域 (pI值约为12),位于小唾液酸化 (SMS) 区域的上游并且紧挨着SMS[32]。这个区域是PSTD。在其它的糖苷转移酶中,包括葡糖氨基葡聚糖的合成或聚合的相关酶类中都不存在PSTD。虽然ST8Sia Ⅲ能够催化几个神经糖蛋白的寡聚唾液酸化,并与ST8SiaⅡ和ST8Sia Ⅳ有中等程度的同源性,但由于它缺乏PSTD,所以也不能催化NCAM多聚唾液酸化。
前面的研究已经确定了ST8Sia Ⅳ和ST8SiaⅡ中的PSTD功能域分別是从第246到第277个氨基酸残基范围和从第261至第292个氨基酸残基范围 (图 2)。ST8Sia Ⅳ的PSTD的点突变模型和突变分析表明,此结构域中带正电荷的残基,如Arg252、Lys276和Arg277对于其多聚唾液酸转移酶的活性是至关重要的[32],并且我们的模型显示了大多数正电荷的氨基酸残基都位于蛋白分子的表面 (图 3b)。在多聚唾液酸化过程中这样的分布可能有利于它们和带有负电荷的唾液产生静电相互作用。
|
图 2 在ST8SiaⅡ(STX) 和ST8Sia Ⅳ(PST) 中的多聚唾液酸转移酶结构域 (PSTD) 和多聚碱性氨基酸区域 (PBR),以及其它唾液酸组分[22]。 Fig.2 The polybasic regions (PSTB and PBR) and sialyl motifs in STX (ST8Sia Ⅱ) and ST8Sia Ⅳ(PST)[22] |
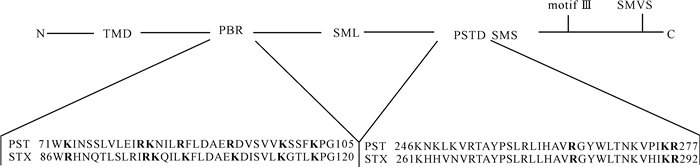
在我们的ST8SiaⅡ模型的PSTD区域内 (K261~R292) 含有11个碱性氨基酸残基。它们是Lys261、H262、H263、R267、R274、H277、R280、K287、H289、K291和R292。这些碱性氨基酸也都处于蛋白分子的CPK模型的表面 (图 3a),因此,它们有可能和负电荷的唾液酸分子之间发生静电相互作用。所以我们的ST8SiaⅡ三维模型支持了前面研究提出的假设,即ST8SiaⅡ中PSTD区域内的碱性氨基酸残基对NCAM的多聚唾液酸化起到关键作用。
|
多聚唾液酸转移酶结构域 (PSTD) 和多聚碱性氨基酸区域 (PBR) 被分别用黄色和蓝色表征;在这两个区域中所有的碱性氨基酸被标记为红色 All basic amino acid residues on the PSTB and PBR are shown in red, and other residues in PSTB and PBR are shown in yellow and blue, respectively 图 3 ST8SiaⅡ(a) and ST8Sia Ⅳ(b) 的三维空间充填原子球体模型 Fig.3 The critical residues within the PSTD and PBR domains required for polysialylation of NCAM in the wild-type 3D space-filling model of ST8Sia Ⅱ(a) and ST8Sia Ⅳ(b) |
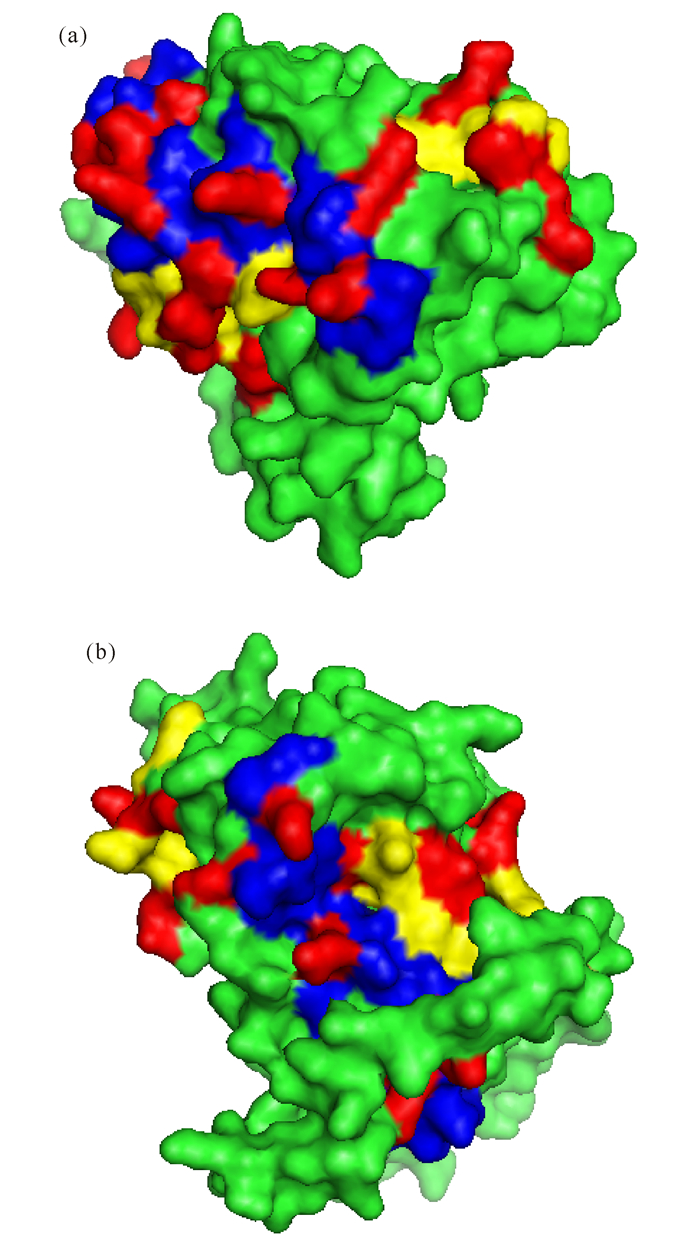
由于PSTD和PBR结构域中的氨基酸顺序对于ST8SiaⅡ和ST8Sia Ⅳ来说是高度保守的[32],我们构建的ST8SiaⅡ和ST8Sia Ⅳ的三维模型也显示了这两个多聚唾液酸转移酶的相应的PSTD和PBR区域具有相似的构象 (图 4),所以不难理解,ST8Sia Ⅳ中的PSTD和PBR结构域与ST8Sia Ⅳ中相应的PSTD和PBR应当有相似的功能。在ST8Sia Ⅳ的PSTD末端的最后3个氨基酸残基 (I275-K276-R277) 与ST8SiaⅡ的PSTD的最后3个氨基酸残基 (I290-K291-R292) 是完全相同的。因为在前面的突变实验和点突变模型已经证明了ST8Sia Ⅳ中的非极性氨基酸I275对ST8Sia Ⅳ的活性有重要影响,所以我们可以合理地推测,在ST8SiaⅡ的PSTD中末端的非极性氨基酸残基I290对于NCAM多聚唾液酸化作用也是十分重要的。
|
在这两个区域中所有的碱性氨基酸被表标记为红色 All basic residues in these motifs are shown by red 图 4 从ST8SiaⅡ(a) 和ST8Sia Ⅳ(b) 三维结构中抽离出的PBR (蓝色) 和PSTD (黄色) 的结构 Fig.4 The conformations of the corresponding PBR (blue) and PSTD (yellow) motifs of ST8Sia Ⅱ and Ⅳ |
通过比较野生型的ST8Sia Ⅳ的模型 (I275-K276-R277) 及其两个突变分子的模型 (I275K (K275-K276-R277) 和K276R/R277K (I275-R276-K277)),或者野生型的ST8SiaⅡ模型及其两个突变体分子模型I290K (K290-K291-R292) 和R291K/R292K (I290-R291-K292),我们发现每一个多聚唾液酸酶的野生型模型及其突变体分子模型之间在构象上具有显著差异,特别是在它们的PSTD结构域上有不同的构型。这些发现说明在ST8Sia Ⅳ中的l275-K276-R277和在ST8Sia Ⅱ中的I290-R291-K292具有专一性的构型。在此之前,Nakata等[32]已经通过突变实验分析发现即使I275突变为K275后,或者将K276-R277的位置对换为R276-K277后,酶的活性反而降低。虽然在ST8Sia Ⅳ中的l275或在ST8Sia Ⅱ中的I290是一个没有电荷的疏水性残基,但是它们和K (Lys) 及R (Arg) 组成的专一性顺序 (I-K-R) 对酶的活性有重要影响。所以我们的野生型和突变体分子模型分析和以上的突变体实验分析是一致的,表明这3个残基 (I-K-R) 在位置上的专一性决定了它们特定的构型。由于分子突变引起的构象变化可能解释了为什么突变的polyST的催化活性减弱。这些结果也再次支持原来的分析,即PSTD结构域中的碱性氨基酸残基并不是唯一的对ST8Sia Ⅳ或Ⅱ酶活性起重要作用的氨基酸。
2.2 PBR结构域几年前Foley等[33]在多聚唾液酸酶中发现了另一个多碱性氨基酸功能区域,并命名为PBR结构域。在PST和STX中的PBR顺序也是保守的。PBR结构域含有35个氨基酸残基,其中7个为碱性氨基酸。Foley等[33]发现,两个多聚唾液酸酶的PBR中用氨基酸Ala替代两个氨基酸残基 (PST中的Arg82和Arg93,STX中的Arg97和Lys108) 后,这两个酶的自动多聚唾液酸化能力和对NCAM的多聚唾液酸化的催化作用显著减弱。通过分析相关的实验数据,他们确定了在PBR中含有的对NCAM蛋白多聚唾液酸化活性作用所需要的特定的氨基酸残基。
我们的模型表明,ST8Sia Ⅳ和ST8SiaⅡ的PBR结构域具有相似的构象,它们都含有一个长螺旋和两个短螺旋 (图 4)。已知ST8Sia Ⅳ的长螺旋中的Arg82位于螺旋的外侧,它是NCAM多聚唾液酸化过程中的关键氨基酸残基。同样,在ST8SiaⅡ中的Lys95和Lys105也位于长螺旋的外侧,所以这两个碱性氨基酸残基也可能是NCAM多聚唾液酸化的关键氨基酸。
Foley等[33]报道了ST8Sia Ⅳ的Arg93两侧分别是Glu92和Asp94 (图 4b),同样地,ST8SiaⅡ的PBR结构域中的Lys108两侧也分别是Asp和Glu氨基酸残基 (图 4a)。早期的研究表明,当Glu92和Asp94替换成Ala时,ST8Sia Ⅳ的自动多聚唾液酸化能力降低并失去对NCAM的多聚唾液酸化作用。相反,当Glu92替换成Asp92,Asp94替换成Glu94后,则该酶的自动多聚唾液酸化的能力明显增强 (为对照的123%),而其对NCAM的多聚唾液酸化作用并没有恢复。
此外,结合突变实验结果和点突变模型,我们已经确定ST8Sia Ⅳ中的Arg82、Glu92-Arg93-Asp94、Lys99和Lys103是多聚唾液酸化作用所需要的关键氨基酸。因此,根据ST8Sia Ⅳ和ST8SiaⅡ这两个多聚唾液酸转移酶在PBR结构域中的相似性,也可合理地推断,ST8SiaⅡ中的Lys95、Lys105、Glu107-Lys108-Asp109、Lys114和Lys118也是其自动多聚唾液酸化和对NCAM多聚唾液酸化作用所需要的关键氨基酸。
3 结论我们用Phyre2软件构建成的ST8SiaⅡ多聚唾液酸酶分子结构是一个相当精确的三维结构模型。ST8SiaⅡ和Ⅳ在它们的PSTD和PBR功能区的氨基酸顺序是高度保守的,在构型上也是相似的,于是根据前面有关ST8Sia Ⅳ多聚唾液酸转移酶的突变分析结果,同时结合目前的ST8SiaⅡ三维结构模型的特征分析,我们初步确定了在ST8SiaⅡ的PSTD和PBR结构域中对NCAM多聚唾液酸化起关键作用的氨基酸,这将在下一步的NMR实验研究中得到进一步证实。ST8SiaⅡ三维模型的构建,不仅有助于我们深入理解多聚唾液酸转移酶调控NCAM多聚唾液酸化过程中的分子机制,也对该酶影响的肿瘤细胞转移具有抑制作用的抑制物和药物的发现和设计提供了重要的理论依据。
| [1] |
NAKAYAMA J, FUKUDA M. A human polysialyltran-sferase directs in vitro synthesis of polysialic acid[J]. J Biol Chem, 1996, 271(4): 1829-1832. DOI:10.1074/jbc.271.4.1829 |
| [2] |
ANGATA K, FUKUDA M. Polysialyltransferases:Major players in polysialic acid synthesis on the neural cell adhesion molecule[J]. Biochimie, 2003, 85(1/2): 195-206. |
| [3] |
SCHACHNER M, MARTINI R. Glycans and the modulation of neural-recognition molecule function[J]. Trends Neurosci, 1995, 18(4): 183-191. DOI:10.1016/0166-2236(95)93899-9 |
| [4] |
PARK K H, YEO S W, TROY Ⅱ F A. Expression of polysialylated neural cell adhesion molecules on adult stem cells after neuronal differentiation of inner ear spiral ganglion neurons[J]. Biochem Biophys Res Commun, 2014, 453(2): 282-287. DOI:10.1016/j.bbrc.2014.05.035 |
| [5] |
INOUE S, IWASAKI M. Isolation of a novel glycoprotein from the eggs of rainbow trout:Occurrence of disialosyl groups on all carbohydrate chains[J]. Biochem Biophys Res Commun, 1978, 83(3): 1018-1023. DOI:10.1016/0006-291X(78)91497-3 |
| [6] |
FINNE J. Occurrence of unique polysialosyl carbohydra-te units in glycoproteins of developing brain[J]. J Biol Chem, 1982, 257(20): 11966-11970. |
| [7] |
EDELMAN G M. Modulation of cell adhesion during induction, histogenesis, and perinatal development of the nervous system[J]. Annu Rev Neurosci, 1984, 7: 339-377. DOI:10.1146/annurev.ne.07.030184.002011 |
| [8] |
RUTISHAUSER U, LANDMESSER L. Polysialic acid in the vertebrate nervous system:A promoter of plasticity in cell-cell interactions[J]. Trends Neurosci, 1996, 19(10): 422-427. DOI:10.1016/0166-2236(96)10041-2 |
| [9] |
KISS J Z, ROUGON G. Cell biology of polysialic acid[J]. Curr Opin Neurobiol, 1997, 7(5): 640-646. DOI:10.1016/S0959-4388(97)80083-9 |
| [10] |
SEKI T, ARAI Y. Distribution and possible roles of the highly polysialylated neural cell adhesion molecule (NCAM-H) in the developing and adult central nervous system[J]. Neurosci Res, 1993, 17(4): 265-290. DOI:10.1016/0168-0102(93)90111-3 |
| [11] |
HU H Y, TOMASIEWICZ H, MAGNUSON T, et al. The role of polysialic acid in migration of olfactory bulb interneuron precursors in the subventricular zone[J]. Neuron, 1996, 16(4): 735-743. DOI:10.1016/S0896-6273(00)80094-X |
| [12] |
SEKI T, RUTISHAUSER U. Removal of polysialic acid-neural cell adhesion molecule induces aberrant mossy fiber innervation and ectopic synaptogenesis in the hippocampus[J]. J Neurosci, 1998, 18(10): 3757-3766. |
| [13] |
MCCOY R D, VIMR E, TROY Ⅱ F A. CMP-Neu5Ac:poly-α-2, 8-sialosyl sialyltransferase and the biosynthesis of polysialosyl units in neural cell adhesion molecules[J]. J Biol Chem, 1985, 260(23): 12695-12699. |
| [14] |
KITAZUME S, KITAJIMA K, INOUE S, et al. Developmental expression of trout egg polysialoglycoproteins and the prerequisite α2, 6-, and α2, 8-sialyl and α2, 8-polysialyltransferase activities required for their synthesis during oogenesis[J]. J Biol Chem, 1984, 269(14): 10330-10340. |
| [15] |
KOJIMA N, TACHIDA Y, YOSHIDA Y, et al. Characterization of mouse ST8Sia Ⅱ(SIA8SIA Ⅱ) as a neural cell adhesion molecule-specific polysialic acid synthase.Requirement of core α1, 6-linked fucose and a polypeptide chain for polysialylation[J]. J Biol Chem, 1996, 271(32): 19457-19463. DOI:10.1074/jbc.271.32.19457 |
| [16] |
KITAZUME-KAWAGUCHI S, KABATA S, ARITA M. Differential biosynthesis of polysialic or disialic acid structure by ST8Sia Ⅱ and ST8Sia Ⅳ[J]. J Biol Chem, 2001, 276(19): 15696-15703. DOI:10.1074/jbc.M010371200 |
| [17] |
SEVIGNY M B, YE J, KITAZUME-KAWAGUCHI S, et al. Developmental expression and characterization of the α2, 8-polysialyltransferase activity in embryonic chick brain[J]. Glycobiology, 1998, 8(9): 857-867. DOI:10.1093/glycob/8.9.857 |
| [18] |
ZHOU G P, TROY F A. Characterization by NMR and molecular modeling of the binding of polyisoprenols (PI) and polyisoprenyl recognition sequence (PIRS) peptides:3D structure of the complexes reveals sites of specific interactions[J]. Glycobiology, 2003, 13(2): 51-71. DOI:10.1093/glycob/cwg008 |
| [19] |
ZHOU G P, TROY F A. NMR study of the preferred membrane orientation of polyisoprenols (dolichol) and the impact of their complex with polyisoprenyl recognition sequence peptides on membrane structure[J]. Glycobiology, 2005, 15(4): 347-359. |
| [20] |
ZHOU G P, TROY F A. NMR studies on how the binding complex of polyisoprenol recognition sequence peptides and polyisoprenols can modulate membrane structure[J]. Curr Protein Pept Sci, 2005, 6(5): 399-411. DOI:10.2174/138920305774329377 |
| [21] |
NAKAYAMA J, FUKUDA M N, HIRABAYASHI Y, et al. Expression cloning of a human GT3 synthase.GD3 AND GT3 are synthesized by a single enzyme[J]. J Biol Chem, 1996, 271(7): 3684-3691. DOI:10.1074/jbc.271.7.3684 |
| [22] |
ZHOU G P, HUANG R B, TROY F A. 3D Structural conformation and functional domains of polysialyltransferase ST8Sia Ⅳ required for polysialylation of neural cell adhesion molecules[J]. Protein Pept Lett, 2015, 22(2): 137-148. DOI:10.2174/0929866521666141019192221 |
| [23] |
KELLEY L A, STERNBERG M J E. Protein structure prediction on the web:A case study using the Phyre server[J]. Nat Protoc, 2009, 4(3): 363-371. DOI:10.1038/nprot.2009.2 |
| [24] |
CLOSE B E, MENDIRATTA S S, GEIGER K M, et al. The minimal structural domains required for neural cell adhesion molecule polysialylation by PST/ST8Sia Ⅳ and STX/ST8Sia Ⅱ[J]. J Biol Chem, 2003, 278(33): 30796-30805. DOI:10.1074/jbc.M305390200 |
| [25] |
VOLKERS G, WORRALL L J, KWAN D H, et al. Structure of human ST8Sia Ⅲ sialyltransferase provides insight into cell-surface polysialylation[J]. Nat Struct Mol Biol, 2015, 22(8): 627-635. DOI:10.1038/nsmb.3060 |
| [26] |
AL-SARAIREH Y M J, SUTHERLAND M, SPRINGETT B R, et al. Pharmacological inhibition of polysialyltransferase ST8Sia Ⅱ modulates tumour cell migration[J]. PLoS One, 2013, 8(8): e73396. |
| [27] |
SMITH L J, FIEBIG K M, SCHWALBE H, et al. The concept of a random coil:Residual structure in peptides and denatured proteins[J]. Fold Des, 1996, 1(5): R95-R106. DOI:10.1016/S1359-0278(96)00046-6 |
| [28] |
KOJIMA N, YOSHIDA Y, TSUJI S. A developmentally regulated member of the sialyltransferase family (ST8Sia Ⅱ, STX) is a polysialic acid synthase[J]. FEBS Lett, 1995, 373(2): 119-122. DOI:10.1016/0014-5793(95)01024-9 |
| [29] |
ECKHARDT M, MÜHLENHOFF M, BETHE A, et al. Molecular characterization of eukaryotic polysialyltransferase-1[J]. Nature, 1995, 373(6516): 715-718. DOI:10.1038/373715a0 |
| [30] |
SCHEIDEGGER E P, STERNBERG L R, ROTH J, et al. A human STX cDNA confers polysialic acid expression in mammalian cells[J]. J Biol Chem, 1995, 270(39): 22685-22688. DOI:10.1074/jbc.270.39.22685 |
| [31] |
NAKAYAMA J, FUKUDA M N, FREDETTE B, et al. Expression cloning of a human polysialyltransferase that forms the polysialylated neural cell adhesion molecule present in embryonic brain[J]. P Natl Acad Sci USA, 1995, 92(15): 7031-7035. DOI:10.1073/pnas.92.15.7031 |
| [32] |
NAKATA D, ZHANG L R, TROY F A. Molecular basis for polysialylation:A novel polybasic polysialyltransferase domain (PSTD) of 32 amino acids unique to the α2, 8-polysialyltransferases is essential for polysialylation[J]. Glycoconj J, 2006, 23(5): 423-436. |
| [33] |
FOLEY D A, SWARTZENTRUBER K G, COLLEY K J. Identification of sequences in the polysialyltransferases ST8Sia Ⅱ and ST8Sia Ⅳ that are required for the protein-specific polysialylation of the neural cell adhesion molecule, NCAM[J]. J Biol Chem, 2009, 204(23): 15505-15516. |
 2017, Vol. 24
2017, Vol. 24 


