2. 亚热带农业生物资源保护与利用国家重点实验室,广西南宁 530005
2. State Key Laboratory for Conservation and Utilization of Subtropical Agro-bioresources, Nanning, Guangxi, 530005, China
枯草芽孢杆菌(Bacillus subtilis)作为一种模式菌株,具有生理生化和遗传背景清晰、非致病性和分泌性良好的优良特性,因此被用作食品安全级别的微生物。随着Bacillus subtilis 168菌株全基因组测序的完成,越来越多的外源基因已经在枯草芽孢杆菌及其亲缘菌株中成功高效表达,这使其成为基因表达系统的研究热点之一[1]。
相比大肠杆菌表达系统,枯草芽孢杆菌的表达系统研究起步较晚,其本身具有一些独特的优势[2]:1)安全无毒,已被美国食品药品监督管理局(FDA)评为生物安全(Generally regarded as safe,GRAS)菌株;2)较强的蛋白分泌能力,方便下游表达产物的分离;3)发酵工艺成熟,容易进行高密度发酵。但是枯草芽孢杆菌表达系统也存在许多不足之处,主要表现在[3]:启动子转录水平低,某些目的蛋白不能大量表达;重组蛋白折叠不完全,部分没有活性;菌株感受态细胞转化率低,内源性质粒结构不稳定;表达产物在胞外容易被本身分泌的蛋白酶降解。这些因素限制了枯草芽孢杆菌表达系统不能完全满足工业化生产的需求,因此构建新的蛋白表达系统仍是研究枯草芽孢杆菌的重点。
稳定的载体骨架和高效可控的启动子是保证工业上枯草芽孢杆菌高效表达外源蛋白的关键因素,本文对枯草芽孢表达系统的特点做简单的介绍,着重介绍表达系统中的转录调控元件——启动子,详细叙述启动子的种类,新启动子的克隆方法,改造思路以及启动子的预测与分析。
1 枯草芽孢杆菌表达载体克隆载体和表达载体在分子遗传操作领域中具有十分重要的作用,是基因重组技术的重要工具。根据类型的不同可以将枯草芽孢杆菌表达载体分为3类[4]:独立自主复制型载体、整合型载体和噬菌体载体。
枯草芽孢杆菌内源性质粒DNA含量很少,现有的枯草芽孢杆菌表达系统采用的质粒主要来源于葡萄球菌和链球菌[5]等其他微生物。根据不同的复制方式可以将这些质粒载体分为滚环复制型载体和θ复制型载体。只有一些大的质粒载体以θ复制方式进行复制,而一大部分质粒载体则通过滚环复制方式复制,这些质粒载体大小均在在12 Kb以下。来源于金黄色葡萄球菌的质粒pC194、pE194、pT181和pUB110均是以滚环复制方式进行复制的。
pC194大小为2 906 bp,抗氯霉素特性,质粒的拷贝数约15个染色体[6],该质粒较小,容易克隆,当插入外源片段时容易产生大分子的畸形复制产物,从而高度不稳定,这就决定了该质粒载体在宿主中的应用性偏低;pE194大小为3 728 bp,温度敏感型质粒载体,拷贝数大约为10个染色体,该质粒载体在32℃正常复制,随着温度的升高拷贝数会逐渐降低,达到45℃时停止拷贝,所以在枯草芽孢杆菌中也不容易稳定地遗传;pT181具有抗四环素特性,每个细胞中拷贝数约为20个染色体[2];pUB110大小为4 548 bp,抗卡那霉素和博来霉素,每个细胞约有30~50个拷贝数[7],该质粒已被完全测序,但是因其容易产生一些不稳定的衍生物,所以应用价值也不大。
以上这些种类载体能在枯草芽孢杆菌中独立自主地复制表达其抗性基因,但是在传代培养中会出现结构不稳定性和分离不稳定性的问题[8]。载体的不稳定性严重限制了其在研究工作中的实际应用,由此造成在枯草芽孢杆菌中分子遗传操作的不便。
由于枯草芽孢杆菌提取质粒和构建质粒较为困难,而在大肠杆菌中操作则很简单方便,所以可通过构建大肠杆菌-枯草芽孢杆菌穿梭载体,先将外源目的基因在大肠杆菌中构建完成后再转入枯草芽孢杆菌中表达。目前常见的大肠杆菌-枯草芽孢杆菌穿梭载体有pEB10[9]、pEB20[10]、pEB60[10]、pUB18[11]、pUB19[12]和pWB980[13]等,详情见表 1。
| 表 1 常见大肠杆菌-枯草芽孢杆菌穿梭载体 Table 1 The common shuttle vector of E.coli-B.Subtilis |
游离型质粒的不稳定性是制约枯草芽孢杆菌高效表达外源蛋白的主要障碍,一般是通过构建整合型载体来克服这一问题。构建枯草芽孢杆菌的整合型载体主要部分包括:大肠杆菌质粒pBR322或其衍生质粒的复制起点,供筛选的抗性标记基因,一段到两段与枯草芽孢杆菌基因组序列同源的基因序列。整合型载体也需要先构建大肠杆菌-枯草芽孢杆菌穿梭载体,再导入枯草芽孢中进行同源交换把表达单元整合到枯草芽孢杆菌染色体上,根据整合方式的不同可以将整合型载体分为同源单交换整合和同源双交换整合。
(1) 同源单交换整合
同源单交换整合是将外源基因某侧和枯草芽孢杆菌基因组同源的序列与目的序列进行同源重组,利用这种方式将外源基因整合到载体上面。单交换时同源臂不要低于25 bp,同源臂达到70 bp时整合效率较高[14]。
(2) 同源双交换整合
同源双交换整合是将外源基因两侧的同源序列与枯草芽孢杆菌基因组目标序列进行同源重组,为了发生有效的同源重组,同源臂必须要在400~500 bp[15],其次还要考虑重组DNA的线性稳定性。一般在构建重组载体时在同源臂中加入Chi位点就可以解决该类问题[4]。
2 芽孢杆菌的σ因子类型在基因转录过程中RNA聚合酶具有重要的作用,它能特异性识别启动子并且与之结合后转录才能开始。RNA聚合酶由5个亚基构成:α亚基、β亚基、β'亚基、ω亚基和一个Sigma(σ)因子[16]。全酶包含5个亚基,核心酶不包括σ因子,σ因子的作用是帮助核心酶识别和结合到启动子的转录起始位点上面。枯草芽孢杆菌表达基因时不同生长阶段的基因表达不同,σ因子出现的种类和数量也不同。目前已知的枯草芽孢杆菌σ因子一共有14种,这些σ因子功能不尽相同,有的与芽孢的形成有关,有的与非重叠、重叠启动子和转录级有关[17-18],具体描述如下(表 2)。
| 表 2 枯草芽孢杆菌σ因子分类及其描述 Table 2 Classification and description of Bacillus subtilis σ factor |
大肠杆菌只含有7种σ因子(σ70、σ54、σ38、σ32、σ28、σ24、σ19),与枯草芽孢杆菌有极大的差别。因此,枯草芽孢杆菌的启动子在大肠杆菌中可能很容易被识别并且表达,反之,大肠杆菌中表达原件大多数不能在枯草芽孢杆菌中使用,因为很难被识别,或者活性低甚至没有活性。
3 枯草芽孢杆菌启动子的结构启动子作为表达系统的一个重要元件,对基因的高水平表达起着关键的调控作用。原核启动子的转录起始位点上游200 bp到下游100 bp范围为启动子的核心区域,该区域的显著特征就是存在小段的保守序列,可以用来预测和分析启动子。对于典型的原核启动子,其保守区域特征有转录起始位点(TSS)、Sextama-35区(TTGACA)、Pri-bmow-10区(TATAAT)[19],Sextama-35区和Pri-bmow-10区序列之间的间隔距离(图 1)。
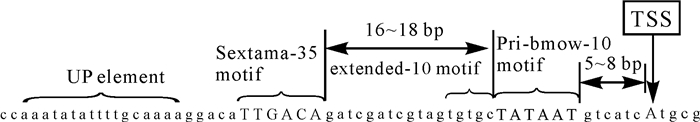
|
图 1 原核启动子核心区域保守序列 Fig.1 The core conservative regional sequence of Prokaryotic |
转录起始位点TSS一般超过90%是嘌呤碱基,CAT序列的中心。几乎所有的启动子TSS上游10 bp处都有一个6 bp的保守区域,这就是Pri-bmow-10区,详细情况为T80A95T45A60A50T96(下标为出现该碱基的最大频率百分数)。由此可知Pri-bmow-10区序列中开始的两个碱基TA和最后一个碱基T高度保守,为该区域最重要的碱基。另外一个6 bp的保守区域为Sextama-35,详细序列为T82T84G78A65C54A45,其碱基的保守性较Pri-bmow-10低[20]。Sextama-35区和Pri-bmow-10区的间隔序列一般在(17±1)bp,也有的小于15 bp或者大于20 bp,尽管间隔区域的序列构成并不重要,但是距离却十分重要,间隔太小或者太大均会不同程度地影响启动子的强度,该距离对影响着RNA聚合酶的几何构像。综上得出,启动子核心区域的保守序列按顺序排列,间隔一般在17 bp左右,这个距离被称为最佳距离。
除此之外,原核启动子的区域内还存在这些“非典型”元件:Pri-bmow-10区延伸序列[21]、UP元件[22]、TSS下游AT富集区[23],与典型特征相比它们的保守性要差一些。实际上,各段特征序列会有灵活的变化。除了Pri-bmow-10区的保守性强一些以外,几乎没有其他哪段序列是必须的。因为丢失了某段特征性弱的序列而损失的启动子活性,可以通过加强其他位置进行一定程度的弥补。如在枯草芽孢杆菌中,丰富某些启动子Sextama-35区上游的某处的AT含量可以增加转录强度[24]。
核糖体结合位点(Ribosomebinding site,RBS),位于起始密码子AUG上游的一段富含嘌呤的DNA序列,也称作Shine-Dalg-arno (SD)序列,该序列在1974年被Shine J和Dalgarno L一起发现[25],所以以此命名。细菌RBS位于细菌mRNA起始密码子AUG上游10 bp的一段富含嘌呤的序列,该序列能识别核糖体16S rRNA并与之进行互补配对,促使mRNA与核糖体结合,从而促进翻译的起始。大肠杆菌的RBS序列为“AAGG”,枯草芽孢杆菌的RBS序列为“GGAGG”,其中以GGAG 4个碱基比较重要。Kozak[26]研究发现突变起始密码子AUG周围的碱基在一定程度上会影响转录和翻译的效率。有研究者通过随机突变RBS提高了目的蛋白的表达量;也有研究者通过对RBS的长度进行增加或者删减达到了高水平调控目的蛋白表达的目的。在线工具RBS Calculator 2.0可以对启动子自身的RBS进行预测和设计。目前利用该软件根据需求设计的RBS进行实验取得了不错的效果[27]。
4 枯草芽孢杆菌表达载体常用的启动子类型枯草芽孢杆菌表达载体常用的启动子按照其诱导的方式可以分为组成型和诱导型启动子[28]。
4.1 组成型启动子组成型启动子是指不需诱导也可以持续表达的启动子,已经报道的枯草芽孢杆菌启动子有Papre、P43、PHpaⅡ[29]、Plapsd等。其中P43启动子为强启动子,在枯草芽孢杆菌中应用最为广泛,它来自枯草芽孢杆菌胞苷脱氨酶(cdd)基因的启动子,有两个重叠的Sextama-35区和Pri-bmow-10区,分别由σA和σB识别[30]。以W800为宿主,以pMA0911-PHpaⅡ为载体表达支链淀粉酶(PUL),最高酶活力可达3.9 U/mL。通过替换P43启动子后pMA0911-P43-PUL同等条件下最高酶活力可达8.7 U/mL[31]。Yang等[32]筛选到一个来自地衣芽孢杆菌的启动子Pshuttle-09,强度为P43的8倍,又在Pshuttle-09基础上构建了一个双启动子Plaps,强度为P43的13倍,是目前在枯草芽孢杆菌表达中启动能力最强的组成型启动子。组成型启动子能高质量表达目的蛋白,成本较低,但同时也存在一些不足之处,如利用组成型启动子表达目的蛋白时不容易控制表达量,而且组成型启动子构建成的载体不稳定,当用一个组成型启动子表达多个外源蛋白的时候,容易造成共抑制和基因沉默等问题。
4.2 诱导型启动子诱导型启动子不同于组成型启动子,它具有独特的优势,可以根据需求给予特定的诱导剂。根据诱导方式可以将诱导型启动子分为两类:1)环境诱导型启动子,该类启动子受低温、热休克、氧气匮乏、渗透压、pH值等因素的影响而做出相应的基因表达来适应环境的变化;2)化学诱导型启动子,可以通过添加诱导物来调节目的基因的表达量。
最常见的是受异丙基-β-D-硫代半乳糖苷(IPTG)诱导的启动子Pspac,该启动子由大肠杆菌lac操纵子和枯草芽孢杆菌噬菌体SPO1融合而成[33]。随后Phan等[34]又在Pspac的基础上优化其调控元件,得到一个启动能力更强的启动子Pgrac100。这些启动子在IPTG的诱导下能高效表达目的蛋白,而且可以控制,但仍存在一些问题:1)IPTG具有毒性,不能用于表达食品或者医疗用途的蛋白产品,而且其价格昂贵,成本偏高;2)Pspac启动子表达外源蛋白量不够理想;3)Pspac启动子不具有严谨可控性,在没有诱导剂添加的时候仍有少许的目的蛋白进行表达。以上这些因素限制了IPTG诱导型启动子在工业上的大规模应用。
以木糖为诱导剂的启动子Pxyl也是枯草芽孢杆菌中常用的诱导型启动子。该启动子来源于巨大芽孢杆菌木糖操纵元件,严格受xylR编码的阻遏蛋白控制。木糖启动子对大部分代谢产物不敏感,受葡萄糖的抑制[35]。木糖价格昂贵,工业应用中成本较高。
蔗糖启动子PsacB是枯草芽孢杆菌第3个常用的启动子系统。该启动子是枯草芽孢杆菌基因组上SACB基因编码的蔗糖果聚糖酶的启动子。PsacB启动子的转录不受蔗糖的严格调控,在没有诱导剂时也能进行转录,但较有蔗糖诱导时强度低100倍[36]。与前两个诱导型启动子相比PsacB启动子强度较弱,不符合工业上大量表达目的蛋白的要求。
除此之外,麦芽糖诱导型启动子Pglv也是枯草芽孢杆菌中应用广泛的诱导型启动子,包括以下3个部分[37]:1)6-磷酸-α-葡萄糖苷酶(glvA)基因,含有449个氨基酸,在NAD(H)(还原型辅酶Ⅰ或辅酶Ⅱ)和二价金属离子存在的情况下才具有活性。2)调控蛋白(glvR)基因,含有254个氨基酸,与RpiR/YebK/YfhH家族相似性很高。3)磷酸烯醇丙酮酸酯-糖磷酸转移系统透性酶(glvC)基因,是一个有12个跨膜区域和527个氨基酸组成的转运酶。Pglv启动子上存在典型的代谢反应元件cre序列,该序列位于启动子的转录起始位点和核糖体的结合位点,它的存在使启动子受葡萄糖特异性抑制,如图 2。
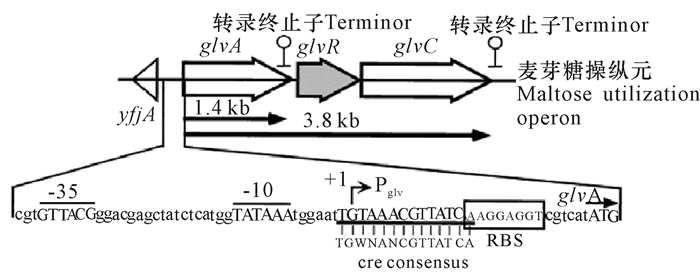
|
图 2 麦芽糖操纵元基因分布与调控元件的保守序列[38] Fig.2 The gene distribution of maltose utilization operon and conserved sequence of the promoter[38] |
Pglv启动子以麦芽糖为诱导物,价格便宜,工业成本低,安全无毒,使用价值比较大。但是发酵中产生的葡萄糖会对启动子有强烈的反馈抑制,这是工业应用上面急需解决的一个问题。
5 枯草芽孢杆菌启动子的克隆和改造 5.1 枯草芽孢杆菌启动子的克隆枯草芽孢杆菌启动子的克隆方法大体可以分为启动子探针筛选法和PCR技术克隆法。
(1) 启动子探针法筛选启动子
启动子探针又称启动子诱捕系统,核心部分的探针质粒载体由一个筛选标记基因和不含启动子成分的报告基因构成,是一种简单有效而且精准的筛选启动子的方法[39]。筛选启动子的时候用合适的限制性内切酶来消化基因组DNA,启动子探针载体也同时用相同的酶切处理,然后将酶切处理好的基因组片段和相同方式处理的启动子探针连接,根据报告基因的表达与否、表达的强弱来确定启动子的筛选情况[40]。在构建探针中报告基因的选取也至关重要,一般要求报告基因序列已知,目的蛋白特性清晰,检测方便快捷,且在宿主中表达时不存在背景干扰。目前常用的报告基因有[41]:1)β-半乳糖苷酶基因,能够水解底物X-gal,可以通过蓝白斑来筛选具有启动子的阳性克隆;2)氯霉素转乙酰酶(CAT)基因,该酶可以使宿主失去抗性,通过检测放射性可确定启动子插入情况;3)绿色荧光蛋白(GFP)基因,无需底物和辅助因子,可以在生物体内高效表达并且发出荧光,对机体无毒性、稳定、检测直观便捷,被广泛用于原核生物和真核生物中[16];4)抗性基因,不需要底物,通过设置抗性梯度可以筛选出强弱不同的启动子。
利用启动子探针系统筛可随机筛选到大量的启动子,简单轻松,但是该方法也存在着工作量大,需要构建启动子文库,后期还要对启动子进行分析等问题。
(2) PCR技术克隆法筛选启动子
该方法结合了现代生物学技术和PCR技术,先用计算机相关软件对基因序列进行分析和预测,然后利用PCR技术获取已知序列中的启动子,该方法逐渐成为获取启动子的研究热点。
目前国内外有很多利用这种方法获取启动子的报道,Wang等[42]利用生物信息学预测到了一些粗略的RNA的转录起始位点,预测分析了该转录起始位点上游500 bp处的序列,并用pri-miRNA PCR(pPCR)技术克隆出该片段,得到了所要的启动子,并在此基础上筛选获得苏云金芽孢杆菌20多个不同时期表达的启动子;黄君健等[43]利用Panhandle PCR技术扩增得到人体外周血单核细胞端粒酶纯化亚基hTERT上游2 090 bp处的DNA序列;王新国等[44]通过将胡萝卜的基因组酶切后,将酶切片段作为模板,利用衔接头PCR方法扩增,成功得到一个新的sⅡ基因启动子序列。
除了以上介绍的几种PCR扩增技术外,常用于扩增启动子序列的PCR技术还有定向反向PCR、交错式热不对称(PCR TAIL-PCR)、Universal Fast Walking(UFW)等。利用PCR技术获得启动子的速度快、特异性强,避免了DNA本身的连接和环化的问题。其缺点是对启动子的预测和分析要求比较精准。
5.2 枯草芽孢杆菌启动子的改造启动子自身构造决定着启动子的转录能力强弱,目前用来表达外源基因的启动子一般是天然的启动子。研究实验表明,改造启动子的主要结构区域,可以不同程度提高启动子的活性[45-46]。目前主要从以下两个思路对启动子进行改造:
(1) 将不同启动子的保守序列进行替换、杂交成为新的启动子
启动子典型的核心保守区域有:①Sextama-35区(Sextama盒),它是RNA聚合酶的识别位点或松弛型结合位点,该区域的结构决定了它和RNA酶的亲和性,因为RNA聚合酶更容易识别强启动子;②Pri-bnow-10区(Pribnow盒)是启动子与RNA聚合酶的紧密结合部位,决定着转录的方向,RNA聚合酶在此区域与DNA序列形成稳定的结合物;③Sextama-35区和Pri-bnow-10的间隔区,该区的序列类型不重要,但是该区的间距长度决定了RNA聚合酶的构型,间距趋近17 bp时启动子启动能力表现为上调,远离17 bp时表现为下调趋势。随着现代生物信息学的发展,我们可以对得到的已知启动子进行预测分析,得出每个启动子核心区域中保守区域的具体情况,然后可以将多个启动子进行优势互补的杂交,融合每个启动子最有优势的核心的保守区域于一个启动子中,从而使启动子的能力得到提高,获得一加一大于二的效果。
(2) 将启动子的特征区域突变为典型启动子的保守区域
不同的启动子都含有保守的区域,与典型的原核启动子相比保守区域可能有所出入。Pri-bnow-10区详细情况为T80A95T45A60A50T96(下标出现该碱基的的最大频率百分数),最开始的两个碱基TA和第6位的碱基T最为保守。Sextama-35区详细序列为T82T84G78A65C54A45, 其中前3个碱基TTG保守性较好。有研究表明通过将启动子的核心区域突变为保守区域,启动子的活性相应得到提高,重组蛋白的表达量也相应地提高了一倍[46]。
6 启动子的分析和预测方法启动子分析和预测方法[47]有很多,如生物信息学预测法、酵母单杂交实验法、染色质免疫共沉淀法、瞬时转染法和凝胶阻滞分析实验法,以下主要介绍生物信息学预测法。
目前,启动子的预测软件有Cpgplot、CpG Prediction、EMBOSS、NNPP、Promoter 2.0、Promoter Scan等。此外,还需要参考许多数据库,如:1)真核生物启动子数据库(EPD),主要收集和RNA聚合酶Ⅱ相关的启动子数据,用户只需输入核苷酸序列就可以查找到启动子的位置,数据库会提供该数据的相关数据链接、转录起始位点以及原始文献[48];2)植物顺式作用调控元件(Plant-CARE)数据库[49],收录的数据有植物顺式作用元件、植物的抑制子和增强子等;3)转录因子(TRANS-FAC)数据库,数据来自人类和酵母的转录因子,是有关转录因子与基因组结合位点的数据库[50];4)Bacterial Promoter, Operon and Gene Finding(BPROM)(bacterial),该数据库能够预测原核生物启动子的结构,此外还具有Single Nucleotide Polymorphisms(SNP)筛选、剪切位点寻找、表达谱分析等功能。现在很多在线工具可以用来分析和预测基因组的启动子、Untranslated Region(UTR)、内含子等结构,如表 3。
| 表 3 启动子生物信息学分析网站 Table 3 Bioinformatic analysis site of the promoter |
生物信息学提供了大量的参考信息,为在线预测和分析启动子奠定了坚实的基础[51]。通过生物信息学的相关软件分析和预测启动子的相关调控元件及其序列,为深入研究启动子结构和功能提供了便利[52]。
7 展望启动子是枯草芽孢杆菌高效表达目的蛋白的关键元件之一,近年来,国内外开展了大量的有关启动子的研究,并取得了较大的进展,获得大批可用于枯草芽孢杆菌的启动子,但是就现有的启动子而言,可以完美用于工业上的启动子数量有限,启动能力和调控方式存在着诸多问题。要获得更多转录强度高、诱导机制方便的启动子,还需要不断深入的研究。
启动子的研究是分子遗传操作十分重要的工作,对于研究枯草芽孢杆菌基因组的功能也有着重要的意义。未来启动子的研究主要集中在两个方面:1)新型高效可控启动子的的发现,这是解决工业和市场生产高要求的最佳的措施;2)对现有启动子的改造,启动子是控制目的基因表达的开关,如果能对启动子进行精确的定位,弄清其各部分元件的工作原理及意义,然后加以改造就能使启动子利用效果最大化。
| [1] |
HARWOOD C R, WIPAT A. Sequencing and functional analysis of the genome of Bacillus subtilis strain 168[J]. Febs Letters, 1996, 389(1): 84-87. DOI:10.1016/0014-5793(96)00524-8 |
| [2] |
IORDǍNESCU S. Three distinct plasmids originating in the same Staphylococcus aureus strain[J]. Arch Roum Pathol Exp Microbiol, 1976, 35(1/2): 111-118. |
| [3] |
SCHUMANN W. Production of recombinant proteins in Bacillus subtilis[J]. Advance in Applied Microbiology, 2007, 62: 137-189. DOI:10.1016/S0065-2164(07)62006-1 |
| [4] |
HARWOOD C R, CUTTING S M. Molecular biological methods for Bacillus[M]. Chichester: John Wiley and Sons Ltd, 1990.
|
| [5] |
VOSKUIL M I, CHAMBLISS G H. The -16 region of Bacillus subtilis and other gram-positive bacterial promoters[J]. Nucleic Acids Res, 1998, 26(15): 3584-3590. DOI:10.1093/nar/26.15.3584 |
| [6] |
ALONSO J C, TAILOR R H. Initiation of plasmid pC194 replication and its control in Bacillus subtilis[J]. Molecular and General Genetics Mgg, 1987, 210(3): 476-484. DOI:10.1007/BF00327200 |
| [7] |
BROSIUSS J, ERFLE M, STORELLA J. Spacing of the-10 and-35 regions in the tac promoter expression.Effect on its in vivo activity[J]. Journal of Biological Chemistry, 1985, 260(6): 3539-3541. |
| [8] |
杜耀华, 王志正. 原核启动子识别研究进展[J]. 生物技术, 2005, 15(5): 80. DU Y H, WANG Z Z. A review for prokaryotic promoter recognition[J]. Biotechnology, 2005, 15(5): 80. |
| [9] |
MITCHELL J E, ZHENG D, BUSBY S J, et al. Identification and analysis of 'extended-10' promoters in Escherichia coli[J]. Nucleic Acids Research, 2003, 31(16): 4689-4695. DOI:10.1093/nar/gkg694 |
| [10] |
ESTREM S T, GAAL T, ROSS W, et al. Identification of an UP element consensus sequence for bacterial promoters[J]. National Academy of Sciences, 1998, 95(17): 9761-9766. DOI:10.1073/pnas.95.17.9761 |
| [11] |
OZOLINE O N, CHASOV V V, DEEV A A, et al. Proximal transcribed regions of bacterial promoters have a non-random distribution of A/T tracts[J]. Nucleic Acids Research, 1999, 27(24): 4768-4774. DOI:10.1093/nar/27.24.4768 |
| [12] |
WILLAMS D M, DUVALL E J, LOVETT P S. Cloning restriction fragmentst that promote expression of a gene in Bacillus subtilis[J]. Journal of Bacteriology, 1981, 146(3): 1162-1165. |
| [13] |
SHINE J, DALGARNO L. The 3' terminal sequence of Escherichia coli 16S ribosomal RNA:Complementarity to nonsenset triplet and ribosome binding sites[J]. Proc Nat Acad Sci USA, 1974, 71(4): 1342-1346. DOI:10.1073/pnas.71.4.1342 |
| [14] |
KOZAK M. Point mutations define a sequence flanking the AUG initiator codon that modulates translation by eukaryotic ribosomes[J]. Cell, 1986, 44: 283-292. DOI:10.1016/0092-8674(86)90762-2 |
| [15] |
TIAN T, SALIS H M. A predictive biophysical model of translational coupling to coordinate and control protein expression in bacterial operons[J]. Nucleic Acids Research, 2015, 43(14): 7137-7151. DOI:10.1093/nar/gkv635 |
| [16] |
KUNST F, OGASAWARA N, MOSZER I, et al. The complete genome sequence of the Gram-positive bacterium Bacillus subtilis[J]. Nature, 1997, 390(6657): 249-256. DOI:10.1038/36786 |
| [17] |
IORDANESCU S, SURDEANU M, DELLA L P, et al. Incompatibility and molecular relationships between small Staphylococcal plasmids carrying the same resistance marker[J]. Plasmid, 1978, 1(4): 468-479. DOI:10.1016/0147-619X(78)90005-7 |
| [18] |
LACEY R W, CHOPRA I. Genetic studies of a multi-resistant strain of Staphylococcus aureus[J]. Journal of Medical Microbiology, 1974, 7(2): 285-297. DOI:10.1099/00222615-7-2-285 |
| [19] |
BRON S, MEIJER W, HOLSAPPEL S, et al. Plasmid instability and molecular cloning in Bacillus subtilis[J]. Research in Microbiology, 1991, 142(7/8): 875-883. |
| [20] |
BRON S, LUXEN E, SWART P. Instability of recombinant pUB110 plasmids in Bacillus subtilis:Plasmid-encoded stability function and effects of DNA inserts[J]. Plasmid, 1988, 19(3): 231-241. DOI:10.1016/0147-619X(88)90041-8 |
| [21] |
NAGARAJAN V, ALBERTSON H, CHEN M, et al. Modular expression and secretion vectors for Bacillus subtilis[J]. Gene, 1992, 114(1): 121-126. DOI:10.1016/0378-1119(92)90717-4 |
| [22] |
WANG L F, WONG S L, LEE S G, et al. Expression and secretion of human atrial natriuretic alpha-factor in Bacillus subtilis using the subtilisin signal peptide[J]. Gene, 1988, 69(1): 39-47. DOI:10.1016/0378-1119(88)90376-9 |
| [23] |
WU S C, WONG S L. Development of improved pUB110-based vectors for expression and secretion studies in Bacillus subtilis[J]. Journal of Biotechnology, 1999, 72(1/2): 185-195. |
| [24] |
MELNIKOV A, YOUNGMAN P J. Random mutagenesis by recombinational capture of PCR products in Bacillus subtilis and Acinetobacter calcoaceticus[J]. Nucleic Acids Research, 1999, 27(4): 1056-1062. DOI:10.1093/nar/27.4.1056 |
| [25] |
KHASANOV F K, ZVINGILA D J, ZAINULLIN A A, et al. Homologous recombination between plasmid and chromosomal DNA in Bacillus subtilis requires approximately 70 bp of homology[J]. Mol Gen Genet, 1992, 234: 494-497. DOI:10.1007/BF00538711 |
| [26] |
KOZAK M. Point mutations define a sequence flanking the AUG initiator codon that modulates translation by eukaryotic ribosomes[J]. Cell, 1986, 44: 283-292. DOI:10.1016/0092-8674(86)90762-2 |
| [27] |
CHÉDIN F, NOIROT P, BIAUDET V, et al. A five-nucleotide sequence protects DNA from exonucleolytic degradation by AddAB, the RecBCD analogue of Bacillus subtilis[J]. Molecular Microbiology, 1998, 29(6): 1369-1377. DOI:10.1046/j.1365-2958.1998.01018.x |
| [28] |
张晓舟. 枯草杆菌新型表达系统和遗传操作体系的建立及应用[D]. 南京: 南京农业大学, 2006. ZHANG X Z. Establishment and application of the new expression and genetic manipulation system in Bacillus subtilis[D]. Nanjing: Nanjing Agricultural University, 2006. http://cdmd.cnki.com.cn/Article/CDMD-10307-2007010271.htm |
| [29] |
王金斌, 陈大超, 李文, 等. 食品级枯草芽孢杆菌表达系统的最新研究进展[J]. 上海农业学报, 2014, 30(1): 115-120. WANG J B, CHEN D C, LI W, et al. The latest research progress of the expression system of edible Bacillus subtilis[J]. Acta Agriculturae Shanghai, 2014, 30(1): 115-120. |
| [30] |
ZHANG X Z, CUI Z L, HONG Q, et al. High-level expression and secretion of methyl parathion hydrolase in Bacillus subtilis WB800[J]. Applied and Environmental Microbiology, 2005, 71(7): 4101-4103. DOI:10.1128/AEM.71.7.4101-4103.2005 |
| [31] |
SONG W, NIE Y, MU X Q, et al. Enhancement of extracellular expression of Bacillus naganoensis pullulanase from recombinant Bacillus subtilis:Effects of promoter and host[J]. Protein expression and purification, 2016, 124: 23-31. DOI:10.1016/j.pep.2016.04.008 |
| [32] |
YANG M M, ZHANG W W, JI S Y, et al. Generation of an artificial double promoter for protein expression in Bacillus subtilis through a pomoter trap system[J]. Plos One, 2013, 8(2): e56321. DOI:10.1371/journal.pone.0056321 |
| [33] |
YANSURA D G, HENNER D J. Use of the Esch-erichia coli lac repressor and operator to control gene expression in Bacillus subtilis[J]. Proc Natl Acad Sci U S A, 1984, 81(2): 439-443. DOI:10.1073/pnas.81.2.439 |
| [34] |
PHAN T T, NGUYEN H D, SCHUMANN W. Development of a strong intracellular expression system for Bacillus subtilis by optimizing promoter elements[J]. Journal of biotechnology, 2012, 157(1): 167-1672. DOI:10.1016/j.jbiotec.2011.10.006 |
| [35] |
KIM L, MOGK A, SCHUMANN W. A xylose-inducible Bacillus subtilis integration vector and its application[J]. Gene, 1996, 181(1/2): 71-76. |
| [36] |
ZUKOWSKI M M, MILLER L. Hyperproduction of an intracellular heterologous protein in a sacUh mutant of Bacillus subtilis[J]. Gene, 1986, 46(2/3): 247-255. |
| [37] |
YAMAMOTO H, SERIZAWA M, THOMPSON J, et al. Regulation of the glv operon in Bacillus subtilis:YfiA (GlvR) is a positive regulator of the operon that is repressed through CcpA and cre[J]. Journal of Bacteriology, 2001, 183(17): 5110-5121. DOI:10.1128/JB.183.17.5110-5121.2001 |
| [38] |
张云雁, 杨明明, 周煌凯, 等. 麦芽糖诱导耐碱性木聚糖酶基因在枯草芽孢杆菌中的表达[J]. 西北农业学报, 2011(20): 14-17, 23. ZHANG Y Y, YANG M M, ZHOU H K, et al. Optimization of expression of alkali-tolerant xylanase introduced by Bacillus subtilis[J]. Acta Agriculturae Boreali-occidentalis Sinica, 2011(20): 14-17, 23. |
| [39] |
GAT O, INBAR I, ALONI-GRINSTERI R, et al. Use of a promoter trap system in Bacillus anthracis and Bacillus subtilis for the development of recombinant protective antigen-based vaccines[J]. Infection and Immunity, 2003, 71(2): 801-813. DOI:10.1128/IAI.71.2.801-813.2003 |
| [40] |
杨燕芳. 枯草芽孢杆菌启动子的克隆及功能分析[D]. 南宁: 广西大学, 2017. YANG Y F. Study on the cloning and functional analysis of Bacillus subtilis promoter[D]. Nanning: Guangxi University, 2017. |
| [41] |
余小霞, 田健, 刘晓青, 等. 枯草芽孢杆菌表达系统及其启动子研究进展[J]. 生物技术通报, 2015, 31(2): 35-44. YU X X, TIAN J, LIU X Q, et al. Research progress of Bacillus subtilis expression system and its promoter regulatory elements[J]. Biotechnology Bulletin, 2015, 31(2): 35-44. |
| [42] |
WANG J P, AI X L, MEI H, et al. High-throughput identification of promoters and screening of highly active promoter-5′-UTR DNA region with different characteristics from Bacillus thuringiensis[J]. PLOS ONE, 2013, 8(5): e62960. DOI:10.1371/journal.pone.0062960 |
| [43] |
黄君健, 李杰之, 林坚, 等. 人端粒酶催化亚基hTERT基因启动子的克隆[J]. 生物技术通讯, 1999, 10(3): 167-170. HUANG J J, LJ J Z, LIN J, et al. Cloning and characterization of human telomerase catalytic subunit (hTERT) gene promoter[J]. Letters in Biotechnology, 1999, 10(3): 167-170. |
| [44] |
王新国, 肖成祖, 张国华, 等. 用衔接头PCR克隆新的胡萝卜Ⅱ型转化酶基因启动子[J]. 中国生物化学与分子生物学报, 2001, 17(1): 61-65. WANG X G, XIAO C Z, ZHANG G H, et al. Isolation of a novel carrot invertaseⅡ promoter by adaptor-PCR[J]. Chinese Journal of Biochemistry and Molecular Biology, 2001, 17(1): 61-65. |
| [45] |
GUAN C R, CUI W J, CHENG J T, et al. Development of an efficient autoinducible expression system by promoter engineering in Bacillus subtilis[J]. Microbial Cell Factories, 2016, 15: 66. DOI:10.1186/s12934-016-0464-0 |
| [46] |
JIAO S, LI X, YU H M, et al. In situ enhancement of surfactin biosynthesis in Bacillus subtilis using novel artificial inducible promoters[J]. Biotechnol and Bioengineering, 2017, 114(4): 832-842. DOI:10.1002/bit.v114.4 |
| [47] |
王秋岩, 何淑雅, 马云, 等. 启动子分析方法的研究进展[J]. 现代生物医学进展, 2015, 15(14): 2794-2800. WANG Q Y, HE S Y, MA Y, et al. Advances in analytical methods of promoter[J]. Progress in Modern Biomedicine, 2015, 15(14): 2794-2800. |
| [48] |
DREOS R, AMBROSINI G, PÉRIER R C, et al. EPD and EPDnew, high-quality promoter resources in the next-generation sequencing era[J]. Nucleic Acids Research, 2013, 41(Database issue): D157-D164. |
| [49] |
ROMBAUTS S, DÉHAIS P, MONTAGU M V, et al. PlantCARE, a plant cis-acting regulatory element database[J]. Nucleic Acids Research, 1999, 27(1): 295-296. DOI:10.1093/nar/27.1.295 |
| [50] |
MATYS V, FRICKE E, GEFFERS R, et al. TRANSFAC®:Transcriptional regulation, from patterns to profiles[J]. Nucleic Acids Research, 2003, 31(1): 374-378. DOI:10.1093/nar/gkg108 |
| [51] |
管晓翔, 陈巍魏, 陈龙邦, 等. p27Kip1基因启动子区的生物信息学分析[J]. 医学研究生学报, 2010, 23(10): 1029-1032. GUAN X X, CHEN W W, CHEN L B, et al. Bioinformatic analysis of the p27Kip1 promoter region[J]. J Med Postgra, 2010, 23(10): 1029-1032. DOI:10.3969/j.issn.1008-8199.2010.10.005 |
| [52] |
张洪渊. 生物化学原理[M]. 北京: 科学出版社, 2008. ZHANG H Y. Principles of biochemistry[M]. Beijing: Science Press, 2008. |
 2018, Vol. 25
2018, Vol. 25 


