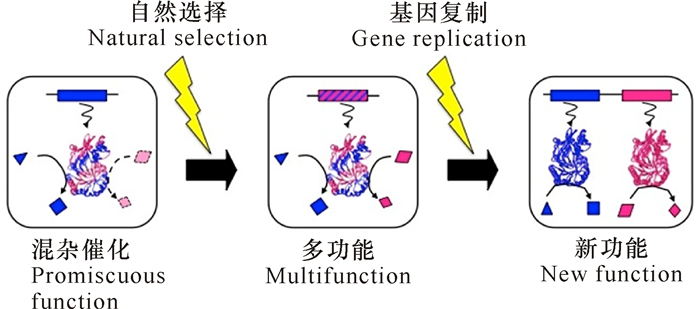
酶催化生物体内化学反应,是生命形成运转的重要动力来源。传统观点认为酶催化化学反应是非常精确专一的,然而越来越多的研究表明酶可以具有多种兼职功能,酶蛋白具备除其生理活性以外其他活性的特性称为酶催化功能的混杂性(Enzyme promiscuity)。事实上,酶的混杂性是新功能酶演化的基础,对生物体适应环境具有重要意义。Jensen[1]于上世纪70年代提出祖先酶蛋白可能具有多种催化功能,在过去的几十亿时间里生物体内的酶蛋白一直处于演化过程中,伴随着基因复制、突变和环境选择,新的催化功能可能在几年甚至几个月的时间内出现,例如细菌对抗生素的耐药性及对环境人工合成化学品的生物降解。Jensen的模型(图 1)强调了酶的多功能性是新功能演化的重要起点,基于此,许多科学家认为酶的混杂性具有普适性,即绝大部分(或者全部)酶蛋白都可能具有多种功能[2-3]。当前有关酶的混杂性研究除尝试阐释其对生物适应性进化的贡献外,还关注其开发利用前景,通过蛋白质工程手段在实验室内定向进化酶的混杂催化功能,以推进其在环境治理、工业催化等领域的应用。本文就酶催化功能混杂性的普遍性、分子机理、可进化性等方面的相关研究进展进行综述。
|
图 1 基于混杂活性的酶新功能演化示意 Fig.1 Schematic representation of new functional evolution of enzymes based on promiscuous activity |
酶具有混杂催化活性是生物适应环境的基础,为适应环境,生物体内的酶将处于不断演化状态。根据Jensen提出的酶演化模型,现代酶由具有多种功能的祖先蛋白进化而来,与此同时每个现代酶都有可能作为演化模板参与生物适应环境演化过程。因此,具有混杂活性很可能是酶蛋白的共性特征。进化生物化学家Copley[3]推测每个酶蛋白可能催化10种以上的反应,细菌体内有约2 000个基因,这样一个细菌细胞可能具有20 000种混杂活性,这就赋予了细菌更新演化适应多变环境的潜能。事实上,大规模的生物信息学统计和生化实验研究都表明现有的酶蛋白家族成员存在催化功能多样性特征。
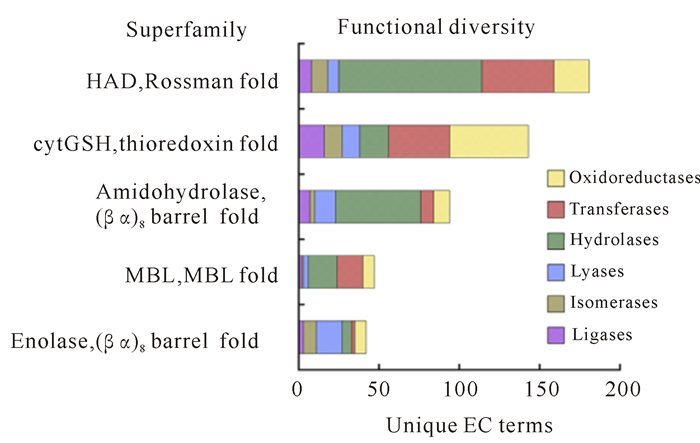
按照催化反应类型,酶可分为六类:氧化还原酶、转移酶、水解酶、裂解酶、异构酶和连接酶,对应于国际酶学委员会编号EC 1~6。Baier等[4]挑选统计了5个酶蛋白超家族(脱卤酶、谷胱甘肽转移酶、氨基水解酶、β-内酰胺酶和烯醇酶)成员的催化功能,每个蛋白家族包含13 000~91 000条蛋白序列,发现尽管同一蛋白超家族成员具备相近的蛋白折叠模式,其功能可能包含了所有六类催化反应(图 2)。Furnham等[5]构建了379个蛋白超家族的系统发育关系,在此基础上分析所有蛋白超家族内成员催化功能的变化,其研究表明尽管一类催化反应可能会主导某一蛋白超家族(81%的催化功能的变化是在同类化学反应内部发生不同程度的调整),但是不同催化反应类型之间的转换,即EC大类编号发生变化也时有发生。大规模计算生物学统计说明相同折叠模式结构的蛋白其催化功能并非处于固化状态,而是动态可变的。
在实验验证研究方面,为表征酶催化功能多样性,多个研究组挑选蛋白超家族内具有代表性的数十个酶蛋白进行重组表达并测定其对多样底物的催化活性。所研究的蛋白超家族包括谷胱甘肽转移酶[6]、β-酮酸裂解酶[7]、β-内酰胺酶[8]和脱卤酶[9]超家族,结果都表明上述超家族酶蛋白成员在体外普遍能催化多种底物反应。以脱卤酶为例,Huang等[9]表达脱卤酶超家族的217个蛋白,同时用167种磷酸化底物进行活性筛选,发现平均每个酶蛋白可作用15.5个底物,且其中50个酶能作用40个以上的底物,另外有一个酶能对143个底物分子起作用。
2 酶混杂活性的分子基础由于未经过自然压力的充分选择,相较于其生理活性,酶的混杂活性往往效率较低。例如从海洋细菌中发现一种脯氨酸肽酶具有混杂水解有机磷化合物的活性,其肽酶催化效率常数(kcat/Km)是磷酸三酯酶的100余倍[10]。通常酶行使混杂功能使用与天然功能相同的活性中心,催化机制也可能接近[11]。混杂活性的形成机制一方面可促进从分子层面理解酶蛋白的演化过程,另一方面也为蛋白质工程研究者提供理论依据。
2.1 底物结合模式混杂底物与酶的结合方式往往是非最优化的,即酶-底物复合物构象无法最大程度降低反应活化能,从而导致酶的混杂活性相对较低。限制酶与底物相互作用的因素包括电离状态[12]、底物结合方式[13]等。来源于苏云金芽孢杆菌β-内酰胺酶AiiA可混杂催化农药对氧磷水解,将AiiA与对氧磷复合物进行分子动力学模拟发现,对氧磷与活性中心的结合构象不利于活化水分子攻击磷酸三酯键[14]。定向进化使得AiiA残基Phe68的构象发生变化,这种构象细微调整形成堆积效应让底物对氧磷更加适宜与活性中心相互作用,进而其催化效率得到1 000倍的提高。人血清酯酶PON1水解内酯的同时也能混杂水解磷酸三酯。内酯水解发生在PON1蛋白闭合状态,残基His115、Glu53与负责进攻内酯键的羟基发生作用。分子对接发现与内酯的结合不同,混杂底物对氧磷体积较大,只能与底物腔开放状态的PON1蛋白结合并嵌入至活性中心,处于开放状态的蛋白残基His115不再参与催化反应[15]。
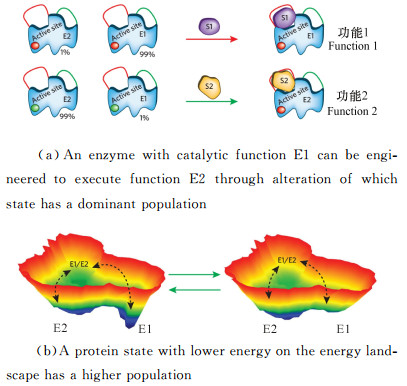
2.2 蛋白结构的动态性混杂活性还可能与蛋白质结构的动态性相关,即酶蛋白分子可能具有多种构象,这些不同构象的分子可能具有不同的催化活性[3, 16]。该现象最早被Lu等[17]在对胆固醇氧化酶催化过程的单分子实验观察到,他们发现由于活性中心构象的柔性,单个胆固醇氧化酶分子之间的催化速率存在500倍的差异。后续研究证实酶的确是以蛋白混合物状态存在的,混合物中各分子在部分区域具有不同构象[18-20],仅一部分构象的蛋白分子能高效结合、催化某一底物。蛋白结构动态性观点认为酶的混杂活性不高是因为混杂底物只能与极少一部分构象的蛋白发生结合、反应[21]。如图 3,突变引起的酶催化功能的改变可被看做是由一种构象占优势的酶分子混合体转变为另一种构象占优势的新的混合体,这种不同构象蛋白分子组分变化源于突变引起的构象自由能变化(混合体中能量低的构象倾向于占较高的比例)[16]。Campbell等[22]和Kaltenbach等[23]在体外构建了酯酶逐步向有机磷水解酶转变的系列突变,通过比较进化轨迹中不同阶段突变酶的蛋白结构,证实远离催化中心位点的改变会扰动蛋白的动力学构象组分,处于演化中期的多功能酶蛋白关键区域的B-factor值越高,对应于构象形式越多样。
|
图 3 图 3不同构象酶蛋白分子组分变化引起的功能变化示意图[16] Fig.3 Schematic diagram of functional changes caused by changes in the molecular composition of different conformational enzymatic proteins |
酶蛋白的混杂活性具有可进化性(evolvability)。蛋白质演化是固定并遗传有益于适应环境突变表型的过程,在进化过程中很多突变属于中性突变并不影响表型,即具有鲁棒性(robustness),另一方面单个或几个碱基的改变可能形成完全不同的表型,即具有可变性(innovability),鲁棒性与可变性在蛋白进化过程中共存,共同组成蛋白质的可进化性[11]。Tóth-Petróczy等[24]认为“鲁棒-可变”矛盾二元体都对酶蛋白的进化具有积极作用,鲁棒性的意义可能在于为适应潜在环境的其他改变提供进化起点[25]。蛋白质可进化性及其在适应性进化过程中的意义理论体系仍然处于形成发展阶段,有待具体实验数据反复验证。例如酶蛋白在进化过程中,天然活性与混杂活性之间的变化规律是如Khersonsky等[11]描述的天然活性往往在进化初期对突变不敏感,还是如Kaltenbach等[26]观察到的混杂活性的提升需以天然活性的快速损失为代价?蛋白质结构的稳定性是利于进化还是阻碍进化[27-28]?追踪表征蛋白质功能分化过程可为研究其可进化性提供直接数据。
在实验室内获取处于不同进化阶段的蛋白变体对象是研究其功能演化的关键。目前主要有两类方法:一是基于生物信息学手段模拟计算祖先蛋白序列,如Nguyen等[29]重建30亿年跨度的厚壁菌腺苷酸激酶祖先序列,揭示祖先蛋白适应远古高温环境的分子基础及进化轨迹;另一种方法属于现代蛋白质工程技术,通过构建随机突变文库,在人工设计的选择压力下模拟自然界蛋白进化过程,从突变文库中筛选处于不同进化阶段的蛋白模型[30],Bloom等[27]应用易错PCR构建P450氧化酶突变文库,比较具有不同稳定性母本蛋白的突变体对各类底物的催化效率,阐释了该酶稳定性与可进化性之间的关系。从生物化学角度研究不同来源蛋白适应进化的过程与机制将会有助于充实蛋白质进化理论内涵,更可加深对整个生物体适应环境分子基础的认知。
4 展望达尔文进化论在宏观层面展示了生物表型演化的客观规律。随着生物学的发展,人们逐渐认识到表型是基因及其编码产物的表现形式,酶分子作为生命代谢流运转的引擎,其演化过程对生物适应环境发挥关键作用。从这个意义上讲,酶混杂催化功能的研究是达尔文进化论在分子层面的精细解读。当前国际对酶的混杂性研究仍然处于理论形成阶段,美国与以色列科学家在该领域已有深厚理论积累,在揭示酶演化过程中的异位显性[31-32]、可进化性[24]、结构动态性[25]对功能分化影响方面做出了先锋性研究。国内对酶的混杂性领域研究涉足较少,一些文献将酶的混杂性与酶非水相催化、酶对非关键基团差异的同类型底物催化概念混淆。基于本文论述,酶混杂性研究将集中突破以下几方面:一是结合结构生物学与催化机制的大规模蛋白超家族生物信息学研究,阐释酶分子功能、结构和机理的演化轨迹;二是实验室模拟自然进化,人工创造酶蛋白不同演化阶段分子并表征其生化、生物物理学特性,以探讨其内在规律与机理;三是酶混杂活性的应用,蛋白质工程技术改良酶的混杂活性,并推进其在工业、环境领域的应用,尤其在药物分子特殊化学键的酶法合成及环境污染物的降解修复方面具有重要的开发潜力。
| [1] |
JENSEN R A. Enzyme recruitment in evolution of new function[J]. Annual Review of Microbiology, 1976, 30: 409-425. DOI:10.1146/annurev.mi.30.100176.002205 |
| [2] |
NEWTON M S, ARCUS V L, GERTH M L, et al. Enzyme evolution:Innovation is easy, optimization is complicated[J]. Curr Opin Struct Biol, 2018, 48: 110-116. DOI:10.1016/j.sbi.2017.11.007 |
| [3] |
COPLEY S D. Shining a light on enzyme promiscuity[J]. Curr Opin Struct Biol, 2017, 47: 167-175. DOI:10.1016/j.sbi.2017.11.001 |
| [4] |
BAIER F, COPP J N, TOKURIKI N. Evolution of enzyme superfamilies:Comprehensive exploration of sequence-function relationships[J]. Biochemistry, 2016, 55(46): 6375-6388. DOI:10.1021/acs.biochem.6b00723 |
| [5] |
FURNHAM N, DAWSON N L, RAHMAN S A, et al. Large-scale analysis exploring evolution of catalytic machineries and mechanisms in enzyme superfamilies[J]. Journal of Molecular Biology, 2016, 428(2 Pt A): 253-267. |
| [6] |
MASHIYAMA S T, MALABANAN M M, AKIVA E, et al. Large-scale determination of sequence, structure, and function relationships in cytosolic glutathione transferases across the biosphere[J]. Plos Biology, 2014, 12(4): e1001843. DOI:10.1371/journal.pbio.1001843 |
| [7] |
BASTARD K, SMITH A A T, VERGNE-VAXELAIRE C, et al. Revealing the hidden functional diversity of an enzyme family[J]. Nature Chemical Biology, 2014, 10(1): 42-49. DOI:10.1038/nchembio.1387 |
| [8] |
BAIER F, TOKURIKI N. Connectivity between catalytic landscapes of the metallo-beta-lactamase superfamily[J]. Journal of Molecular Biology, 2014, 426(13): 2442-2456. DOI:10.1016/j.jmb.2014.04.013 |
| [9] |
HUANG H, PANDYA C, LIU C, et al. Panoramic view of a superfamily of phosphatases through substrate profiling[J]. Proc Natl Acad Sci USA, 2015, 112(16): E1974-E1983. DOI:10.1073/pnas.1423570112 |
| [10] |
XIAO Y Z, YANG J, TIAN X P, et al. Biochemical basis for hydrolysis of organophosphorus by a marine bacterial prolidase[J]. Process Biochemistry, 2017, 52: 141-148. DOI:10.1016/j.procbio.2016.10.008 |
| [11] |
KHERSONSKY O, TAWFIK D S. Enzyme promiscuity:A mechanistic and evolutionary perspective[J]. Annual Review of Biochemistry, 2010, 79: 471-505. DOI:10.1146/annurev-biochem-030409-143718 |
| [12] |
WANG S C, JOHNSON W H Jr, WHITMAN C P. The 4-oxalocrotonate tautomerase- and YwhB-catalyzed hydration of 3E-haloacrylates:Implications for the evolution of new enzymatic activities[J]. Journal of the American Chemical Society, 2003, 125(47): 14282-14283. DOI:10.1021/ja0370948 |
| [13] |
LU X, LI L, WU R, et al. Kinetic analysis of Pseud-omonas aeruginosa arginine deiminase mutants and alternate substrates provides insight into structural determinants of function[J]. Biochemistry, 2006, 45(4): 1162-1172. DOI:10.1021/bi051591e |
| [14] |
YANG G, HONG N, BAIER F, et al. Conformational tinkering drives evolution of a promiscuous activity through indirect mutational effects[J]. Biochemistry, 2016, 55(32): 4583-4593. DOI:10.1021/acs.biochem.6b00561 |
| [15] |
BEN-DAVID M, ELIAS M, FILIPPI J J, et al. Catalytic versatility and backups in enzyme active sites:The case of serum paraoxonase 1[J]. Journal of Molecular Biology, 2012, 418(3/4): 181-196. |
| [16] |
MA B, NUSSINOV R. Protein dynamics:Conforma-tional footprints[J]. Nature Chemical Biology, 2016, 12(11): 890-891. DOI:10.1038/nchembio.2212 |
| [17] |
LU H P, XUN L, XIE X S. Single-molecule enzymatic dynamics[J]. Science, 1998, 282(5395): 1877-1882. DOI:10.1126/science.282.5395.1877 |
| [18] |
ENGLISH B P, MIN W, VAN OIJEN A M, et al. Ever-fluctuating single enzyme molecules:Michaelis-Menten equation revisited[J]. Nature Chemical Biology, 2006, 2(2): 87-94. DOI:10.1038/nchembio759 |
| [19] |
CALLENDER R, DYER R B. The dynamical nature of enzymatic catalysis[J]. Accounts of Chemical Research, 2015, 48(2): 407-413. DOI:10.1021/ar5002928 |
| [20] |
MASTERSON L R, SHI L, METCALFE E, et al. Dynamically committed, uncommitted, and quenched states encoded in protein kinase:A revealed by NMR spectroscopy[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(17): 6969-6974. DOI:10.1073/pnas.1102701108 |
| [21] |
JAMES L C, TAWFIK D S. Conformational diversity and protein evolution-a 60-year-old hypothesis revisited[J]. Trends in Biochemical Sciences, 2003, 28(7): 361-368. DOI:10.1016/S0968-0004(03)00135-X |
| [22] |
CAMPBELL E, KALTENBACH M, CORREY G J, et al. The role of protein dynamics in the evolution of new enzyme function[J]. Nature Chemical Biology, 2016, 12(11): 944-950. DOI:10.1038/nchembio.2175 |
| [23] |
KALTENBACH M, JACKSON C J, CAMPBELL E C, et al. Reverse evolution leads to genotypic incompatibility despite functional and active site convergence[J]. Elife, 2015, 4: e06492. |
| [24] |
TÓTH-PETRÓCZY Á, TAWFIK D S. The robustness and innovability of protein folds[J]. Curr Opin Struct Biol, 2014, 26: 131-138. DOI:10.1016/j.sbi.2014.06.007 |
| [25] |
TOKURIKI N, TAWFIK D S. Protein dynamism and evolvability[J]. Science, 2009, 324(5924): 203-207. DOI:10.1126/science.1169375 |
| [26] |
KALTENBACH M, EMOND S, HOLLFELDER F, et al. Functional trade-offs in promiscuous enzymes cannot be explained by intrinsic mutational robustness of the native activity[J]. PLoS Genet, 2016, 12(10): e1006305. DOI:10.1371/journal.pgen.1006305 |
| [27] |
BLOOM J D, LABTHAVIKUL S T, OTEY C R, et al. Protein stability promotes evolvability[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(15): 5869-5874. DOI:10.1073/pnas.0510098103 |
| [28] |
DEPRISTO M A, WEINREICH D M, HARTL D L. Missense meanderings in sequence space:A biophysical view of protein evolution[J]. Nature Reviews Genetics, 2005, 6(9): 678-687. DOI:10.1038/nrg1672 |
| [29] |
NGUYEN V, WILSON C, HOEMBERGER M, et al. Evolutionary drivers of thermoadaptation in enzyme catalysis[J]. Science, 2017, 355(6322): 289-294. DOI:10.1126/science.aah3717 |
| [30] |
ROMERO P A, ARNOLD F H. Exploring protein fit-ness landscapes by directed evolution[J]. Nature Reviews Molecular Cell Biology, 2009, 10(12): 866-876. DOI:10.1038/nrm2805 |
| [31] |
STARR T N, THORNTON J W. Epistasis in protein evolution[J]. Protein Science, 2016, 25(7): 1204-1218. DOI:10.1002/pro.2897 |
| [32] |
STEINBERG B, OSTERMEIER M. Shifting fitness and epistatic landscapes reflect trade-offs along an evolutionary pathway[J]. Journal of Molecular Biology, 2016, 428(13): 2730-2743. DOI:10.1016/j.jmb.2016.04.033 |
 2018, Vol. 25
2018, Vol. 25