2. 广西大学生命科学与技术学院, 广西南宁 530004
2. College of Life Science and Technology, Guangxi University, Nanning, Guangxi, 530004, China
环糊精水解酶属于糖苷水解酶13家族成员,具有水解环糊精中α-1,4-糖苷键能力[1]。环糊精水解酶具有广泛的底物特异性,能水解环糊精、普鲁兰糖、可溶性淀粉及支链淀粉等多种底物[2]。但是环糊精水解酶在淀粉工业上的应用并不广泛。研究发现,不同微生物来源的环糊精水解酶对底物作用的专一性不同,主要表现在对各种支链低聚糖的分解能力上[3-4]。环糊精水解酶在底物识别方面存在一个共同的问题:它们对小分子底物的亲和力高,对大分子底物的亲和力弱,因而对小分子环糊精具有很强的水解能力,对支链淀粉水解能力很弱[5]。较低的水解效率阻碍环糊精水解酶在水解支链淀粉上的实际应用。
cds1-3是从宏基因组文库中筛选得到的一个新环糊精水解酶。cds1-3的底物特异性异于传统环糊精水解酶,对支链淀粉的水解能力是水解环糊精能力的1.9倍[6]。cds1-3与模式菌Thermus sp.IM6501的环糊精水解酶ThMA[7]氨基酸序列一致性高达94.7%,仅存在30个氨基酸差异。但两者对环糊精和支链淀粉的作用能力却有较大的差别:ThMA对环糊精等小分子物质的作用力最强,对支链淀粉的水解能力最弱;而cds1-3对支链淀粉的水解能力却很强[6-7]。本研究以环糊精水解酶酶ThMA(id为O69007)的晶体结构1SMA为模板,对cds1-3进行同源建模并根据构建好的结构模型对支链淀粉酶cds1-3进行空间结构分析,选取底物通道相关氨基酸进行定点突变,比较突变酶和野生酶的功能差异,定位决定cds1-3特殊功能的氨基酸。
1 材料与方法 1.1 材料大肠杆菌XL10-Gold和DH5α由实验室保存,表达质粒pSE380-cds1-3由实验室构建保存。质粒提取试剂盒、胶回收试剂盒购自Tiangen公司;分子生物学工具酶包括限制性内切酶、PrimeSTAR HS DNA聚合酶、DNA Marker和Protein Marker等购自TaKaRa公司;α-环糊精购自上海生物工程有限公司;支链淀粉购自Sigma公司;可溶性淀粉购自国药集团化学试剂有限公司;IPTG购自Aldrich公司;Yeast extract和Tryptone购自Oxoid公司;其他试剂均为国产分析纯试剂;寡聚核苷酸引物由Invitrogen公司合成。
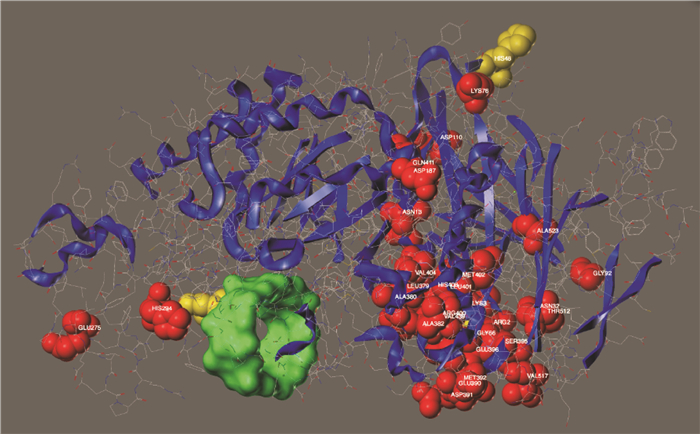
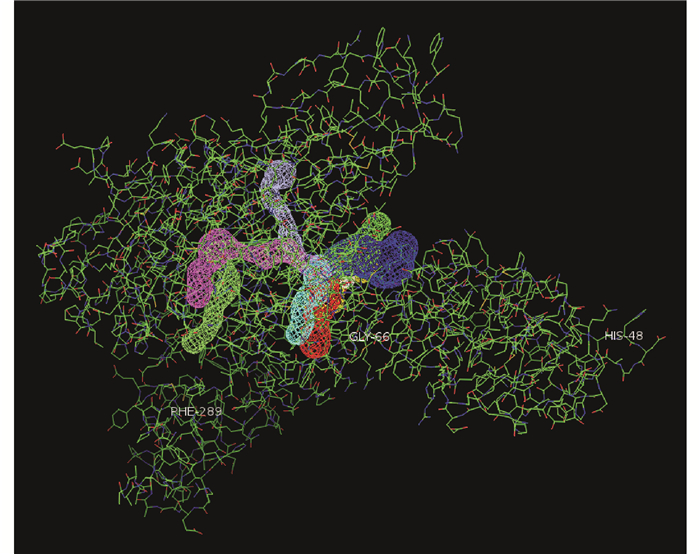
1.2 突变体的构建 1.2.1 突变位点选择以氨基酸序列高度一致的模式菌Thermus sp.IM6501的环糊精水解酶ThMA(id为O69007)的晶体结构1SMA为模板,利用M4T server进行同源建模构建cds1-3的三维结构模型[8]。通过cds1-3和ThMA氨基酸序列比对,以及cds1-3同源建模结构分析,发现序列高度相似、三维结构外观一致的两个酶对大分子底物降解存在较大的差异。为探求造成这一差异的具体原因,采用sybyl 1.2和PyMOL软件工具进行蛋白质底物结合分析及蛋白结构显示,从ThMA蛋白质和cds1-3蛋白质的差异中选取和多聚体形成、底物结合以及底物通道相关的氨基酸进行模拟分析(图 1~2)。由图 1可知,除黄色球体表示的289位氨基酸外,其余球状体均为差异氨基酸。绿色环状体为环糊精底物分子,示意底物环糊精的结合位置。差异氨基酸主要位于远离底物结合位点的底物通道区域,只有少数几个氨基酸在底物结合区域附近。由图 2可知,Glu66、Pro48、Phe289 3个氨基酸位点在空间上距离底物通道远近具有差异,分属不同的空间结构,这3个位点所属的空间结构对酶与底物的结合和功能影响可通过对3个氨基酸位点进行突变研究来确定。cds1-3蛋白序列中的Glu66、Pro48、Phe289这3个氨基酸对应ThMA蛋白序列的Gly66、His48、Ala289氨基酸。为找到决定cds1-3功能差异的关键氨基酸,选择Glu66、Pro48、Phe289为突变位点,构建突变体E66G、P48H、F289A。通过研究突变后突变酶的底物特异性变化,缩小cds1-3的功能差异关键氨基酸的选择范围,为后续进一步选点突变研究提供参考。
|
| 图 1 cds1-3蛋白质结构模拟分析 Fig. 1 Structure simulation analysis of protein cds1-3 |
|
| 图 2 cds1-3酶的底物通道模拟 Fig. 2 Substrate channel simulation of cds1-3 enzyme |
1.2.2 定点突变
利用PCR等技术向模板DNA片段导入所需变化的定点突变,是常用的体外诱变技术。本研究采用核苷酸引物介导的定点突变方法对环糊精水解酶cds1-3进行定点突变,此方法关键在于引物设计的合理与否。定点突变引物设计要遵循一定的原则和注意事项[9]。本实验的引物设计如表 1所示。
| Primer | Sequences |
| 3P48H-f | 5′-CGAATGGCACGATGGCGCCTGGCAGTTTG-3′ |
| 3P48H-r | 5′-GCGCCATCGTGCCATTCGTACGGGTCGCC-3′ |
| F289A-f | 5′-CGACACAGCAGCGTTCGTGCCGCAAATGCCC-3′ |
| F289A-r | 5′-CACGAACGCTGCTGTGTCGTAATTCGGGCGCGG-3′ |
| E66G-f | 5′-GAAGCGACGGCTTGTTTGACTATTGGCTCGCCG-3′ |
| E66G-r | 5′-CAAACAAGCCGTCGCTTCCCGTTTTCCGCATCGG-3′ |
PCR体系参照TaKaRa的PrimeSTAR HS DNA Polymerase的说明书。PCR反应程序:98℃,3 min;98℃ 10 s,55℃ 15 s,72℃ 6 min,30个循环;72℃,10 min。
模板消化:PCR结束后,每20 μL扩增产物加1 μL Dpn Ⅰ,混匀,37℃消化模板质粒1 h。
取消化产物转化大肠杆菌XL10-Gold,涂布氨苄抗性平板,过夜培养。挑取6株单菌落提取质粒,送华大基因公司进行测序。
1.3 突变酶的表达与纯化所有突变体经送测序验证为正确突变后,转化到大肠杆菌DH5α中,构建重组菌,分别命名为E66G、P48H、F289A。重组菌经IPTG诱导表达,收集菌体经超声破胞离心后定性测定上清中的酶活力,有酶活的上清继续用镍柱亲和层析法除去大部分的杂蛋白。然后用HiTrap Desalting预装柱脱盐(洗脱buffer为50 mmol/L pH值6.5的NaH2PO4-Na2HPO4缓冲液)、置换缓冲液,超滤管浓缩。最后再用HiTrap Q FF阴离子交换柱进行纯化、浓缩,得到纯酶(平衡液:50 mmol/L pH值6.5的NaH2PO4-Na2HPO4缓冲液,洗脱液:含1 mol/L NaCl的50 mmol/L pH值6.5的NaH2PO4-Na2HPO4缓冲液)。
1.4 酶活力的测定酶活力测定:往EP管中加入500 μL底物(底物用100 mmol/L pH值5.5的乙酸-乙酸钠缓冲液溶解),加入适当浓度的酶液10 μL,混匀,置于50℃水浴锅中反应30 min,然后立即加入1 mL DNS,沸水浴5 min;反应结束后冷却到室温,并测定OD540,进而计算出酶反应所产还原糖的量。
酶活定义:在反应条件下,每分钟支链淀粉酶水解底物产生的1 μmol还原糖(用葡萄糖来计算)所需的酶量为一个酶活单位。
1.5 突变酶与原始酶的底物特异性比较分别以α-环糊精、普鲁兰糖、可溶性淀粉、木薯淀粉和支链淀粉作为底物,测定突变酶与cds1-3对不同底物的降解效果,比较分析突变酶与cds1-3的底物特异性差异。
1.6 协同作用比较将地衣芽孢杆菌α-淀粉酶分别与cds1-3、突变酶按酶活1:0、1:1、1:2、1:3、1:4、1:5的比例混匀,加入用500 μL 100 mmol/L pH值5.5的乙酸-乙酸钠缓冲液配制的1%底物(α-环糊精、普鲁兰糖、可溶性淀粉、支链淀粉、木薯淀粉),50℃反应1 h,用DNS测定还原糖含量计算协同率。
2 结果与分析 2.1 突变酶的构建、表达与纯化定点突变构建得到突变体E66G、P48H、F289A,重组菌经过诱导表达,菌体经超声破胞离心后定性测定上清中的酶活力,发现只有突变体E66G有酶活。突变体E66G经诱导表达、纯化后得到纯酶。由SDS-PAGE电泳分析图(图 3)可知,经镍柱亲和层析、脱盐浓缩及阴离子交换后获得突变酶E66G的纯酶,为单一蛋白质条带,大小约为65 kDa,与原始酶cds1-3大小一致。

|
| M:标准蛋白Marker; 1:镍柱纯化后的E66G;2:离子交换后超滤浓缩的E66G M:Normalized protein Marker; 1:The purified enzyme E66G by Ni-chelating affinity chromatography; 2:The concentration of mutational enzyme E66G after IEC 图 3 突变酶E66G的SDS-PAGE电泳分析 Fig. 3 The SDS-PAGE electrophoresis analysis of mutational enzyme E66G |
由表 2可知,粗酶液经镍柱亲和层析,突变酶比活力由16.07 U/mg提高到83.68 U/mg,纯化倍数为5.21倍,回收率为51%。经HiTrap Q FF阴离子交换纯化,得到重组酶的比活力达到111.85 U/mg,纯化倍数为6.96。纯化后的突变酶E66G比活力是原始酶cds1-3纯酶的98.2%,在95%的可信度范围内认为两者比活力没有区别。
| 纯化步骤 Purification step |
总蛋白 Total protein (mg) |
总活力 Total enzyme activity (U) |
比活力 Specific activity (U/mg) |
纯化倍数 Purification fold |
回收率 Recovery rate (%) |
| 初始粗酶液 Crude enzyme |
52.93 | 850.62 | 16.07 | 1.00 | 100.00 |
| 镍柱纯化 Nickel purification |
5.19 | 433.86 | 83.68 | 5.21 | 51.00 |
| 阴离子交换 Anion exchange |
0.84 | 94.29 | 111.85 | 6.96 | 11.08 |
2.2 突变酶与原始酶的底物特异性比较
以降解α-环糊精的酶活力为100%,将突变酶E66G和原始酶cds1-3的底物特异性进行列表比较,并以cds1-3的相对酶活力为参照计算E66G的相对酶活增长率(表 3)。由表 3可知:E66G和cds1-3对不同底物降解能力相差较大。降解能力大小排序依次为α-环糊精>可溶性淀粉>普鲁兰糖≈支链淀粉≈木薯淀粉。突变酶E66G降解可溶性淀粉和小分子底物普鲁兰糖的能力下降,对大分子底物木薯淀粉和支链淀粉的降解能力有所提高。其中,对木薯淀粉的增长率为26.96%,对支链淀粉的增长率为23.15%。
| 底物 Substrate |
cds1-3相对酶活力 cds1-3 relative enzyme activity (%) |
E66G相对酶活力 E66G relative enzyme activity (%) |
相对酶活力增长率 Growth rate of relative enzy me activity (E66G-cds1-3)/cds1-3 |
| α-环糊精 α-cyclodextrin |
100 | 100 | - |
| 普鲁兰糖 Pullulan |
8.78 | 7.63 | -13.09 |
| 木薯淀粉 Cassava starch |
6.22 | 7.90 | 26.96% |
| 可溶性淀粉 Soluble starch |
28.8 | 26.28 | -8.74% |
| 支链淀粉 Amylopectin |
6.9 | 8.49 | 23.15% |
2.3 协同作用
协同率为两个酶协同作用时所产的还原糖与单酶反应时所产还原糖含量的比值。由表 4可知,与地衣芽孢杆菌α-淀粉酶协同作用降解小分子底物时,突变酶E66G跟原始酶cds1-3协同率都小于1,而且协同率相近,表明原始酶和突变酶在水解小分子底物时结果相似。但对于大分子底物,尤其是支链淀粉,其产生的协同率明显大于1,且与原始酶cds1-3协同作用结果相比而言,也有较明显的增加,表明突变酶E66G对大分子底物的作用能力比原始酶高。
| 底物 Substrate |
cds1-3与α-淀粉酶协同率 Collaborative rate of cds1-3 and α-amylase |
E66G与α-淀粉酶协同率 Collaborative rate of E66G and α-amylase |
|||||||||
| 1:1 | 1:2 | 1:3 | 1:4 | 1:5 | 1:1 | 1:2 | 1:3 | 1:4 | 1:5 | ||
| α-环糊精 α-cyclodextrin |
0.78 | 0.76 | 0.69 | 0.82 | 0.64 | 0.66 | 0.77 | 0.80 | 0.73 | 0.73 | |
| 普鲁兰糖 Pullulan |
0.61 | 0.72 | 0.73 | 0.76 | 0.64 | 0.66 | 0.80 | 0.80 | 0.76 | 0.71 | |
| 可溶性淀粉 Soluble starch |
0.77 | 0.95 | 1.03 | 1.05 | 0.99 | 0.88 | 1.05 | 0.99 | 0.97 | 0.94 | |
| 木薯淀粉 Cassava starch |
1.06 | 1.07 | 0.99 | 1.06 | 1.01 | 0.98 | 1.19 | 1.15 | 1.09 | 1.06 | |
| 支链淀粉 Amylopectin |
1.17 | 1.27 | 1.19 | 1.14 | 1.15 | 1.50 | 1.51 | 1.24 | 1.17 | 1.16 | |
3 讨论
突变酶E66G对大分子底物支链淀粉的水解能力有所提高,其可能原因是因为突变后氨基酸侧链空间位阻变小,但关键位点的空间构型几乎没有变化。由于第66位氨基酸由原来的谷氨酸(Glu,E)突变成甘氨酸(Gly,G),前者的侧链为—CH2—CH2—COOH,后者侧链为一个—H,远比Glu侧链小,因此突变后氨基酸侧链的空间位阻比突变前小,所以突变酶E66G对大分子底物支链淀粉的水解能力有所提高。但由于R基为—H,甘氨酸的性质介于极性和非极性之间,故由亲水的谷氨酸突变为甘氨酸,66位氨基酸的特性并没有太大的改变,所以cds1-3的构型几乎也没有变化。
酶蛋白活力的维持不仅与氨基酸的空间位阻、氨基酸的性质相关,还与氨基酸相互作用折叠形成的蛋白质空间结构有密切的关系。突变体F289A的突变位点靠近酶活性中心的底物结合区域,其没有酶活力的原因可能是氨基酸相互作用折叠形成的蛋白质空间结构改变,从而使酶活力丧失。对于突变体P48H,该突变体的突变位点位于远离底物结合区域的底物通道区域上,第48位氨基酸由最初的疏水Pro突变成亲水His,两个疏水性完全相反的氨基酸的替换,可能造成突变位点的氨基酸与附近氨基酸相互作用的范德华力的改变,使得蛋白质构型发生改变。
环糊精水解酶、麦芽糖淀粉酶和新普鲁兰酶这些具有环糊精降解能力的酶在氨基酸上具有40%~60%的序列同源性,在酶结构上同样具有相似性[10]。它们在N端有一段延伸序列是典型的α-淀粉酶不具有的。这段延伸序列被认为是参与酶的二聚体形成、影响酶底物的选择关键肽段[1, 11-12]。所以,cds1-3和ThMA在N端的氨基酸序列差异,造成两个酶底物特异性上差异的可能性更高。E66G突变酶在酶比活力上与野生酶一致,但是在底物特异性上发生了变化,其对小分子的普鲁兰糖水解能力降低,对大分子的木薯淀粉和支链淀粉水解能力增强。这一结果表明N端序列确实参与酶的底物选择。cds1-3和ThMA在N端的序列上共有9个氨基酸差异,对这9个氨基酸差异位点的突变研究将有助于对环糊精水解酶的蛋白质改造,提高支链淀粉的水解效率。同时有助于我们解析决定支链淀粉酶底物专一性的相关氨基酸,开拓新的支链淀粉水解酶及其应用。
4 结论本研究在氨基酸序列比对、蛋白质同源建模及蛋白质结构模拟分析的基础上,选定突变位点,成功构建得到3个突变体: E66G、P48H、F289A。通过定性测定破胞上清酶液发现,只有突变体E66G有酶活。对E66G进行诱导表达纯化,得到纯化的突变酶E66G。比较分析突变酶E66G与原始酶cds1-3的底物特异性,以及与地衣芽孢杆菌α-淀粉酶协同作用的协同率。结果表明,与原始酶cds1-3相比,突变酶E66G对大分子底物木薯淀粉和支链淀粉的降解能力有所提高。其中,对木薯淀粉降解的增长率为26.96%,对支链淀粉降解的增长率为23.15%。
E66G突变酶的鉴定为揭示大分子底物,特别是支链淀粉底物的水解作用方式提供了一个新的切入点。实验结果为进一步研究cds1-3中与底物专一性相关的氨基酸打下基础,有助于环糊精水解酶的蛋白质改造,从而提高支链淀粉的水解效率。
| [1] |
LEE H S, KIM M S, CHOS H S, et al. Cyclomaltodextrinase, neopullulanase, and maltogenic amylase are nearly indistinguishable from each other[J]. Journal of Biological Chemistry, 2002, 277(24): 21891-21897. DOI:10.1074/jbc.M201623200 |
| [2] |
LEE M H, KIM Y W, KIM T J, et al. A novel amylolytic enzyme from Thermotoga maritima, resembling cyclodextrinase and α-glucosidase, that liberates glucose from the reducing end of the substrates[J]. Biochemical and Biophysical Research Communication, 2002, 295(4): 818-825. DOI:10.1016/S0006-291X(02)00748-9 |
| [3] |
SONG H N, JUNG T Y, PARK J T, et al. Structural rationale for the short branched substrate specificity of the glycogen debranching enzyme GlgX[J]. Proteins, 2010, 78(8): 1847-1855. |
| [4] |
CHOI J H, LEE H, KIM Y W, et al. Characterization of a novel debranching enzyme from Nostoc punctiforme possessing a high specificity for long branched chains[J]. Biochemical and Biophysical Research Communications, 2009, 378(2): 224-229. DOI:10.1016/j.bbrc.2008.11.020 |
| [5] |
KAULPIBOON J, PONGSAWASDI P. Purification and characterization of cyclodextrinase from Paenibacillus sp.A11[J]. Enzyme and Microbial Technology, 2005, 36(2/3): 168-175. |
| [6] |
莫莉, 韦廷宗, 闭海, 等. 支链淀粉水解酶水解支链淀粉的特异氨基酸分析[J]. 广西科学, 2015, 22(1): 31-36. DOI:10.3969/j.issn.1005-9164.2015.01.005 |
| [7] |
OH S W, JANG M U, JEONG C K, et al. Modulation of hydrolysis and transglycosylation activity of Thermus maltogenic amylase by combinatorial saturation mutagenesis[J]. Journal of Microbiology and Biotechnology, 2008, 18(8): 1401-1407. |
| [8] |
FERNANDEZ-FUENTES N, MADRID-ALISTE C J, RAI B K, et al. M4T:A comparative protein structure modeling server[J]. Nucleic Acids Research, 2007, 35(web server): W363-W368. DOI:10.1093/nar/gkm341 |
| [9] |
ZHENG L, BAUMANN U, REYMOND J L. An efficient one-step site-directed and site-saturation mutagenesis protocol[J]. Nucleic Acids Research, 2004, 32(14): e115. DOI:10.1093/nar/gnh110 |
| [10] |
NAKAGAWA Y, SABURI W, TAKADA M, et al. Gene cloning and enzymatic characteristics of a novel γ-cyclodextrin-specific cyclodextrinase from alkalophilic Bacillus clarkii 7364[J]. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2008, 1784(12): 2004-2011. DOI:10.1016/j.bbapap.2008.08.022 |
| [11] |
HONDOH H, KURIKI T, MATSUURA Y. Three-dimensional structure and substrate binding of Bacillus stearothermophilus neopullulanase[J]. Journal of Molecular Biology, 2003, 326(1): 177-188. |
| [12] |
KIM J S, CHA S S, KIM H J, et al. Crystal structure of a maltogenic amylase provides insights into a catalytic versatility[J]. The Journal of Biological Chemistry, 1999, 274(37): 26279-26286. DOI:10.1074/jbc.274.37.26279 |



