2. 广西科学院广西红树林研究中心, 广西红树林保护与利用重点实验室, 广西北海 536000
2. Guangxi Key Lab of Mangrove Conservation and Utilization, Guangxi Mangrove Research Center, Guangxi Academy of Sciences, Beihai, Guangxi, 536000, China
红树林研究具有重要的理论意义和现实经济意义,近年来围填海是广西乃至我国红树林面积减少的最直接原因,由于大量的围垦造田、围海养殖和道路码头的修建,大量红树林受到破坏[1-2],严重威胁到海岸生物的多样性。对红树林生态系统的生物多样性进行持续性监测,是针对性制定和实施生态系统保护策略和合理有效利用资源的基本前提。红树林的种类组成和群落结构比陆地森林简单得多,但它有丰富的形态结构和地面微景观地貌,这些细化的生境对生物多样性的丰富至关重要[3]。目前,对红树林生物多样性的调查和研究主要采用传统的生态学调查方法,传统的调查方法费时费力且存在着诸多缺点,在分子生物学高速发展的今天,急需对传统的生物多样性调查方法作出改变。
环境DNA(Environmental DNA, eDNA)的概念于20世纪80年代末首次被提出,是指不对目标生物进行分离,而直接从环境样品(比如土壤、沉积物、空气、水体等)中提取的DNA[4]。环境DNA提取技术最早出现在环境微生物学领域,被用来分离和纯化沉积物中微生物的DNA。近年来,环境DNA技术被应用在生物多样性分析、生物量估测、珍稀物种发现、生物入侵物种的监控、隐存种的发现、群落系统发育重建、物种间食物链关系分析等生态学研究领域[5-8]。2008年至今,伴随着分子条形码数据库(DNA barcoding)的不断完善以及测序技术的飞速发展,环境DNA技术在水生生态系统的应用经历了由定性到相对定量,由检测单一物种到同时检测多物种,再到调查物种多样性的发展[9],水体应用范围已由一开始的淡水水域(湖泊、池塘、河流等)扩展至海洋,调查物种也由小型底栖动物扩展至鱼类、两栖类、哺乳动物等[10-13]。相比较而言,传统方法除采样过程艰辛外,其结果更大程度上取决于调查人员的专业素养和采样经验,对一些珍稀物种、隐存物种、动物幼体而言,随机性更大;而环境DNA技术经济效力高,省时省力,灵敏度高,对生态系统干扰低,采样受限小,同时环境DNA技术可以用非常标准化的方式在特定类型的栖息地中获取环境样本[14-15]。红树林地处大陆和海洋交界处,由于其生境的复杂性,使得红树林具有较高的生态价值和经济价值[16-17]。红树林具有特殊的生态系统,潮沟区复杂且生物多样性丰富,随着潮起潮落,红树林规律性地受到海水浸泡和露空,其生境具有较强的还原性、强酸性以及高盐含量等特性[18],因而红树林土壤的理化性质十分复杂,不能够稳定地提取土壤DNA,再加上红树林中物种的分子条形码信息相对缺乏,因此目前尚未有人通过环境DNA的方法开展红树林领域的生物多样性研究。利用环境DNA技术对红树林生态系统的生物多样进行调查研究,如何提取到高质量的环境DNA是这项研究最基础也是决定性的一步。
目前,针对红树林土壤DNA的研究主要集中于土壤微生物多样性,也有学者对提取土壤微生物DNA进行一些方法学研究,如液氮-SDS-溶菌酶法[19]、玻璃珠-SDS-溶菌酶法、SDS-GITC-PEG法[20]、Bio 101 kit法、Nycodenz法[21]、SDS-GITC-FastPrep-PEG法、细胞包埋法[22]以及基于Zhou等[23]方法的改良法[24],以提取土壤DNA。但尚未有人对红树林土壤环境DNA的提取做过全面的方法学研究。本研究通过预实验,发现前人推荐的方法更偏向于提取优质的微生物DNA,并不能有效地覆盖土壤环境DNA中所包含动植物DNA信息。本研究将比较多种方法提取和纯化红树林土壤环境DNA的效率,并讨论环境DNA中所包含的物种覆盖度(细菌、真菌、无脊椎动物、植物),为今后开展全方面的红树林生物多样性的研究提供方法学依据和基础。
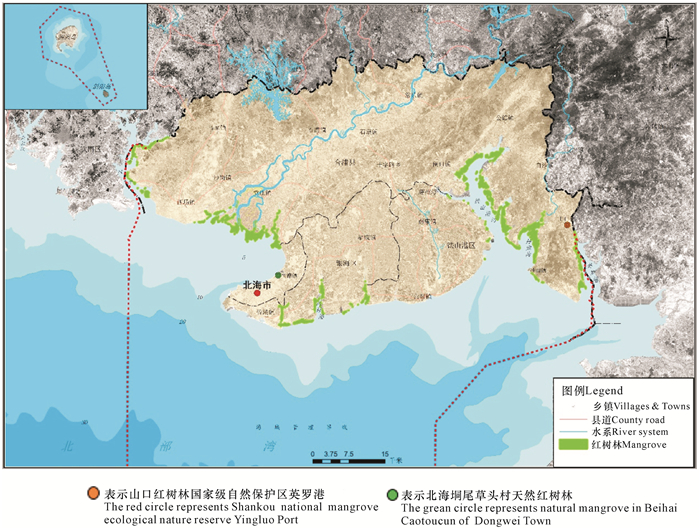
1 材料与方法 1.1 样品采集采样地为广西北海山口红树林国家级自然保护区英罗港(21°49′N,109°76′E)和北海垌尾草头村天然红树林(21°33′N,109°9′E),采样点见图 1,采集时间为2019年第二季度,在低潮区潮间带林间间隔10 m的树下,去表层浮土,收集10个点的土壤放入无菌袋中,将无菌袋放入有冰袋的隔温箱中,带回实验室放入-20℃的冰箱保存。
|
| 图 1 样品采集地 Fig. 1 Location of sample collection |
1.2 土壤粒径测定
使用Mastersizer 2000激光粒度仪(马尔文公司)对采回的土壤进行粒度测定,计算砾、砂、粉砂、黏土的比例。
1.3 土壤环境DNA提取 1.3.1 SDS-GITC-PEG法提取本法参照文献[20],并进行部分步骤的改良(为行文方便,下文及图表中该方法简称C),将采集的样品均匀混合,对土壤进行预处理[25]。取适量的土样于50 mL无菌离心管中,加5倍体积的TENP buffer(50 mmol/L Tris-Hcl,20 mmol/L EDTA,100 mmol/L NaCl,0.01 g/mL PVPP,pH值为10.0),涡旋振荡5 min,12 000×g室温离心5 min,收集沉淀(该步骤重复两次),加5倍体积的PBS buffer,涡旋混匀,12 000×g室温离心5 min,收集沉淀(该步骤重复两次)。沉淀冷冻干燥后装于无菌离心管中备用。称取10 g经预处理的土壤于50 mL离心管,加16 mL SDS裂解液(0.25 mol/L NaCl,0.1 mol/L EDTA,4% SDS),涡旋混匀,68℃温育30 min。加4 mL 5 mol/L异硫氰酸胍混匀后68℃温育1 h,每10 min轻轻摇动,12 000×g离心10 min,将上清液转移入50 mL干净的离心管,加0.125倍体积的5 mol/L醋酸钾和0.42倍体积40% PEG8000,-20℃下沉淀2-4 h,14 000×g离心20 min。沉淀用18 mL 2×CTAB (2% CTAB,1.4 mol/L NaCl,0.1 mol/L EDTA)溶解,68℃温育15 min,加等体积的氯仿:异戊醇,轻柔混匀,12 000×g室温离心10 min,加0.1倍体积3 mol/L NaAc和0.6倍体积的异丙醇,静置于室温下1 h,14 000×g,25℃离心20 min,沉淀物用70%乙醇清洗两次,加适量TE溶解沉淀,并收集于1.5 mL的微型离心管中。
1.3.2 试剂盒法提取采用土壤基因组DNA快速提取试剂盒(Bioteke公司)(以下简称B+),TIANamp Soil DNA试剂盒(天根公司)(以下简称T),FastDNA Spin Kit for Soil试剂盒(MP公司)(以下简称F)和DNeasy PowerSoil试剂盒(Qiangen公司)(以下简称P),提取方法参照各试剂盒操作说明书。其中Bioteke试剂盒实验中省去溶菌酶处理的一步,其他步骤不变,也进行了提取DNA的尝试(以下简称B-)。
因此本研究中一共用6种方法提取红树林土壤环境DNA。测定提取后的DNA浓度、A260/A280和A260/A230,并用琼脂糖凝胶电泳检验DNA质量。所有实验均设置3次以上的实验重复。
1.4 DNA纯化用2种试剂盒对上述提取的DNA进行纯化:柱式腐殖酸清除试剂盒法(北京天恩泽)(以下简称H)和One-step PCR Inhibitor Removal试剂盒(ZYMO Research公司)(以下简称O),实验方法参照试剂盒说明书。测定纯化后的DNA浓度,并用琼脂糖凝胶电泳检测DNA质量,同时以未用纯化试剂盒纯化的样品(以下简称W)作对照。
1.5 PCR扩增使用不同的细菌、真菌、底栖无脊椎动物、植物和线虫的通用引物对提取的土壤环境DNA进行PCR扩增,筛选到以下扩增效果较好的引物,引物序列及对应的PCR程序见表 1。
| 物种名称 Name of species |
引物 Primer |
引物序列 Primer sequence |
PCR程序 PCR procedure |
| 细菌Bacteria | F341GC | 5′-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGG- CACGGGGGGCCTACGGGAGGCAGCAG-3′ | 94℃ 30 s, 57℃ 30 s, 72℃ 30 s, 35 cycles |
| R534 | 5′-ATTACCGCGGCTGCTGG-3′ | ||
| 真菌 Fungus |
ITS1F | 5′-CTTGGTCATTTAGAGGAAGTAA-3′ | 94℃ 30 s, 53℃ 30 s, 72℃ 30 s, 37 cycles |
| ITS2R | 5′-GCTGCGTTCTTCATCGATGC-3′ | ||
| 植物 Plant |
matK3F-KIM | 5′-CGTACAGTACTTTTGTGTTTACGAC-3′ | 94℃ 30 s, 55℃ 30 s, 72℃ 1 min, 38 cycles |
| matKIR-KIM | 5′-ACCCAGTCCATATGGAAATCTTGGTTC-3′ | ||
| 无脊椎动物 Invertebrate |
LCO1490 | 5′-GGTCAACAAATCATAAAGATATTGG-3′ | 94℃ 30 s, 50℃ 30 s, 72℃ 1 min, 35 cycles |
| HCO2198 | 5′-TAAACTTCAGGGTGACCAAAAAATCA-3′ | ||
| 线虫 Nematode |
Nem-18SF | 5′-CGCGAATRGCTCATTACAACAGC-3′ | 94℃ 30 s, 57℃ 30 s, 72℃ 1 min, 37 cycles |
| Nem-18SR | 5′-GGGCGGTATCTGATCGCC-3′ |
1.6 数据分析
使用Graphpad Prism 7软件分别对两个地方样品不同提取方法得到的DNA浓度两两进行单因素t检验,分析不同提取方法之间是否存在差异性,以及绘制A260/A280折线图,使用Origin 6软件绘制所提DNA浓度的柱状图,使用Excel对纯化后的DNA得率进行计算。
2 结果与分析 2.1 土壤粒径分析使用激光粒度仪对草头村和山口采集回来的样品进行粒径分析,山口样品砂粒的含量是草头村的2.46倍,而草头村的土样中粉砂和黏土的含量比山口土样中的高(表 2)。综合来说,草头村的土壤更黏,而山口红树林取样点的土壤沙质更多。
| 采样地点 Sampling site |
粒组含量Grain group content (%) | |||
| 砾Gravel | 砂Grit | 粉砂Grit | 黏土Clay | |
| 山口Shankou | 0.00 | 37.08 | 44.28 | 18.04 |
| 草头村Caotoucun | 0.00 | 15.04 | 6.93 | 23.03 |
2.2 土壤环境DNA提取和纯化
用6种方法提取两处不同红树林的土壤环境DNA,其中5种试剂盒法每次实验均提取0.3 g土壤,SDS-GITC-PEG法取10 g土壤(为了数据对比的合理性和统一性,此法提取到的DNA量也换算成以0.3 g土壤为起始量),将得到的DNA浓度分别进行两两t检验(表 3)。对于草头村土壤样品,各方法提取的土壤环境DNA浓度大小为B+>B->F>P>T>C,即Bioteke试剂盒的提取效率最高。统计结果表明,对于草头村样品,B+和B-提取的DNA浓度P>0.1,差异不显著,由此看出Bioteke试剂盒省去用溶菌酶消化这一步骤对总DNA提取影响不大;但其他试剂盒两两对比,得到的P值均小于0.05,说明其他几种试剂盒DNA提取效率存在显著性差异。对于山口红树林土样,用不同方法提取后的DNA浓度对比情况和草头村的有所差异,土壤环境DNA浓度大小为B-≥B+>F≈T≈C≥P。将所得浓度值同样也进行两两t检验,P、F、T和C 4种方法同B+对比得到的P < 0.05,说明B+与这4种方法存在明显差异,虽然B-方法所提取的DNA浓度平均值最高,但标准差也最大,t检验显示B-法与其余5种方法的差异不显著,此外,也可以看到P和F之间存在明显差异。不同试剂盒对于不同采样地提取DNA的结果有差异,推测与采样地样品的理化性质有关。总的来说,就总DNA的提取效率而言,Bioteke土壤基因组DNA快速提取试剂盒表现最优,FastDNA Spin Kit for Soil试剂盒次之,诸多文献中推荐的DNeasy PowerSoil试剂盒在提取山口红树林土壤DNA时表现欠佳。
| 采样地点 Sampling site |
提取方法 Extraction methods |
浓度 Concentration (μg/μL) |
P(vs B-) | P(vs P) | P(vs F) | P(vs T) | P(vs C) |
| 草头村Caotoucun | B+ | 91.14±10.09 | 0.239 0 | < 0.000 1 | 0.000 2 | < 0.000 1 | < 0.000 1 |
| B- | 70.68±13.34 | 0.006 5 | 0.040 1 | 0.000 1 | 0.000 1 | ||
| P | 28.800±1.167 | 0.000 1 | < 0.000 1 | < 0.000 1 | |||
| F | 40.540±1.975 | < 0.000 1 | < 0.000 1 | ||||
| T | 17.130±1.029 | < 0.000 1 | |||||
| C | 2.178 0±0.934 6 | ||||||
| 山口Shankou | B+ | 88.9±25.67 | 0.857 5 | 0.008 7 | 0.016 1 | 0.016 0 | 0.017 1 |
| B- | 99.29±50.76 | 0.105 4 | 0.137 0 | 0.130 6 | 0.130 1 | ||
| P | 12.13±1.35 | < 0.000 1 | 0.383 1 | 0.548 3 | |||
| F | 19.820 0±0.402 6 | 0.747 4 | 0.756 7 | ||||
| T | 17.800±6.169 | 0.956 9 | |||||
| C | 17.230±8.204 |
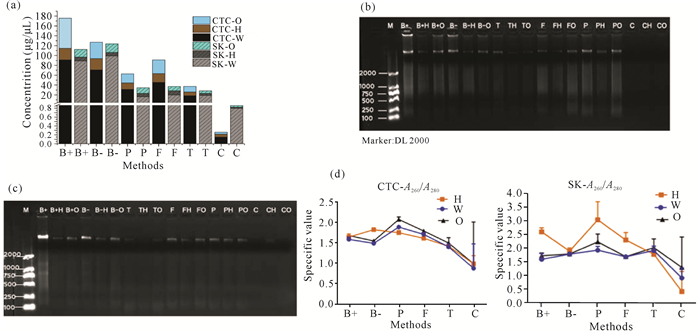
本研究使用2种试剂盒对6种方法提取的总DNA进行纯化。总体来说,One-step PCR Inhibitor Removal试剂盒纯化效率比腐殖酸清除剂试剂盒纯化效率要高,各种方法提取的草头村土壤环境DNA经两种试剂盒纯化后的得率总体要比山口红树林土壤的高(表 4)。但使用这两种试剂盒对DNA纯化后都对土壤环境DNA造成不小的损失(图 2a),损失率在37.40%-96.25%。特别是之前表现最佳的Bioteke试剂盒提取的DNA经腐殖酸清除剂试剂盒纯化后造成DNA大量损失,损失率高达65.80%-93.45%,其中,山口红树林土壤由Bioteke试剂盒提取的DNA经腐殖酸清除剂试剂盒纯化后损失率尤其大,得率仅有7.33%,经One-step PCR Inhibitor Removal试剂盒纯化后的得率也仅为14.59%(表 4)。6种方法提取以及2种方式纯化后的DNA均经过琼脂糖凝胶电泳的检验,可以看到总DNA清晰明亮的条带(图 2b-c)。
| 采样地点 Sampling site | 提取方法 Extraction methods |
浓度平均值 Mean concentraction (μg/μL) |
试剂盒H处理浓度 Treatment of Kit H (μg/μL) |
得率 Yield (%) |
试剂盒O处理浓度 Treatment of Kit O (μg/μL) |
得率 Yield (%) |
| 草头村 Caotoucun |
B+ | 84.200 | 23.420 | 27.80 | 61.140 | 72.60 |
| B- | 66.900 | 22.880 | 34.20 | 33.360 | 49.90 | |
| P | 31.080 | 13.390 | 41.80 | 18.120 | 58.30 | |
| F | 45.480 | 17.690 | 38.90 | 27.970 | 61.50 | |
| T | 18.360 | 7.610 | 41.40 | 11.580 | 63.10 | |
| C | 0.147 | 0.068 | 46.30 | 0.045 | 30.60 | |
| 山口 Shankou |
B+ | 107.680 | 7.890 | 7.33 | 15.710 | 14.59 |
| B- | 106.520 | 6.980 | 6.55 | 17.390 | 16.33 | |
| P | 16.430 | 6.620 | 40.29 | 11.540 | 70.24 | |
| F | 19.790 | 8.760 | 44.26 | 8.760 | 44.26 | |
| T | 18.880 | 4.180 | 22.14 | 5.400 | 28.60 | |
| C | 0.800 | 0.030 | 3.75 | 0.040 | 5.00 |
|
| CTC:草头村;SK:山口;(a)DNA浓度的综合比较;(b)草头村土壤环境DNA电泳图;(c)山口红树林土壤环境DNA电泳图;(d)DNA的A260/A280示意图 CTC:Caotoucun, SK:Shankou; (a)Comprehensive comparison of eDNA concentration, (b)Electrophoresis of soil eDNA in Caotoucun, (c)Electrophoresis of total DNA of mangrove soil in Shankou, (d)A260/A280 schematic diagram of eDNA 图 2 土壤环境DNA提取及纯化结果 Fig. 2 Extraction and purification results of soil eDNA |
DNA在260 nm和280 nm下的吸光光度比值体现了DNA的纯度,纯DNA的A260/A280的比值为1.7-1.8较好[22]。两个地点的样品采用6种提取方法,经2个纯化试剂盒纯化后,A260/A280的变化趋势和纯化前基本一致;草头村样品提取的DNA经腐殖酸清除剂试剂盒纯化后A260/A280基本在1.8附近,而One-step PCR Inhibitor Removal试剂盒纯化后的DNA的A260/A280波动较大;与之相反的是,山口红树林的土壤样品提取的DNA经腐殖酸清除剂试剂盒纯化后A260/A280波动很大,而One-step PCR Inhibitor Removal试剂盒处理的DNA的A260/A280相对稳定,维持在1.6-2.0(图 2d)。推测由于两地土壤粒径差异较大,造成土壤中的微生物丰富程度有差异,腐殖酸及生物酶含量也有差异,导致两种试剂盒的表现不一致。
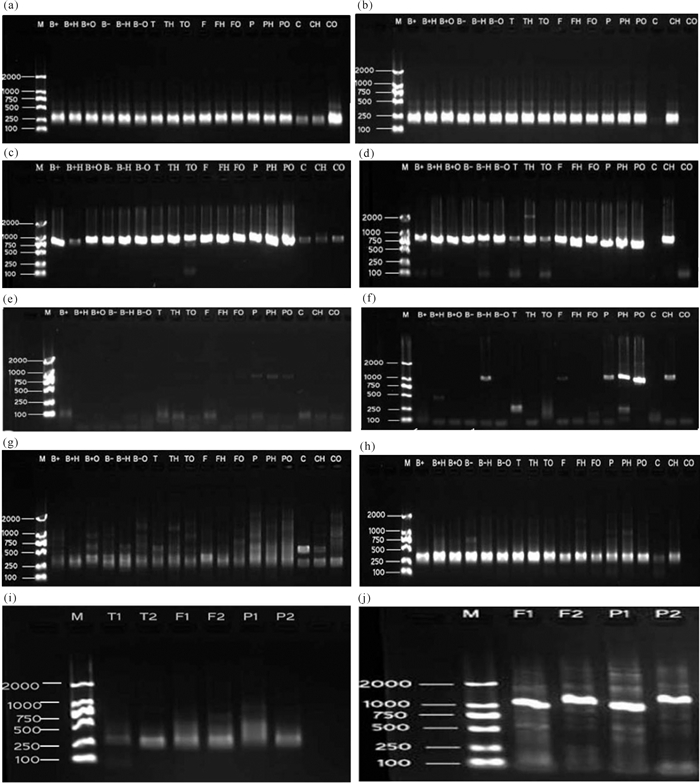
2.3 PCR扩增结果图 3a-b为两个地点提取的DNA对细菌16S的引物PCR扩增结果,4种试剂盒(1-3,7-15泳道)直接提取的DNA和经过两种纯化试剂盒处理的DNA针对细菌16S引物都能够有效扩增,Bioteke土壤基因组DNA快速提取试剂盒省去用溶菌酶消化这一步骤(4-6泳道)也能有效地扩增细菌DNA。草头村样品使用SDS-GITC-PEG法提取的DNA能扩增出单一的条带,但未经纯化和经腐殖酸清除剂试剂盒纯化的DNA扩增出的目的条带亮度较暗,经One-step PCR Inhibitor Removal试剂盒纯化后的DNA扩增出的条带清晰明亮;山口样品使用SDS-GITC-PEG法提取的DNA无法扩增出目的条带,而经过腐殖酸清除剂试剂盒纯化处理后的DNA能够有效扩增,但经One-step PCR Inhibitor Removal试剂盒处理后的DNA,未能扩增出目的条带。两种纯化试剂盒再次有不同的表现。
|
| (a)草头村-细菌,(b)山口-细菌,(c)草头村-无脊椎动物,(d)山口-无脊椎动物,(e)草头村-植物,(f)山口-植物,(g)草头村-真菌,(h)山口-真菌,(i)草头村、山口-真菌(高速振荡),(j)草头村、山口-线虫; 1表示草头村,2表示山口 (a)Caotoucun-bacteria, (b)Shankou-bacteria, (c)Caotoucun-invertebrate, (d)Shankou-invertebrate, (e)Caotoucun-plant, (f)Shankou-plant, (g)Caotoucun-fungus, (h)Shankou-fungus, (i)Caotoucun, Shankou-fungus(the high speed shock), (j)Caotoucun, Shankou-nematode; 1 is Caotoucun, 2 is Shankou 图 3 细菌、植物、无脊椎动物、真菌、线虫PCR扩增结果 Fig. 3 PCR amplification result of bacteria, plants, invertebrates, fungus, and nematodes |
图 3c-d为无脊椎动物线粒体COI基因扩增结果,5种试剂盒方法直接提取的DNA和经两种纯化试剂盒处理的DNA都能扩增出有效条带;SDS-GITC-PEG法提取的草头村样品DNA也能扩增出单一条带,但条带的亮度较暗,而山口红树林样品DNA,经腐殖酸清除剂试剂盒纯化后可以扩增出单一且明亮的条带。
图 3e-f为植物叶绿体基因matK的扩增结果,草头村样品中,只有用DNeasy PowerSoil试剂盒提取的DNA扩增出单一条带,但条带亮度比较暗,而山口红树林样品提取的DNA,在进行二次PCR扩增时,Bioteke试剂盒去溶菌酶一步提取后再经去腐殖酸试剂盒纯化、FastDNA Spin Kit for Soil试剂盒直接提取、DNeasy PowerSoil试剂盒直接提取和2种纯化方式,以及SDS-GITC-PEG法提取后去腐殖酸纯化均出现有效条带,且用DNeasy PowerSoil试剂盒提取的DNA经两种方式纯化后扩增出来的目的条带最优。
草头村和山口的真菌PCR扩增图基本都出现了符合目的条带区间的条带,但两个地点的DNA扩增出的主要条带亮度明显有差异(图 3g-h),表明两个不同红树林区域的土壤可能因为土壤粒径、植被种类不同等原因,造成真菌种类的差异。有文献指出,由于真菌特殊的细胞壁结构以及不同的土壤性质的复杂性,提取土壤的真菌DNA需要对常规的提取方法进行高速振荡的优化,本研究按照文献[26]的方法,加了高速振荡处理后,用3个试剂盒进行DNA的提取并如前述方法进行PCR扩增,从PCR电泳图中(图 3i)看出,条带并没有明显的变化,甚至山口样品DNA的扩增条带比之前亮度要低,推测高速振荡可能导致真菌破壁过度而影响了DNA的提取。
因为小型底栖动物如线虫类,不是我们今后的研究范畴,在此只选择了两种方法提取的DNA对线虫18S rDNA进行了扩增。使用FastDNA Spin Kit for Soil和DNeasy PowerSoil试剂盒直接提取的DNA对线虫进行PCR扩增,来自两个红树林采样点的土壤DNA都能扩增出有效的单一条带(图 3j)。
3 讨论本研究对北海两处红树林潮间带采集土壤样品进行土壤环境DNA提取的方法学研究。采用6种方法提取土壤环境DNA,实验结果显示,对于草头村的土壤样品,Bioteke土壤基因组DNA快速提取试剂盒的提取效率显著高于其他4种方法;对于山口红树林采集的土壤样品,也是Bioteke试剂盒提取效率最高,与其他4种方法存在显著性差异。FastDNA Spin Kit for Soil试剂盒和DNeasy PowerSoil试剂盒这2种在外文文献中最常用到的试剂盒提取的DNA浓度差异显著,且浓度均较低,远远低于Bioteke试剂盒提取的。FastDNA Spin Kit for Soil试剂盒和DNeasy PowerSoil试剂盒在国外多篇论文中均有推荐和使用[27-28],但也有国内学者得出和本研究相似的结果,即这两个进口试剂盒提取土壤环境DNA量较少[29]且价格昂贵。
红树林的强还原性、强酸性和高盐含量等特性使得在该生境下的物种也必有其特殊性,同时,红树林土壤中有机物通过植物碎屑和腐烂的根系不断积累,腐殖酸和各种生物酶丰富[3]。腐殖酸等因子会影响PCR的扩增效率[30],因此在做PCR扩增实验之前,通常需要对提取的红树林土壤环境DNA进行纯化处理,以去除腐殖酸等PCR抑制因子。两种纯化试剂盒纯化DNA后,虽然A260/A280有一定的改善,但DNA的得率均不高,DNA的损失高达37.40%-96.25%,尤其是在提取DNA时,表现优秀的Bioteke试剂盒提取的DNA损失率最高。
土壤环境DNA包括了胞内和胞外的DNA,其中胞内DNA通常为原核、真核微生物的DNA以及动植物脱落的细胞,而胞外DNA是环境DNA中非常重要的一部分,这些游离的DNA来自于生物体脱落的皮肤、排泄尿液、粪便、体液等[31],正是这些来源的胞外DNA使得通过环境DNA来检测生物多样性成为可能。为检验提取的DNA是否包含有丰富的物种信息,首先使用广西北部湾红树林中的广布物种红树蚬(Polymesoda erosa)和歪红树蚬(Polymesoda expansa)的线粒体COI特异引物和红树植物秋茄(Kandelia candel)的叶绿体matK特异引物进行PCR扩增,得到相应的特异性扩增产物条带,并测序验证成功(结果未展示)。其次,为检验所提取的DNA是否具有广泛的物种覆盖度,使用细菌、真菌、底栖无脊椎动物、植物和线虫的通用引物分别进行扩增,目的扩增序列分别为细菌16S rDNA的V3区[32],真菌ITS1[33],无脊椎动物的线粒体COI[34],植物叶绿体matK[35],线虫18S rDNA[36](表 1)。
目前市售的土壤环境DNA提取试剂盒主要针对土壤微生物多样性研究,因此相应地对提取微生物DNA的效果进行了优化,本研究实验结果也可以看出,4个试剂盒都能有效提取土壤中的细菌DNA,PCR结果良好。在前人发表的论文中提到红树林土壤中含有腐殖酸等PCR抑制剂,需要去除后再进行PCR扩增,但本研究结果表明,这4种试剂盒无论是根据试剂盒说明书直接提取,还是用国产腐殖酸去除试剂盒以及One-step纯化试剂盒处理后得到的DNA,均能实现对细菌引物和无脊椎动物引物的有效扩增,说明试剂盒本身已经对PCR抑制剂实现了去除。稍意外的是,将土壤基因组DNA快速提取试剂盒中溶菌酶消化这一步给省去后,得到的实验结果从电泳结果上看,与不省去步骤所得结果相比也并没有差别。对于植物而言,需要两次扩增后,土壤基因组DNA快速提取试剂盒、FastDNA Spin Kit for soil试剂盒、DNeasy PowerSoil试剂盒提取的DNA的扩增产物才出现单一的条带,但即使两次扩增,土壤基因组DNA快速提取试剂盒和FastDNA Spin Kit for soil试剂盒提取的DNA扩增的条带也较暗,DNeasy PowerSoil试剂盒提取的DNA扩增效果最佳,可能是因为红树林中的植物落叶被微生物和动物快速分解和食用,导致土壤中含有植物的DNA量很少,所以较难扩增。本研究使用秋茄特异性引物对土壤环境DNA进行扩增时,也得到一样的结果,说明DNeasy PowerSoil试剂盒虽然在提取DNA的量上并不突出,但所提取的DNA覆盖面更加全面。由于土壤理化性质的复杂性和真菌细胞壁结构的特殊性,草头村和山口土样所提取的DNA在真菌扩增中存在明显的差异,推测与土壤径粒差异相关。有文献表明,进行高速振荡可以有效地提高真菌细胞的破壁率,所以本研究也尝试在原有基础上将样品进行高速振荡,但对结果并没有实质性的改变。底栖物种除微生物、植物、大型底栖动物外,还包括以线虫为主的小型底栖动物,而线虫由于体型小,传统分类学的研究人员较少,多样性的研究难度较大,所以本研究也尝试采用文献报道的线虫的通用引物来进行扩增,发现也可以有效地扩增出DNA条带。所提DNA的PCR扩增实验结果在一定程度上反映提取到的总DNA中细菌、真菌、无脊椎动物、线虫和植物的覆盖率,但还不能量化说明所提的各类物种的覆盖度和丰度,在后续实验中,建议通过高通量测序对不同处理方法得到的DNA中各类物种的覆盖度和丰度并进行进一步的研究。
4 结论本研究比较了不同的提取方法、纯化方法在红树林土壤中提取环境DNA的效率,并使用细菌、真菌、底栖无脊椎动物、植物、线虫的不同通用引物进行PCR扩增来讨论提取的环境DNA中物种的覆盖度。结果显示,不同的方法提取不同类群生物产生的环境DNA的效率有一定的差异;但两种方法纯化前后DNA的质量并没有显著差异;5种试剂盒方法提取的DNA均可以有效扩增出细菌和无脊椎动物的DNA;DNeasy PowerSoil试剂盒提取的DNA扩增植物引物结果最佳;来自不同区域红树林土壤的DNA在扩增真菌对应引物时差异较大;使用FastDNA Spin Kit for Soil和DNeasy PowerSoil试剂盒直接提取的DNA可以有效扩增出线虫的DNA。本研究为在红树林研究工作中进一步开展基于环境DNA技术的生物多样性研究奠定了一定的基础。今后的研究工作将对PCR扩增得到的产物进行高通量测序,并通过生物信息学来分析物种的种类及丰度,最终建立一套基于环境DNA技术的红树林生物多样性调查方法,对传统的生物多样性监测提供一个有力的补充,为红树林生物多样性的监测、保护和修复提供科学依据。
| [1] |
范航清, 陆露, 阎冰. 广西红树林演化史与研究历程[J]. 广西科学, 2018, 25(4): 343-351. |
| [2] |
林鹏. 中国红树林研究进展[J]. 厦门大学学报:自然科学版, 2001, 40(2): 592-603. |
| [3] |
何斌源, 范航清, 王瑁, 等. 中国红树林湿地物种多样性及其形成[J]. 生态学报, 2007, 27(11): 4859-4870. |
| [4] |
OGRAM A, SAYLER G S, BARKAY T. The extraction and purification of microbial DNA from sediments[J]. Journal of Microbiological Methods, 1987, 7(2/3): 57-66. |
| [5] |
PIAGGIO A J, ENGEMANR R, HOPKEN M W, et al. Detecting an elusive invasive species:A diagnostic PCR to detect Burmese python in Florida waters and an assessment of persistence of environmental DNA[J]. Molecular Ecology Resources, 2014, 14: 374-380. |
| [6] |
DEINER K, WALSER J C, MACHLER E, et al. Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA[J]. Biological Conservation, 2015, 183: 53-63. |
| [7] |
BALASINGHAM K D, WALTER R P, MANDARK N E, et al. Environmental DNA detection of rare and invasive fish species in two Great Lakes tributaries[J]. Molecular Ecology, 2018, 27(1): 112-127. |
| [8] |
SHELTON A O, O'DONNELL J L, SAMHOURI J F, et al. A framework for inferring biological communities from environmental DNA[J]. Ecology Applications, 2016, 26(6): 1645-1659. |
| [9] |
DRUMMOND A J, NEWCOMB R D, BUCKLEY T R, et al. Evaluating a multigene environmental DNA approach for biodiversity assessment[J]. Giga Science, 2015, 4: 46. |
| [10] |
PILLOD D S, GOLDBERG C, ARKLER R S, et al. Estimating occupancy and abundance of stream amphibians using environmental DNA from filtered water samples[J]. Canadian Journal of Fisheries and Aquatic Sciences, 2013, 70: 1123-1130. |
| [11] |
高天翔, 陈治, 王晓艳. 近海鱼类多样性调查新方法-环境DNA分析技术[J]. 浙江海洋大学学报:自然科学版, 2018, 37(1): 1-7. |
| [12] |
BOHMANN K, EVANS A, GILBERT M T P, et al. Environmental DNA for wildlife biology and biodiversity monitoring[J]. Trends in Ecology & Evolution, 2014, 29(6): 358-367. |
| [13] |
SANSOM B J, SASSOUBRE L M. Environmental DNA (eDNA) shedding and decay rates to model freshwater mussel eDNA transport in river[J]. Environmental Science & Technology, 2017, 51(24): 14244-14253. |
| [14] |
DEINER K, WALSER J C, MÄCHLER E, et al. Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA[J]. Biological Conservation, 2015, 183: 53-63. |
| [15] |
TAKAHARA T, MINAMOTO T, DOI H. Effects of sample processing on the detection rate of environmental DNA from the common carp (Cyprinus carpio)[J]. Biological Conservation, 2015, 183: 64-69. |
| [16] |
范航清, 王文卿. 中国红树林保育的若干重要问题[J]. 厦门大学学报:自然科学版, 2017, 56(3): 323-330. |
| [17] |
甘加俊. 红树林湿地生态系统价值及保护探讨[J]. 绿色科技, 2019(12): 46-47. |
| [18] |
林初夏, 吴志峰. 酸性硫酸盐土的酸度类型及其测定方法[J]. 生态环境, 2003, 12(4): 505-507. |
| [19] |
DOJKA M A, HARRIS J K, PACE N P. Expanding the known diversity and environmental distribution of an uncultured phylogentic division of bacteria[J]. Environ Microbiology, 2000, 66: 1617-1621. |
| [20] |
PORTEOUS L A, ARMSTRONG J L, SEIDLER R J, et al. An effective method to extract DNA from environmental samples for polymerase chain reaction amplification and DNA fingerprint analysis[J]. Current Microbiology, 1994, 29(5): 301-301. |
| [21] |
BERTRAND H, POLY F, VAN V T, et al. High molecular weight DNA recovery from soil prerequisite for biotechnological metagenomic library construction[J]. Journal of Microbiological Methods, 2005, 62(1): 1-11. |
| [22] |
杨建, 洪葵. 红树林土壤总DNA不同提取方法比较研究[J]. 生物技术通报, 2006(S1): 366-371. |
| [23] |
ZHOU J Z, BRUNS M A, TIEDJE J M. DNA recovery from soil of diverse composition[J]. Applied and Environmental Microbiology, 1996, 62(2): 316-322. |
| [24] |
熊明华, 曹焰晖, 杨芮, 等. 一种改良的可直接用于PCR扩增的土壤DNA提取方法[J]. 基因组学与应用生物学, 2019, 38(4): 1643-1648. |
| [25] |
BERRY A E, CHIOCCHINI C, SELBY T, et al. Isolation of high molecular weight DNA from soil for cloning into BAC vectors[J]. FEMS Microbiology Letters, 2003, 223(1): 15-20. |
| [26] |
袁小凤, 路京, 宋皓军, 等. 浙贝母土壤真菌DNA提取方法的优化[J]. 浙江中医药大学学报, 2013, 37(1): 69-73. |
| [27] |
PENARRUBIA L, ALCARAZ C, VAATE A B D, et al. Validated methodology for quantifying infestation levels of dreissenid mussels in environmental DNA (eDNA) samples[J]. Scientific Reports, 2016, 6: 39067. |
| [28] |
MARÉCHAL C L, FOUROUR S, BALLAN V, et al. Detecting of Clostridium botulinum group Ⅲ in environmental samples from farms by real-time PCR using four commercial DNA extraction kits[J]. BMC Research Notes, 2018, 11: 441. |
| [29] |
梁文贤, 黄墩福, 崔朋辉, 等. 森林土壤微生物总DNA的提取方法[J]. 福建林学院学报, 2014, 34(2): 126-130. |
| [30] |
李超, 梁俊峰. 土壤微生物总DNA提取及其PCR优化[J]. 桉树科技, 2015, 32(2): 17-21. |
| [31] |
ANDERSON-CARPENTER L L, MCLACHLAN J S, JACKSON S T, et al. Ancient DNA from lake sediments:Bridging the gap between paleoecology and genetics[J]. BMC Evolutionary Biology, 2011, 11: 30. |
| [32] |
ACOSTA-MARTÍNEZ V, DOWD S, SUN Y, et al. Taq-encoded pyrosequencing analysis of bacterial diversity in a single soil type as affected by management and land use[J]. Soil Biology and Biochemistry, 2008, 40(11): 2762-2770. |
| [33] |
BUÉE M, REICH M, MURAT C, et al. 454 Pyrosequ-encing analysis of forest soils reveal an unexpectedly high fugal diversity[J]. New Phytologist, 2009, 184(2): 449-456. |
| [34] |
FOLMER O, BLACK M, HOECH W, et al. DNA primers for amplification of mitochondrial Cytochrome C oxidase subunit Ⅰ from diverse metazoma invertabarates[J]. Molecular Marine Biology and Biotechnology, 1994, 3(5): 294-299. |
| [35] |
COSTION C, FORD A, CROSS H, et al. Plant DNA barcodes can accurately estimate species richness in poorly know floras[J]. PLoS One, 2011, 6(11): e26841. |
| [36] |
CHAUDHARY A, KANSAL G, SINGH N, et al. New molecular data for parasites Hammerrs chmidtiella indicus and Thelandros sclertus (Nematoda:Oxyurida) to infer phylogenetic position[J]. Turkish Journal of Zoology, 2015, 39: 251-255. |



