2. 融水苗族自治县国营贝江河林场, 广西柳州 545300;
3. 融安县西山林场, 广西柳州 545400
2. State-owned Beijianghe Forest Farm, Rongshui Miao Autonomous County, Liuzhou, Guangxi, 545300, China;
3. Rong'an State-owned Xishan Forest Farm, Liuzhou, Guangxi, 545400, China
种质资源是动植物遗传改良的物质基础,在品种选育以及优异材料创制和利用研究中起重要作用。由于盲目开发及生态环境问题,导致物种多样性锐减,种质资源受到世界各国的关注,并相继创建了种质资源库[1],保存收集各类种质资源。随着收集数量增多,保存管护、评价利用方面出现各种困难,且种质资源未能充分发挥其重要作用,故核心种质的概念及其构建的方法得以提出和发展[2, 3], 其思想是通过合理的方法,从原始种质中筛选代表性的材料构成核心种质,其数量规模远小于原始种质,意在提高种质资源利用效率,发掘优异基因。
我国核心种质研究工作起步于1994年,最先在部分农作物上建立核心种质[4-8],在林木中的有关研究工作相对滞后。目前白桦Betula platyphylla[9]、柿Diospyros kaki[10]、灰楸Catalpa fargesii[11]、欧洲黑杨Populus nigra[12]、尾叶桉Eucalyptus urophylla[13]、日本柳杉Cryptomeria japonica[14]、银杏Ginkgo biloba[15]、木荷Schima superba[16]和水青树Tetracentron sinense[17]等树种构建了核心种质。多年生林木树体高大,占地面积广,需耗费大量经费管护,建立核心种质可更高效地保护和利用林木种质资源。
杉木Cunninghamia lanceolata是我国南方重要的针叶用材树种之一,具速生、材质优等特点,在国家木材储备战略中具有重要地位[18]。广西从20世纪70年代开始进行杉木遗传改良工作,持续收集了不同产区的杉木种质材料,为杉木遗传改良提供了丰富的种质资源[19, 20]。随着杉木种质资源收集数量增多,保存与评价利用的难度加剧,因此构建核心种质对充分利用现有杉木种质资源,挖掘优异基因显得尤为重要。本研究通过SSR分子标记技术检测国家杉木良种基地基因库的864份种质材料的遗传多样性,对比分析不同构建方法的差异,从而确定合适的构建方法和取样比例,为广西杉木种质研究和挖掘优异基因提供理论支撑与材料基础。
1 材料与方法 1.1 材料试验材料来自广西融安县西山林场(25°20′ N,109°23′ E)和融水苗族自治县国营贝江河林场(24°57′ N,109°11′ E)国家级杉木良种基地基因库。杉木种质取样总数为864份,其地理来源分别为广西(605份)、湖南(70份)、广东(55份)、福建(54份)、江西(40份)、贵州(31份)和湖北(9份)。
2019年11月,采集健康且无病害的杉木新鲜嫩叶,放入1.5 mL离心管后,投入液氮罐暂存,带回实验室后于-80℃超低温冰箱保存备用。
1.2 主要仪器及试剂22对SSR引物[21]由北京睿博兴科公司合成,植物DNA提取试剂盒购自生工生物工程(上海)股份有限公司,NanoDrop-2000型超微量分光光度计购自美国热电公司,ABI 3730xl型基因测序仪购自美国ABI公司,Biometra Tone 96G PCR仪购自德国EasyCycler公司。主要试剂有氯仿、β巯基乙醇、异丙醇、50×TAE等。
1.3 基因组DNA提取及PCR扩增——SSR标记法杉木种质样本基因组DNA提取按照植物DNA提取试剂盒提供的方法进行。(1)用1.0%琼脂糖凝胶电泳(150 V、100 mA,15-20 min)检测DNA纯度和完整度。用紫外观测仪观察电泳结果,若DNA分离较好,亮度较好,没有弥散,说明提取的DNA纯度较高且比较完整;若DNA没有分离,亮度较差且存在弥散,则需要重新提取DNA。(2)用NanoDrop-2000超微量分光光度计测定提取的DNA样品浓度。合格标准A260/A280值为1.70-1.90,若不处于此范围则重新提取DNA。提取结果合格的各样品DNA模板(共864份),用TE Buffer统一稀释至15 ng/μL后,于-20 ℃冰箱保存备用。
PCR扩增反应在Biometra Tone 96G PCR仪上进行,反应体系为10 μL:5 μL 2×Taq plus Mix,10 μmol/L正反向引物各0.3 μL,DNA模板2 μL,去离子水加至10 μL。扩增程序:95℃ 5 min;95℃ 30 s,55℃ 30 s,72℃ 30 s (40个循环);72 ℃ 10 min,4℃保存。PCR扩增产物在美国ABI 3730xl基因测序仪上检测,利用GeneMarker V2.2.0软件读取每个位点的片段大小。
1.4 遗传参数分析采用GenAlEx 6.502软件[22]统计分析22对SSR引物的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)和Shannon′s信息指数(I)等5个遗传多样性指标,利用Cervus 3.0.7[23]分析SSR引物的多态信息含量指数(PIC)及无效等位基因频率(Fna)。
1.5 核心种质构建方法按照杉木种质的地理来源进行分组,将864份材料分为广西、湖南、广东、福建、江西、贵州和湖北等7组,根据Core Finder软件[24](简称MC法)、Power Core软件[25] (简称MP法)、模拟退火算法(Simulated annealing algorithm)[26]以等位基因数最大化标准(简称SANA法)和模拟退火算法以遗传多样性最大化(Maximizing genetic diversity)标准(简称SAGD法)分别构建核心种质:
(1) 按照等位基因数最大化(Maximizing allelic richness)标准(M策略),运用MC法和MP法对原始种质进行分析并抽取核心种质,取样比例由软件自动设定。
(2) 根据SANA法,运用Power Marker v3.25软件构建核心种质,取样比例设置为10%、15%、20%、25%和30%。
(3) 根据SAGD法,运用Power Marker v3.25软件构建核心种质,取样比例设置为10%、15%、20%、25%和30%。
1.6 数据分析与处理核心种质构建完成后,利用SPSS 22.0对4种方法构建的核心种质库与原始种质库的相关遗传参数进行有效性t检验,利用主坐标分析法(Principal Coordinates Analysis, PCoA)生成核心种质、原始种质的分布图,进一步确认核心种质的代表性。
2 结果与分析 2.1 PCR扩增结果及遗传多样性分析对864份杉木种质的遗传多样性进行检测,结果见表 1。22个SSR标记的片段大小为96-295 bp。864份材料中:(1)检测到195个等位基因(Na),平均每对引物检测出8.86个等位基因,其中等位基因数最多的引物是SSR12(15)、SSR16(15),其次是SSR8(14),最少的是SSR2(5)、SSR13(5);(2)有效等位基因数(Ne)最多的是SSR8(6.01),其次是SSR12(5.01),最少的是SSR21(1.46),平均为3.13;(3)在观测杂合度(Ho)、期望杂合度(He)和Shannon′s信息指数(I)方面,最高的是SSR8,最低的是SSR21,平均观测杂合度、平均期望杂合度、平均Shannon′s信息指数值分别为0.62, 0.65和1.31;(4) 22对SSR引物的多态信息含量指数(PIC)值为0.30-0.82,平均值为0.60,最高的是SSR8,最低的是SSR21,其中中度多态性引物(0.25<PIC<0.5)有4对,高度多态引物(PIC>0.5)有18对; (5)22对SSR引物的无效等位基因频率(Fna)均小于0.2,均值为0.03,说明22对SSR引物对遗传多样性参数分析没有影响,结果准确可靠。
| 引物 Primers |
引物大小 Primer size (bp) |
等位基因数Na Number of alleles Na |
有效等位基因数Ne Number of effective alleles Ne |
Shannon′s信息指数I Shannon′s information index I |
观测杂合度Ho Observed heterozygosity Ho |
期望杂合度He Expected heterozygosity He |
多态信息含量指数(PIC) Polymorphic information content index (PIC) |
无效等位基因频率(Fna) Frequency of null alleles (Fna) |
| SSR1 | 218 | 7 | 2.13 | 0.95 | 0.51 | 0.53 | 0.46 | 0.02 |
| SSR2 | 173 | 5 | 2.91 | 1.17 | 0.59 | 0.66 | 0.59 | 0.06 |
| SSR3 | 233 | 7 | 3.15 | 1.24 | 0.66 | 0.68 | 0.64 | 0.03 |
| SSR4 | 260 | 9 | 3.32 | 1.46 | 0.71 | 0.70 | 0.65 | -0.01 |
| SSR5 | 171 | 13 | 3.79 | 1.67 | 0.67 | 0.74 | 0.70 | 0.05 |
| SSR6 | 205 | 6 | 2.85 | 1.22 | 0.63 | 0.65 | 0.59 | 0.01 |
| SSR7 | 249 | 11 | 3.43 | 1.44 | 0.67 | 0.71 | 0.67 | 0.03 |
| SSR8 | 246 | 14 | 6.01 | 1.99 | 0.82 | 0.83 | 0.82 | 0.01 |
| SSSR9 | 178 | 8 | 3.23 | 1.42 | 0.67 | 0.69 | 0.66 | 0.02 |
| SSR10 | 218 | 7 | 3.61 | 1.48 | 0.73 | 0.72 | 0.69 | 0.00 |
| SSR11 | 266 | 7 | 2.73 | 1.11 | 0.63 | 0.63 | 0.57 | 0.01 |
| SSR12 | 295 | 15 | 5.01 | 1.87 | 0.73 | 0.80 | 0.78 | 0.05 |
| SSR13 | 96 | 5 | 2.80 | 1.09 | 0.58 | 0.64 | 0.58 | 0.05 |
| SSR14 | 185 | 8 | 3.30 | 1.35 | 0.70 | 0.70 | 0.65 | 0.00 |
| SSR15 | 227 | 6 | 2.51 | 1.09 | 0.61 | 0.60 | 0.53 | -0.01 |
| SSR16 | 266 | 15 | 2.28 | 1.17 | 0.53 | 0.56 | 0.51 | 0.03 |
| SSR17 | 132 | 8 | 2.06 | 1.01 | 0.51 | 0.52 | 0.48 | 0.02 |
| SSR18 | 224 | 8 | 2.53 | 1.11 | 0.61 | 0.61 | 0.54 | 0.00 |
| SSR19 | 262 | 9 | 3.88 | 1.54 | 0.65 | 0.74 | 0.71 | 0.08 |
| SSR20 | 121 | 10 | 3.74 | 1.56 | 0.58 | 0.73 | 0.70 | 0.13 |
| SSR21 | 198 | 7 | 1.46 | 0.70 | 0.30 | 0.32 | 0.30 | 0.03 |
| SSR22 | 231 | 10 | 2.02 | 1.07 | 0.50 | 0.50 | 0.48 | 0.01 |
| 均值Mean | 8.86 | 3.13 | 1.31 | 0.62 | 0.65 | 0.60 | 0.03 |
2.2 4种构建方法的遗传多样性差异分析
对4种方法构建的杉木核心种质情况进行遗传多样性分析,结果见表 2。4种构建方法的等位基因数(Na)保留率均在75%以上,其中:(1)在等位基因数(Na)方面,MC法和MP法构建的核心种质Na保留比例达100%;SANA法取样比例为10%时,Na保留率最低(丢失了48个等位基因,仅保留147个); (2)在有效等位基因数(Ne)方面,SAGD法和SANA法的核心种质Ne随取样比例增大呈先降低后升高的趋势,最高的是MP法,其次是MC法,最少的是取样比例为25%的SAGD法;MP法、MC法和SAGD(25%)法Ne保留率分别为108.7%、107.6%和99.4%;(3)在Shannon′s信息指数(I)方面,MP法、MC法的核心种质高于原始种质,而取样比例为10%-30%的SANA法和SAGD法,其Shannon′s信息指数值随取样比例增加而升高,均能够保留原始种质97.7%的Shannon′s信息指数值,与原始种质相差不大; (4)在观测杂合度(Ho)、期望杂合度(He)方面,4种构建方法的核心种质保留率均在98.4%以上。SANA法、SAGD法5个取样比例构建的核心种质的等位基因数与原始种质的等位基因数存在显著差异,未能保留所有的等位基因,最高的是取样比例为30%的SANA法和SAGD法(仅保留了174个等位基因,丢失了21个),在抽取的种质数量上比MC法多了179份,但保留的等位基因数没有MC法的多。结合5个遗传多样性指标的保留率及种质数量进行综合考虑,可认为MC法、MP法构建的结果较好。
| 构建方法 Construction method |
取样比例 Sampling ratio (%) |
种质数量 Number of germplasm |
等位基因数Na Number of allele Na |
有效等位基因数Ne Number of effective allele Ne |
Shannon′s信息指数I Shannon′s information index I |
观测杂合度Ho Observed heterozygosity Ho |
期望杂合度He Expected heterozygosity He |
| 原始种质 Initial germplasm |
864 | 195 | 68.76 | 1.31 | 0.62 | 0.65 | |
| SANA法 SANA method |
10 | 86 | 147* | 69.01 | 1.28 | 0.62 | 0.64 |
| 15 | 129 | 157* | 68.68 | 1.29 | 0.61 | 0.65 | |
| 20 | 172 | 163* | 68.67 | 1.30 | 0.62 | 0.65 | |
| 25 | 216 | 169* | 68.66 | 1.30 | 0.63 | 0.65 | |
| 30 | 259 | 174* | 68.84 | 1.31 | 0.62 | 0.65 | |
| SAGD法 SAGD method |
10 | 86 | 155* | 70.04 | 1.30 | 0.61 | 0.65 |
| 15 | 129 | 160* | 69.49 | 1.31 | 0.62 | 0.65 | |
| 20 | 172 | 166* | 68.88 | 1.30 | 0.62 | 0.65 | |
| 25 | 216 | 167* | 68.34 | 1.29 | 0.62 | 0.65 | |
| 30 | 259 | 174* | 68.65 | 1.30 | 0.62 | 0.65 | |
| MC法MC method | 9.3 | 80 | 195 | 73.99 | 1.40 | 0.62 | 0.67 |
| MP法MP method | 11.3 | 98 | 195 | 74.73 | 1.41 | 0.62 | 0.68 |
| 注:*表明核心种质与原始种质在0.05水平有显著差异 Note:* indicates significant difference between core germplasm and initial germplasm at 0.05 level | |||||||
2.3 核心种质的确认
为了验证核心种质的有效性,利用SPSS 22.0分别对MC法、MP法构建的核心种质与原始种质、核心种质与保留种质的5个遗传多样性参数(Na、Ne、I、Ho和He)进行t检验,结果见表 3。结果表明:(1)MC法构建的核心种质Na、Ne、I、Ho和He的保留率分别为100%、107.6%、106.9%、100%、103.1%;在等位基因数(Na)和有效等位基因数(Ne) 2个指标上显著高于MC法的保留种质,但与原始种质没有显著差异; (2)MP法构建的核心种质Na、Ne、I、Ho和He的保留率分别为100%、108.7%、107.6%、100%、104.6%;在等位基因数(Na)和有效等位基因(Ne)2个指标上均显著高于Mp法的保留种质,与原始种质没有差异;说明MC法、MP法对原始种质的冗余材料进行了剔除,有效地选择含等位基因多的材料。
| 种质 Germplasm |
种质数 Number of germplasm |
等位基因数Na Number of alleles Na |
有效等位基因数Ne Number of effective allele Ne |
Shannon′s信息指数I Shannon′s information index I |
观测杂合度Ho Observed heterozygosity Ho |
期望杂合度He Expected heterozygosity He |
| 原始种质 Initial germplasm |
864 | 195 | 68.76 | 1.31 | 0.62 | 0.65 |
| MC法核心种质 Core germplasm by MC method |
80(9.3%) | 195(100%) | 73.99 (107.6%) | 1.40 (106.9%) | 0.62(100%) | 0.67(103.1%) |
| MC法保留种质 Reserved germplasm by MC method |
784(90.7%) | 179(91.8%) | 68.17(99.1%) | 1.29 (98.5%) | 0.62(100%) | 0.65(100%) |
| MP法核心种质 Core germplasm by MP method |
98(11.3%) | 195(100%) | 74.73(108.7%) | 1.41(107.6%) | 0.62(100%) | 0.68(104.6%) |
| MP法保留种质 Reserved germplasm by MP method |
766(88.7%) | 179(91.8%) | 67.90(98.7%) | 1.29(98.5%) | 0.62(100%) | 0.64(98.5%) |
| t1 | 0.00 | 9.06 | 1.68 | 0.07 | 0.40 | |
| t2 | 27.71* | 10.09* | 1.92 | 0.09 | 0.45 | |
| t3 | 0.00 | 10.34* | 1.85 | 0.00 | 0.52 | |
| t4 | 27.71* | 11.74* | 2.15 | 0.00 | 0.59 | |
| t5 | 0.00 | 1.28 | 0.17 | -0.07 | 0.12 | |
| 注:括号内为保留率;t1为MC法核心种质与原始种质的t检验值;t2为MC法核心种质与保留种质t检验值;t3为MP法核心种质与原始种质的t检验值;t4为MP法核心种质与保留种质的t检验值;t5为MC法核心种质与MP法核心种质的t检验值;*表示在0.05水平差异显著 Note: Retention rate in parentheses;t1 means the t-test value of core germplasm and initial germplasm by MC method;t2 means the t-test value of core germplasm and reserved germplasm by MC method;t3 means the t-test value of core germplasm and initial germplasm by MP method;t4 means the t-test value of core germplasm and reserved germplasm by MP method;t5 means the t-test value of core germplasm by MC method and core germplasm by MP method;*means significant difference at 0.05 level | ||||||
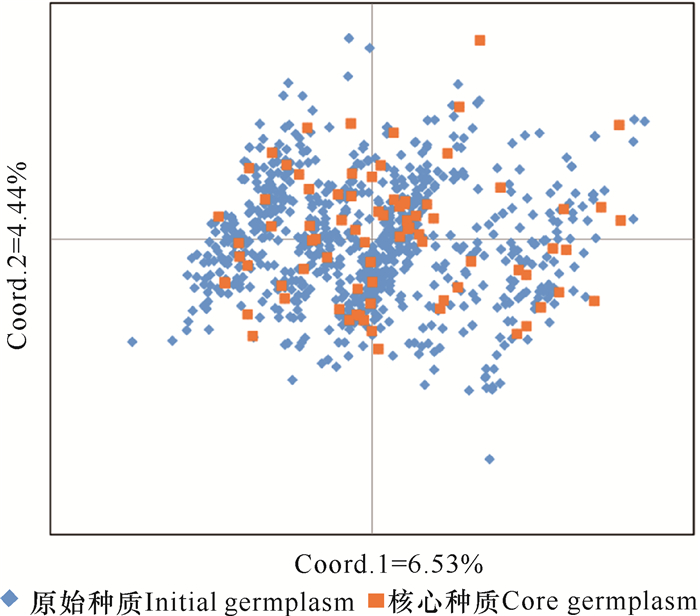
将MC法和MP法构建的核心种质进行检验比较,结果表明,两者在Na、Ne、I、Ho和He上没有差异。相较于MP法,MC法仅保留80份种质材料就能保留原始种质的所有等位基因,其遗传重复和冗余度小于MP法,因此最终选择遗传重复低、冗余度低的MC法筛选的80份材料作为广西杉木核心种质。在这80份核心种质中,地理来源为广西的种质最多,有54份(占8.9%),最少的是湖北(2份,占22.2%)(表 4)。利用主坐标分析法展示原始种质与核心种质的分布情况,对MC法核心种质作进一步确认(图 1),结果显示, 核心种质与原始种质均分布在主坐标内且范围相近,说明MC法构建的核心种质的代表性较好。
| 省区 Province |
原始种质数量 Number of Initial germplasm |
核心种质数量 Number of core germplasm |
比例 Proportion(%) |
省区 Province |
原始种质数量 Number of Initial germplasm |
核心种质数量 Number of core germplasm |
比例 Proportion(%) |
|
| 广西 Guangxi |
605 | 54 | 8.9 | 江西 Jiangxi |
40 | 4 | 10 | |
| 湖南 Hunan |
70 | 5 | 7.1 | 贵州 Guizhou |
31 | 5 | 16.1 | |
| 广东 Guangdong |
55 | 7 | 12.7 | 湖北 Hubei |
9 | 2 | 22.2 | |
| 福建 Fujian |
54 | 3 | 5.6 | 总数 Total |
864 | 80 | 9.3 |
|
| 图 1 MC法核心种质的主坐标分析 Fig. 1 Principal coordinate analysis of core germplasm by MC method |
3 讨论
目前,构建核心种质主要应用表型数据和分子标记数据[27]。多年生林木容易受到环境因素影响,采用传统调查方法得到的表型数据(树高、胸径、叶形、果实大小等)往往不能准确地反映材料间的遗传差异,且数据收集需持续多年观测调查,整个过程耗费巨大的人力和物力[28, 29]。相较于传统表型数据,DNA分子标记技术具有费用低、数据获取速度快和不受外界因素影响的特点,能够真实地反映材料间差异及亲缘关系,比表型数据更适用于构建核心种质和遗传多样性分析,被广泛应用于各类种质资源研究[30-34]。
合理的取样比例是成功构建核心种质的决定因素。国内外研究报道中,大多数取样比例为5%-40%[4, 8, 35-37]。吴昊等[38]利用苗期生长量及种子质量性状的表型数据构建了野生榧树Torreya grandis种群的初级核心种质,取样比例为36%,并利用分子标记检测初级核心种质,剔除了部分遗传重复的材料,最终保留了15份材料,占原始种质的10.56%;钟永达等[39]基于幼苗及种子的表型数据,用4种不同聚类方法从872份材料中构建了含218份种质材料的中国樟树Cinnamomum camphora初级核心种质,取样比例为25%;彭婵等[40]报道了含45份南方型美洲黑杨Populus deltoides的初级核心种质,占原始种质的15%。这些研究结果表明,取样比例随原始种质材料的数量增多而降低。一般来说,由于物种的独特性,其取样比例主要由树种、遗传结构、遗传多样性及种质数量规模决定。本研究中共有864份杉木种质材料,将不同取样比例的结果进行对比分析,其中SANA法和SAGD法的等位基因数随着取样比例增加而增加,但均未能保留原始种质的全部基因,说明这2种方法在构建核心种质时并未能筛选出等位基因数多的材料,存在遗传重复的材料,构建效果较差。而MC法构建结果的取样比例最低(占原始种质的9.3%,筛选出80份种质材料),取样比例低于前人的研究结果[41-43],但在等位基因数(Na)、有效等位基因数(Ne)、Shannon′s信息指数(I)、观测杂合度(Ho)和期望杂合度(He)的保留率上均达到100%,可见Mc法能用较少的种质保留原始种质所有等位基因数,保留效果较好。
不同构建方法直接影响核心种质的代表性和有效性。在进行核心种质构建时,应遵循优先选择特异基因和等位基因数目多的材料,以及尽可能保留含多等位基因的原则,以保证获得的核心种质具有最多的等位基因[44, 45]。M策略是以每个位点的等位基因数目最大化为标准,主要通过Core Finder[24]、Power Core[25]和Power Mstrat[46]这3款软件实现,在构建过程中能够剔除材料中的遗传重复,筛选出具等位基因数目多且冗余度低的材料,是目前最具优势的构建方法之一。本研究的MC法、MP法、SANA法是以等位基因数最大化为标准,能够筛选出具等位基因多的材料,而SAGD法是以遗传多样性最大化为标准,筛选出的材料具有较大遗传多样性。结果发现,SANA法和SAGD法构建结果不佳,而Core Finder软件和Power Core软件构建结果的Na、Ne、I、Ho、He等5个遗传参数的保留率高于SANA法和SAGD法,说明MC法和MP法优于SAGD法和SANA法。在选择核心种质材料时,MC法、MP法均选择等位基因数目多且冗余度低的材料,相比而言,Core Finder软件的构建结果比Power Core软件的少了18份,仅用80份核心种质保留了864份原始种质中的全部等位基因,其构成的核心种质库更为精简,遗传重复和冗余度更低。
广西是全国杉木主产区之一,1976年和1979年,广西区内29个种源参加全国杉木产区的种源试验,广西融水种源在两次试验中表现突出,其因适应范围广,单株材积大、树高和胸径生长量高且稳定,被认定为全国杉木优良种源之一。广西属典型的喀斯特地貌,地形复杂多变,山地丘陵居多,北回归线横穿境内,南北温差大,各地种源在生长量和适应性方面存在差异,具较强的地域性。受喀斯特地理隔离影响,广西境内还有一些特异的种质资源,如念水糠杉、白云糠杉和四荣红心杉等[20, 47, 48]。融江河流域位于全国优良种源南岭的西部,是广西杉木的中心产区[49-51]。本研究广西的605份材料中,主要由融水(262份)、融安(152份)、河池(75份)、桂林(36份)、玉林(34份)、百色(19份)、梧州(13份)、南宁(7份)和钦州(7份)等9个不同市县的种质构成。入选的80份核心种质中,有54份材料来自广西,其中广西杉木中心产区来自融水县(20份)、融安县(15份)的种质材料居多,其余的分别来自河池(7份)、百色(5份)、桂林(3份)、梧州(2份)、玉林(2份)和钦州(1份),本次入选的种质材料基本涵盖了整个广西杉木分布区。未入选核心种质的材料仍不能随意舍弃,当核心种质遭受自然灾害发生衰亡或无当前生产实践所需的高产优质和抗逆性强的重要性状时,可从保留种质中寻找相似材料进行补充[24]。随着核心种质的深入研究,需要及时对核心种质遗传结构和材料进行调整与更新,不断增补新发现的特异材料,使其具有更丰富的遗传变异,更合理的遗传结构。
本研究利用SSR分子标记数据,通过对比分析不同构建方法的差异,从而确定以等位基因数最大化为标准(M策略)、通过Core Finder软件构建广西杉木核心种质的构建方法。由于缺少生长量、抗逆性等表型数据以及SSR分子标记数量的限制,并没有将表型数据和分子数据结合起来构建,今后的研究中仍需收集调查表型生长性状,将两者相结合起来,对核心种质作进一步全面分析和调整。
4 结论本研究基于SSR分子标记技术,以广西杉木864份种质为材料,通过构建方法筛选,最终确定M策略(Core Finder软件)构建方法,并构建了含80份材料的广西杉木核心种质,其中,种质数占原始种质的9.3%,Na、Ne、I、Ho和He遗传多样性参数的保留率达100%,经主坐标分析和t检验,验证所构建的核心种质与原始种质差异不显著,分布范围与原始种质基本一致,说明构建结果有效且具较好的代表性,可为广西杉木种质资源优异基因资源挖掘、创新利用提供理论依据与材料基础。
| [1] |
ESCRIBANO P, VIRUEL M A, HORMAZA J I. Comparison of different methods to construct a core germplasm collection in woody perennial species with simple sequence repeat markers.A case study in cherimoya (Annona cherimola, Annonaceae), an underutilised subtropical fruit tree species[J]. Annals of Applied Biology, 2008, 153(1): 25-32. DOI:10.1111/j.1744-7348.2008.00232.x |
| [2] |
FRANKEL O H, BROWN A H D. Plant genetic reso-urces today: A critical appraisal[M]//HOLDEN J H W, WILLIAMS J T. Crop genetic resources: Conservation and evaluation. London: George Allan and Unwin, 1984: 249-257.
|
| [3] |
BROWN A H D. Core collections: A practical approach to genetic resources management[J]. Genome, 1989, 31(2): 818-824. DOI:10.1139/g89-144 |
| [4] |
董玉琛, 曹永生, 张学勇, 等. 中国普通小麦初选核心种质的产生[J]. 植物遗传资源学报, 2003, 4(1): 1-8. DOI:10.3969/j.issn.1672-1810.2003.01.002 |
| [5] |
WANG J C, HU J, XU H M, et al. A strategy on constructing core collections by least distance stepwise sampling[J]. Theoretical and Applied Genetics, 2007, 115(1): 1-8. DOI:10.1007/s00122-007-0533-1 |
| [6] |
ZHANG H L, ZHANG D L, WANG M X, et al. A core collection and mini core collection of Oryza sativa L.in China[J]. Theoretical & Applied Genetics, 2011, 122(1): 49-61. |
| [7] |
王丽侠, 程须珍, 王素华. 中国饭豆种质资源遗传多样性及核心种质构建[J]. 植物遗传资源学报, 2014, 15(2): 242-247. |
| [8] |
姜俊烨, 杨涛, 王芳, 等. 国内外蚕豆核心种质SSR遗传多样性对比及微核心种质构建[J]. 作物学报, 2014, 40(7): 1311-1319. DOI:10.3969/j.issn.1000-2561.2014.07.011 |
| [9] |
魏志刚, 高玉池, 刘桂丰, 等. 白桦核心种质初步构建[J]. 林业科学, 2009, 45(10): 74-80. |
| [10] |
张艳芳. 利用形态学和SSR标记建立柿核心种质[D]. 武汉: 华中农业大学, 2010. http://cdmd.cnki.com.cn/article/cdmd-10504-2010271732.htm
|
| [11] |
李秀兰, 贾继文, 王军辉, 等. 灰楸形态多样性分析及核心种质初步构建[J]. 植物遗传资源学报, 2013, 14(2): 243-248. DOI:10.3969/j.issn.1672-1810.2013.02.009 |
| [12] |
曾宪君, 李丹, 胡彦鹏, 等. 欧洲黑杨优质核心种质库的初步构建[J]. 林业科学, 2014, 50(9): 51-58. |
| [13] |
刘德浩, 张卫华, 张方秋. 尾叶桉核心种质初步构建[J]. 华南农业大学学报: 自然科学版, 2014, 35(6): 89-93. |
| [14] |
MIYAMOTO N, ONO M, WATANABE A. Con-struction of a core collection and evaluation of genetic resources for Cryptomeria japonica (Japanese cedar)[J]. Journal of Forest Research, 2015, 20(1): 186-196. DOI:10.1007/s10310-014-0460-3 |
| [15] |
王萱, 刘晓静, 邢世岩, 等. 中国部分古银杏资源遗传多样性的AFLP分析及核心种质的构建[J]. 园艺学报, 2016, 43(2): 249-260. |
| [16] |
杨汉波, 张蕊, 王帮顺, 等. 基于SSR标记的木荷核心种质构建[J]. 林业科学, 2017, 53(6): 37-46. |
| [17] |
段帆, 张欢, 李珊, 等. 利用ISSR分子标记构建濒危植物水青树核心种质[J]. 亚热带植物科学, 2018, 47(2): 101-106. |
| [18] |
阮梓材. 杉木遗传改良[M]. 广州: 广东科技出版社, 2003.
|
| [19] |
陈代喜, 莫钊志. 广西杉木遗传改良进展及发展策略[J]. 广西林业科学, 1993, 22(3): 108-112. |
| [20] |
陈代喜, 莫钊志, 班汉珍, 等. 广西杉木优良种质资源保存和利用现状[J]. 广西林业科学, 2002, 31(3): 128-129. |
| [21] |
LI K P, CHEN S C, DONG L M, et al. Characterization of genomic microsatellite markers and analysis of pollen donors number for single cone of Chinese fir[J]. Tree Genetics and Molecular Breeding, 2020, 10(5): 1-7. |
| [22] |
PEAKALL R, SMOUSE P E. GenAlEx 6.5:Genetic analysis in Excel.Population genetic software for teaching and research-An update[J]. Bioinformatics, 2012, 28(19): 2537-2539. |
| [23] |
KARAKET T, POOMPUANG S. COLONY for successful parentage and sibship determinations in freshwater prawn Macrobrachium rosenbergii de Man[J]. Aquaculture, 2012, 324/325: 307-311. |
| [24] |
CIPRIANI G, SPADOTTO A, JURMAN I, et al. The SSR-based molecular profile of 1005 grapevine (Vitis vinifera L.) accessions uncovers new synonymy and parentages, and reveals a large admixture amongst varieties of different geographic origin[J]. Theoretical & Applied Genetics, 2010, 121(8): 1569-1585. |
| [25] |
KIM K W, CHUNG H K, CHO G T, et al. Power-Core: A program applying the advanced M strategy with a heuristic search for establishing core sets[J]. Bioinformatics, 2007, 23(16): 2155-2162. |
| [26] |
LIU K J, MUSE S V. PowerMarker: An integrated analysis environment for genetic marker analysis[J]. Bioinformatics, 2005, 21(9): 2128-2129. |
| [27] |
王建成, 胡晋, 黄歆贤, 等. 植物遗传资源核心种质新概念与应用进展[J]. 种子, 2008, 27(5): 47-50. |
| [28] |
王红霞, 赵书岗, 高仪, 等. 基于AFLP分子标记的核桃核心种质的构建[J]. 中国农业科学, 2013, 46(23): 4985-4995. |
| [29] |
徐海明, 胡晋, 邱英雄. 利用分子标记和数量性状基因型值构建作物核心种质库的研究[J]. 生物数学学报, 2005, 20(3): 97-101. |
| [30] |
刘新龙, 刘洪博, 马丽, 等. 利用分子标记数据逐步聚类取样构建甘蔗杂交品种核心种质库[J]. 作物学报, 2014, 40(11): 1885-1894. |
| [31] |
唐源江, 曹雯静, 吴坤林. 基于SRAP标记的国兰种质资源遗传多样性分析及分子身份证构建[J]. 中国农业科学, 2015, 48(9): 1795-1806. |
| [32] |
燕丽萍, 吴德军, 毛秀红, 等. 基于SSR荧光标记的白蜡核心种质构建[J]. 中南林业科技大学学报, 2019, 39(7): 1-9. |
| [33] |
常利芳, 白建荣, 李锐, 等. 基于SSR标记构建甜玉米群体的核心种质[J]. 玉米科学, 2018, 26(3): 40-49. |
| [34] |
杨美, 付杰, 向巧彦, 等. 利用AFLP分子标记技术构建花莲核心种质资源[J]. 中国农业科学, 2011, 44(15): 3193-205. |
| [35] |
李自超, 张洪亮, 曹永生, 等. 中国地方稻种资源初级核心种质取样策略研究[J]. 作物学报, 2003, 29(1): 20-24. |
| [36] |
崔艳华, 邱丽娟, 常汝镇, 等. 植物核心种质研究进展[J]. 植物遗传资源学报, 2003, 4(3): 279-284. |
| [37] |
WANG Y, FU J X, PAN L J, et al. Construction of core collection of lychee by SSR marker[J]. Acta Horticulturae, 2014, 1029(1029): 87-92. |
| [38] |
吴昊, 喻卫武, 吴慧敏, 等. 基于种子性状及分子标记分析确定榧树核心种质[J]. 分子植物育种, 2019, 17(16): 5513-5520. |
| [39] |
钟永达, 刘立盘, 李彦强, 等. 中国樟树初级核心种质取样方法与策略研究[J]. 西南林业大学学报: 自然科学, 2020, 40(4): 1-13. |
| [40] |
彭婵, 樊孝萍, 苏晓华, 等. 基于SSR分子标记构建南方型美洲黑杨初级核心种质[J]. 西北植物学报, 2019, 39(2): 250-257. |
| [41] |
黄雨芹, 尹光天, 杨锦昌, 等. 基于SSR分子标记的闽楠(Phoebe bournei)核心种质的构建[J]. 分子植物育种, 2020, 18(8): 2641-2648. |
| [42] |
毛秀红, 朱士利, 李善文, 等. 基于荧光SSR标记的毛白杨核心种质构建[J]. 北京林业大学学报, 2020, 42(7): 40-47. |
| [43] |
杨欣超, 张凯权, 王静, 等. 基于SSR分子标记的刺槐遗传多样性分析及核心种质的构建[J]. 分子植物育种, 2020, 18(9): 3086-3097. |
| [44] |
刘娟, 廖康, 曼苏尔·那斯尔, 等. 利用ISSR分子标记构建南疆杏种质资源核心种质[J]. 果树学报, 2015, 32(3): 374-384. |
| [45] |
郭大龙, 刘崇怀, 张君玉, 等. 葡萄核心种质的构建[J]. 中国农业科学, 2012, 45(6): 1135-1143. |
| [46] |
GOUESNARD B, BATAILLON T M, DECOUX G, et al. MSTRAT: An algorithm for building germ plasm core collections by maximizing allelic or phenotypic richness[J]. Journal of Heredity, 2001, 92(1): 93-94. |
| [47] |
李魁鹏, 韦正成, 黄开勇, 等. 广西融水特色红心杉木优树材质性状变异规律研究[J]. 林业科学研究, 2017, 30(3): 424-429. |
| [48] |
戴俊, 谭文婧, 黄开勇, 等. 红心杉不同无性系结实特性及球果类型分析[J]. 广西林业科学, 2019, 48(2): 141-146. |
| [49] |
全国杉木种源试验协作组. 杉木造林区种源选择[J]. 林业科学研究, 1988, 1(1): 1-13. |
| [50] |
广西杉木地理种源试验协作组. 广西杉木种源区划分和造林优良种源选择[J]. 江西林业科技, 1994(3/4): 69-74. |
| [51] |
雷布先, 秦武明, 罗耀峰. 融江流域杉木优良林分数量化立地分类及评价研究[J]. 广西林业科技, 1993, 22(2): 68-77. |



