2. 广西海洋研究所有限责任公司, 广西海水养殖新品种繁育工程技术研究中心, 广西北海 536000;
3. 广西壮族自治区水产科学研究院, 广西水产遗传育种与健康养殖重点实验室, 广西南宁 530021
2. Guangxi Engineering Technology Research Center for Marine Aquaculture, Guangxi Institute of Oceanology Co., Ltd., Beihai, Guangxi, 536000, China;
3. Guangxi Key Laboratory of Aquatic Genetic Breeding and Healthy Aquaculture, Guangxi Academy of Fishery Sciences, Nanning, Guangxi, 530021, China
褐石斑鱼(Epinephelus bruneus)隶属鲈形目(Perciformes)鮨科(Serranidae)石斑鱼亚科(Epinephelinae)石斑鱼属(Epinephelus),俗称土沟龙、假油斑,为西太平洋海区暖水性底层鱼类,主要分布在韩国、日本和中国(南至海南岛)沿海[1]。褐石斑鱼具有生长快、适应能力强、营养价值高等特点,养殖开发潜力巨大[2]。20世纪90年代末,日本、韩国和中国台湾地区开始养殖褐石斑鱼[3]。然而,褐石斑鱼人工规模化育苗技术尚未突破,目前人工养殖的苗种主要来源于野生捕捞[4]。此外,由于褐石斑鱼野生种群数量逐年下降,世界自然保护联盟(International Union for Conservation of Nature, IUCN)于2016年将褐石斑鱼收录到濒危物种红色名录下,现已被列为易危物种,其种质资源极易因人类活动的影响而衰退[5],因此急需开展褐石斑鱼种群遗传多样性和遗传结构等遗传背景相关的调查分析。目前,关于褐石斑鱼的研究主要集中在生理学[3]、早期发育[6]、细胞遗传学[7]、杂交育种[4]和人工养殖[8]等方面,尚未见有关褐石斑鱼地理种群的遗传多样性和遗传结构的研究报道。
动物线粒体DNA(mitochondrial DNA, mtDNA)具有结构简单、严格的母系遗传、几乎不发生重组、进化速度快且不同区域进化速度存在差异等特点,已成为研究动物起源进化、群体遗传、系统发育等的重要标记[9]。其中,线粒体细胞色素b (Cytb)基因的结构和功能在mtDNA的13个蛋白质编码基因中研究得最多,且进化速度适中,因此在鱼类的种群分化与遗传结构中应用广泛[10]。本研究以西太平洋海区3个不同地理群体的褐石斑鱼为研究对象,利用线粒体Cytb基因序列对褐石斑鱼分布区域内的种群遗传多样性和遗传结构进行分析,旨在从分子水平上揭示褐石斑鱼地理种群间的遗传差异和种群分化,不仅为褐石斑鱼种质资源遗传背景调查分析提供基础数据,而且为褐石斑鱼种质资源保护和利用、人工繁育、生态养殖以及良种选育提供更多的理论依据。
1 材料与方法 1.1 实验材料褐石斑鱼分别采自中国海南岛(109.226°N, 20.140°E)、福建厦门(118.241°N, 24.302°E)和韩国济州岛(126.594°N, 33.974°E) 3个近海海域,为当地渔船出海钓捕收获。其中,采集海南岛(HN)地理种群30尾,体长(26.4±2.6) cm,体质量(426.8±52.7) g;福建厦门(XM)地理种群27尾,体长(21.7±3.8) cm,体质量(309.7±65.4) g;韩国济州岛(HG)地理种群31尾,体长(25.2±2.7) cm,体质量(393.5±49.6) g。根据郭明兰等[11]和Liu等[12]对云纹石斑鱼(E.moara)和褐石斑鱼形态比较的特征鉴定为褐石斑鱼,解剖取其背部肌肉,放置于-20 ℃的无水乙醇中保存备用。
1.2 基因组DNA的提取和检测取每个样品0.1 g,用SDS/蛋白酶K消化,采用酚-氯仿抽提法和乙醇沉淀法[13]提取总DNA,用1.0%琼脂糖凝胶电泳检测DNA,并使用超微量紫外分光光度计测定DNA浓度并稀释至100 ng/μL,于-20 ℃冰箱中保存备用。
1.3 PCR扩增及序列测定Cytb基因片段扩增的引物序列为L14724 (5′-GACTTGAAAAACCACCGTTG-3′)和H15149 (5′-CCTCAGAAGGATATTGTCCTC-3′),由北京六合华大基因科技股份有限公司合成。PCR扩增反应体积为50 μL,其中10×PCR buffer (plus Mg2+) 5 μL,dNTPs (10 mmol/L) 1 μL,正反向引物(5 pmol/μL)各1 μL,Taq酶(2.5 U/μL) 1 μL,模板(约100 ng) 1 μL,加超纯水至50 μL。PCR扩增反应条件:94 ℃预变性3 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸45 s,35个循环;再72 ℃链延伸7 min。PCR产物经1%琼脂糖凝胶电泳检测,柱式纯化试剂盒纯化,纯化产物送北京六合华大基因科技股份有限公司进行双向测序。
1.4 数据分析选用Dnastar软件包对序列进行编辑、校对和排序,并对排序结果进行分析和手工调整。用DnaSP软件[14]确定单倍型。单倍型数目、多态位点、转换、颠换、插入/缺失等分子多态性参数使用Arlequin软件[15]统计获得。单倍型多态度(h)、核苷酸多态度(π)、两两序列平均核苷酸差异数(k)以及相应的方差使用Arlequin软件计算。
以巨石斑鱼(E.tauvina)和斜带石斑鱼(E.coioides)的Cytb基因序列为外群,采用jModelTest软件[16]依据贝叶斯信息标准(Bayesian Information Criterion, BIC)从88种核苷酸替换模型中选取最适合本研究的线粒体Cytb基因的最佳替换模型。采用邻接法(Neighbor Joining,NJ),最大简约法(Maximum Parsimony,MP)和最大似然法(Maximum Likelihood,ML)等3种方法构建褐石斑鱼Cytb单倍型分子系统树,使用PAUP软件构建系统树[17]。系统树的可靠性采用1 000次重抽样进行评估,应用NETWORK软件(http://www.fluxus-technology.com)构建单倍型网络关系图。
应用Arlequin软件计算两两群体间的遗传分化指数(FST),检测群体遗传结构,FST值的显著性使用10 000次重抽样检验。单倍型间的遗传距离采用HKY核苷酸进化模型计算。采用分子方差分析(AMOVA)[18]评估褐石斑鱼的群体遗传结构,通过10 000次重抽样来检验不同遗传结构水平上协方差的显著性。采用IBDWS (Isolation by Distance Web Service)[19]检验群体间是否存在距离隔离(Isolation by Distance, IBD)[20]。采用Tajima′s D检验[21]和Fu′s Fs检验[22]两种检验方法进行群体历史动态分析,使用Arlequin软件进行运算。
2 结果与分析 2.1 Cytb基因序列遗传变异和遗传多样性经序列比对并去除引物序列后,获得的DNA序列片段长402 bp,该片段起始的1-3 bp碱基序列为ATG,对应斜带石斑鱼线粒体基因组全序列中的Cytb基因序列的起始密码子[23]。结果显示,本次测序序列为褐石斑鱼线粒体Cytb基因部分序列,其A+T含量(54.2%)高于C+G含量(45.8%)。褐石斑鱼Cytb基因序列遗传变异水平较低,在目的片段内仅检测到18个多态位点,均发生在密码子第3位点,为T/C和A/G两种转换突变。
在88个个体中,共检测到7种单倍型,其中Hap1为3个群体(71个个体)所共有,Hap6为韩国和中国厦门群体共有,而其他单倍型在群体间分布不均,每个地理群体都有其各自特有的单倍型(表 1)。由表 2可知,韩国群体的单倍型类型较中国海南和厦门群体丰富,其单倍型多态度最高,为0.556 67。各群体内平均核苷酸差异度和核苷酸多态度均随突变位点数目的增加而增加,呈正比例关系,韩国群体个体间的平均核苷酸差异度(0.613 33)、核苷酸多态度(0.001 53)均高于中国海南和厦门群体。褐石斑鱼中国海南群体(h=0.338 10,π=0.000 88)和厦门群体(h=0.323 53,π=0.001 13)均属于低单倍型多态度和低核苷酸多态度类群;褐石斑鱼韩国群体(h=0.556 67,π=0.001 53)属于高单倍型多态度和低核苷酸多态度类群。综上可知,西太平洋海区褐石斑鱼总体遗传多样性水平较低,韩国群体的遗传多样性水平高于中国海区的海南群体和厦门群体。
| 群体Population | 单倍型数量分布Number distribution of haplotypes | 总量Overall | ||||||
| Hap1 | Hap2 | Hap3 | Hap4 | Hap5 | Hap6 | Hap7 | ||
| HN | 26 | 1 | 3 | 30 | ||||
| HG | 21 | 1 | 8 | 1 | 31 | |||
| XM | 24 | 1 | 2 | 27 | ||||
| Total | 71 | 1 | 3 | 1 | 1 | 10 | 1 | 88 |
| 群体Population | 样品数Sample size | 单倍型数Haplotype number | 单倍型多态度h | 核苷酸多态度π | 平均核苷酸差异度k |
| HN | 30 | 3 | 0.338 10 | 0.000 88 | 0.352 38 |
| HG | 31 | 4 | 0.556 67 | 0.001 53 | 0.613 33 |
| XM | 27 | 3 | 0.323 53 | 0.001 13 | 0.455 88 |
| Pooled | 88 | 7 | 0.445 47 | 0.001 29 | 0.519 20 |
2.2 单倍型谱系结构
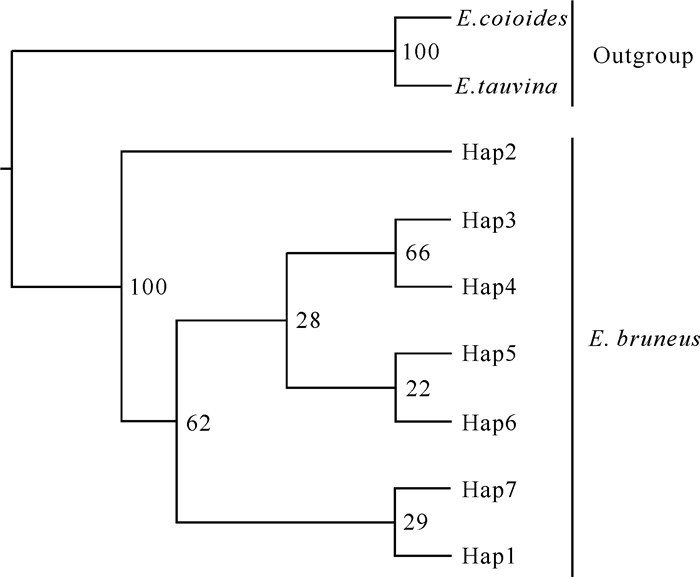
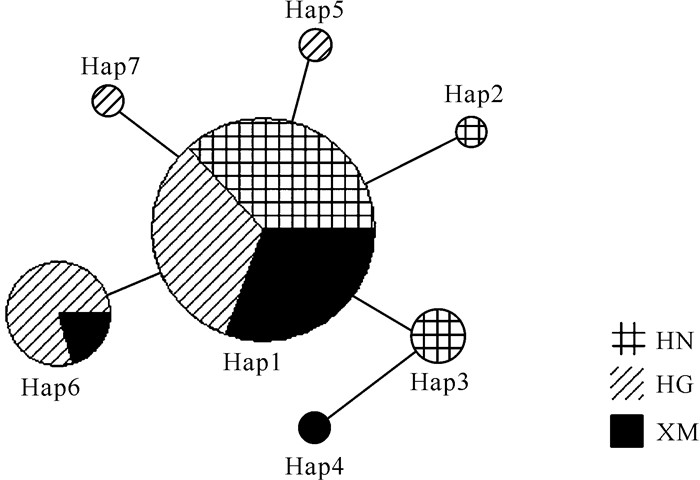
ModelTest结果显示,褐石斑鱼3个地理群体的Cytb基因部分序列的最佳模型是HKY。使用PAUP软件构建了褐石斑鱼单倍型NJ、MP和ML树,结果显示3种方法得出的进化树拓扑结构一致。褐石斑鱼的Hap3、Hap4、Hap5、Hap6、Hap7和Hap1单倍型首先聚合成一个进化分支,然后与Hap2单倍型聚合成褐石斑鱼进化分支,最后与巨石斑鱼和斜带石斑鱼外群分支聚合。总体而言,褐石斑鱼单倍型进化树的拓扑结构较为简单,并且3个地理种群的单倍型不能依据地理分布各自聚类,而是相互聚类为一个大的进化分支,不存在明显的地理谱系结构(图 1)。采用中介网络图法构建褐石斑鱼单倍型的网络关系图,结果显示褐石斑鱼的单倍型网络图为星形结构,不同地理分布的单倍型没有单独聚类,与系统进化树的谱系结构类似,不存在显著的地理谱系结构(图 2)。
|
| 图 1 褐石斑鱼线粒体Cytb基因单倍型的最大释然进化树 Fig. 1 Maximum likelihood phylogenetic tree of the mtDNA Cytb gene haplotypes for E.bruneus |
|
| 图 2 褐石斑鱼3个群体单倍型网络图 Fig. 2 Haplotype network diagram of 3 populations diagram of E.bruneus |
2.3 群体遗传分化
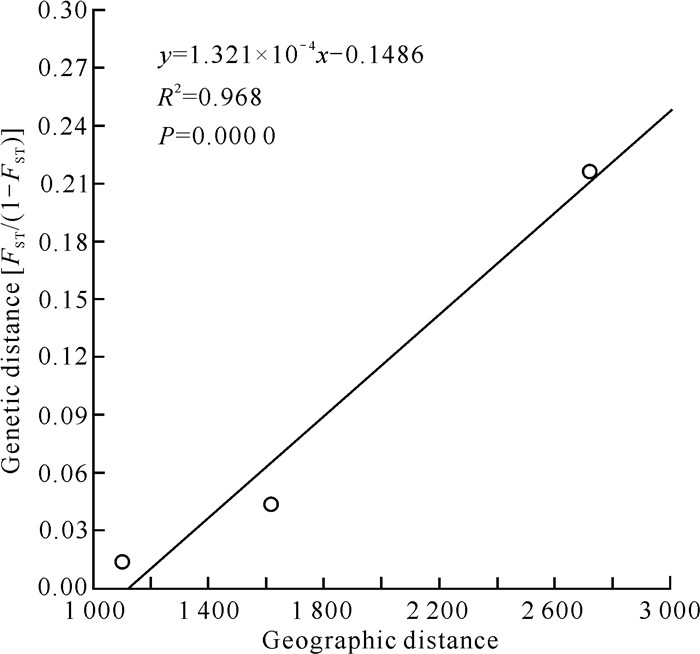
对3个群体的遗传分化进行分子方差分析(表 3),结果显示90.28%的遗传差异来源于群体内,仅9.72%的遗传差异来源于群体间,统计结果差异显著(P=0.005 25),表明褐石斑鱼遗传变异主要来源于群体内,不同群体间存在中等水平的遗传差异,群体间的遗传分化指数(FST)为0.097 16。根据HKY距离模型计算两两群体间的FST值(表 4),结果显示韩国与中国海南群体间遗传分化最高(FST为0.177 5),中国海南与厦门群体间遗传分化最低(FST为0.013 4)。综上,西太平洋海区褐石斑鱼地理群体间存在不同程度的遗传分化,并且分化程度大小与地理距离相关。Mantel检验结果显示,褐石斑鱼群体间遗传距离和地理距离间存在显著相关,褐石斑鱼地理群体间遗传距离与群体间的地理距离成线性相关(图 3)。
| 变异来源Source of variation | 变异组分Variance compo- nents | 变异贡献率/% Variation contribution rate/% | F检验F statistics | P值P value |
| Among populations | 0.026 15 | 9.72 | 0.097 16 | 0.005 25 |
| Within populations | 0.242 96 | 90.28 | ||
| Total | 0.269 11 |
| 中国海南岛HN | 韩国济州岛HG | 中国福建厦门XM | |
| HN | 0.001 3 | 0.256 1 | |
| HG | 0.177 5 | 0.108 2 | |
| XM | 0.013 4 | 0.041 7 |
|
| 图 3 褐石斑鱼两两群体间地理距离与遗传距离FST/(1-FST)的回归分布 Fig. 3 Regression distribution of geographic distance and genetic distance FST/(1-FST) between two groups of E.bruneus |
2.4 群体扩张历史特征
对褐石斑鱼3个地理群体以及其合并群体分别进行Tajima′s D检验和Fu′s Fs检验(表 5),结果显示褐石斑鱼中国海南、厦门和韩国地理群体Tajima′s D和Fu′s Fs值均为负值,统计检验结果不显著(P>0.05)。褐石斑鱼合并群体的Tajima′s D和Fu′s Fs值为较大的负值,统计检验结果显著(P<0.05)。表明褐石斑鱼3个地理种群的线粒体Cytb基因符合中性进化,西北太平洋海区的褐石斑鱼种群可能发生了群体扩张的历史动态。
| 群体Population | Tajima′s D值Tajima′s D value | Fu′s Fs值Fu′s Fs value | |||
| D | P | Fs | P | ||
| HN | -0.843 2 | 0.213 7 | -0.822 7 | 0.185 1 | |
| HG | -0.566 3 | 0.328 2 | -0.901 9 | 0.224 6 | |
| XM | -1.377 1 | 0.082 1 | -0.502 4 | 0.249 4 | |
| Pooled | -1.441 5 | 0.049 5 | -4.195 6 | 0.006 4 | |
3 讨论 3.1 褐石斑鱼的遗传多样性
褐石斑鱼为暖水性底层鱼类,主要分布于西太平洋中国、日本、韩国等3个国家,其生长速度快、适应能力强、经济价值高,有潜力成为我国石斑鱼养殖的名贵新品种。然而,有关褐石斑鱼种质资源遗传背景调查,尤其是种质遗传多样性评估尚未开展系统研究,不利于我国褐石斑鱼种质资源的保护和可持续开发利用。遗传多样性是鱼类生物多样性的重要组成部分,种内遗传多样性越丰富,物种对环境的适应能力越强[24]。线粒体DNA的单倍型多态度(h)和核苷酸多态度(π)是衡量种群遗传多样性的重要指标。Grant等[25]提出鱼类DNA单倍型多态度和核苷酸多态度高低水平的临界值分别是0.5和0.005,根据不同的临界值,可将鱼类遗传多样性分为高单倍型多态度(h>0.5)和高核苷酸多态度(π>0.005)、高单倍型多态度(h>0.5)和低核苷酸多态度(π < 0.005)、低单倍型多态度(h < 0.5)和高核苷酸多态度(π>0.005)以及低单倍型多态度(h < 0.5)和低核苷酸多态度(π < 0.005)等4种类型。本研究结果显示,褐石斑鱼3个地理群体合并后的单倍型多态度(h=0.445 47)和核苷酸多态度(π=0.001 29)属于低单倍型多态度和低核苷酸多态度类群,低于大西洋真鳕(Gadus morhua,h=0.97,π=0.018 6)[26]、小黄鱼(Larimichthys polyactis,h=0.989,π=0.004 8)[27]等海洋鱼类的Cytb基因序列遗传多样性。石斑鱼属的斜带石斑鱼(h=0.740,π=0.003 69)[28]、棕点石斑鱼(E.fuscoguttatus,h=0.747,π=0.004 14)[29]的Cytb基因序列遗传多样性高于本研究的褐石斑鱼群体。推测可能是由于人类对褐石斑鱼过度捕捞及各种活动对其栖息地造成破坏[7],导致其繁殖群体数量大幅度下降,从而使褐石斑鱼遗传多样性水平下降。在非洲海区的安氏石斑鱼(E.andersoni)的遗传多样性研究中也有类似的结论,由于人类捕捞活动造成安氏石斑鱼野生种群数量下降,从而导致其种群遗传多样性水平较低(h=0.367,π=0.001)[30]。本研究结果提示需要加强中国海区褐石斑鱼野生种质资源的保护,人工繁育的亲本有必要补充遗传多样性较高的韩国亲本来保证繁育后代的遗传多样性水平。
3.2 褐石斑鱼群体遗传分化遗传分化指数(FST)是衡量不同地理群体间遗传差异的重要指标。Balloux等[31]依据遗传分化指数的临界值,将群体遗传分化程度划分为4种类型:极小遗传分化类型(0 < FST < 0.05)、中度遗传分化类型(0.05 < FST < 0.15)、较大遗传分化类型(0.15 < FST < 0.25)以及高度遗传分化类型(FST>0.25)。本研究中两两群体间的FST分析结果显示,地理距离最远的韩国和中国海南群体间遗传分化程度最高(FST为0.177 5),属于较大遗传分化类型; 地理距离最近的中国海南和厦门群体间遗传分化最低(FST为0.013 4), 属于极小遗传分化类型;分子方差分析结果也显示,褐石斑鱼3个地理群体间的遗传分化程度为中度遗传分化类型(FST为0.097 16)。地理隔离是群体间基因交流的天然屏障,在海洋这个开放的环境中,通常认为距离隔离是促使遗传分化产生的主要机制[32]。王登玉等[28]对中国东南近海斜带石斑鱼群体遗传分化的研究表明,斜带石斑鱼地理群体间存在极大的遗传分化(FST为0.344),并且距离隔离是斜带石斑鱼3个地理群体间遗传分化的主要因素。傅蒙娜等[33]基于线粒体Cytb基因开展中国海区3个条纹斑竹鲨(Chiloscyllium plagiosum)群体遗传分化的研究也表明,地理距离较远的福建群体和海南群体遗传分化差异显著,距离隔离是条纹斑竹鲨群体遗传分化的重要因素。本研究中Mantel检验结果显示褐石斑鱼群体间遗传距离FST/(1-FST)和地理距离间存在显著相关(r=0.984 0),并且统计检验极显著(P=0.000 0),表明距离隔离是褐石斑鱼群体遗传分化的主要因素。Richardson等[34]在对黑缘石斑鱼(E.morio)的线粒体DNA遗传多样性检测中发现其稚鱼及成鱼的定居习性,导致群体间遗传分化显著。褐石斑鱼为暖水性底层鱼类,栖息于岩礁和底质为沙泥质的海域,成鱼栖息深度为20-200 m,幼鱼经常出现在浅水区[1],是具有较强领域性的定居种。褐石斑鱼的这种生活特性可能是造成其群体存在距离隔离遗传分化的主要原因。由于褐石斑鱼不同地理群体间存在遗传分化,所以在褐石斑鱼人工繁育时,可以选择来自不同地理群体的亲体,来提高繁殖群体的遗传分化水平,避免人工繁育后代出现近交衰退而导致生产性能下降。
3.3 褐石斑鱼群体扩张历史特征Slatkin等[20]指出自然种群遭受瓶颈效应后的群体快速扩张可以导致出现星状结构的基因谱系。本研究中褐石斑鱼单倍型网络图表现为简单星形结构,符合群体扩张历史特征。在丝鳍紫鱼(Pristipomoides filamentosus)[35]和安氏石斑鱼[30]等海洋鱼类中也存在类似的结果,因为群体扩张历史导致单倍型网络图呈现简单星状拓扑结构。Tajima′s D和Fu′s Fs检验是两种常用的检验种群历史是否发生扩张的参数,统计检验显著的负数D值可能是选择作用、群体扩张和瓶颈效应等原因造成的[21]。Fu′s Fs检验的方法主要是基于中性进化的前提下样本中出现所观测到的等位基因(单倍型)数目的概率。Fs检验对群体扩张非常敏感,群体扩张通常产生绝对值较大的负数Fs值[22]。合并褐石斑鱼3个地理群体的数据进行中性检验发现,整个群体的D值为负值,Fs值为较大的负值(-4.195 6),并且统计检验结果显著(P < 0.05)。Tajima′s D和Fu′s Fs的检验结果也证实西太平洋海区褐石斑鱼群体存在扩张的历史特征。Chen等[36]研究表明,赤点石斑鱼(E.akaara)的群体扩张历史动态是由更新世冰期海平面下降造成大部分栖息地消失,导致其种群避难于东海和南海盆地产生的。与赤点石斑鱼类似,褐石斑鱼大部分现有栖息地位于大陆架。伴随每次冰盛期(Glacial maximum),海平面下降120-140 m[37]。褐石斑鱼栖息地也会因为海平面的下降而逐渐形成陆地,导致其种群避难于海洋盆地,待海平面上升后重新进入现有栖息地,分布区迅速扩大,呈现种群扩张的历史动态。本研究结果表明,褐石斑鱼不仅整体遗传多样性水平较低,而且容易受到气候变化导致种群数量波动,在全球气候变化的背景下,加强对褐石斑鱼种质资源的保护尤为关键。
4 结论本研究开展了褐石斑鱼西太平洋海区的中国海南岛(HN)、福建厦门(XM)和韩国济州岛(HG) 3个地理群体的遗传多样性和遗传结构研究。结果显示:各地理群体总体遗传多样性水平较低,韩国群体遗传多样性水平高于中国海南和厦门群体。褐石斑鱼群体遗传变异主要来源于群体内,群体间存在中度遗传分化。距离隔离是褐石斑鱼群体遗传分化的主要因素。总之,褐石斑鱼总体遗传多样性水平较低,其种群存在扩张的历史特征,易受到渔业捕捞和气候变化的影响导致其种群衰退,因此急需加强褐石斑鱼野生种群的监测和保护。
| [1] |
HEEMSTRA P C, RANDALL J E. Groupers of the world[M]. Rome: Food and Agriculture Organization of the United Nations, 1993: 382.
|
| [2] |
王波, 孙丕喜, 张朝晖, 等. 褐石斑鱼的生物学特性及室内养殖初步试验[J]. 渔业现代化, 2006(1): 28-29. |
| [3] |
SAWADA Y, KATO K, OKADA T, et al. Growth and morphological development of larval and juvenile Epinephelus bruneus (Perciformes: Serranidae)[J]. Ichthyological Research, 1999, 46(3): 245-257. DOI:10.1007/BF02678510 |
| [4] |
吴玉萍, 田永胜, 成美玲, 等. 鞍带石斑鱼(♂)与褐石斑鱼(♀)、云纹石斑鱼(♀)杂交后代变态发育和生长比较[J]. 渔业科学进展, 2020, 41(4): 23-32. |
| [5] |
TO A, AMORIM P, CHOAT J H, et al. Epinephelus bruneus[EB/OL]. [2022-02-05]. http://dx.doi.org/10.2305/IUCN.UK.2018-2.RLTS.T135381188A100573599.en.
|
| [6] |
SONG Y B, OH S R, SEO J P, et al. Larval development and rearing of longtooth grouper Epinephelus bruneus in Jeju Island, Korea[J]. Journal of the World Aquaculture Society, 2005, 36(2): 209-216. |
| [7] |
GUO M L, SU Y Q, ZHANG Z W, et al. Differentiation of Epinephelus moara from E.bruneus by improved nest-tetra-primer-specific PCR[J]. Progress in Natural Science, 2009, 19(10): 1221-1226. DOI:10.1016/j.pnsc.2009.04.003 |
| [8] |
WATARI T, NAKAMURA Y, KOTCHAROEN W, et al. Application of down-flow hanging sponge: upflow sludge blanket system for nitrogen removal in Epinephelus bruneus closed recirculating aquaculture system[J]. Aquaculture, 2021, 532: 735997. DOI:10.1016/j.aquaculture.2020.735997 |
| [9] |
郭新红, 刘少军, 刘巧, 等. 鱼类线粒体DNA研究新进展[J]. 遗传学报, 2004, 31(9): 983-1000. |
| [10] |
高天翔, 毕潇潇, 赵林林, 等. 基于线粒体Cytb基因全序列的松江鲈群体遗传结构分析[J]. 水生生物学报, 2013, 37(2): 199-207. |
| [11] |
郭明兰, 苏永全, 陈晓峰, 等. 云纹石斑鱼与褐石斑鱼形态比较研究[J]. 海洋学报(中文版), 2008, 30(6): 106-114. |
| [12] |
LIU M, LI J L, DING S X, et al. Epinephelus moara: a valid species of the family Epinephelidae (Pisces: Perciformes)[J]. Journal of Fish Biology, 2013, 82: 1684-1699. DOI:10.1111/jfb.12112 |
| [13] |
MANIATIS T, FRITSCH E F, SAMBROOK J. Molecular cloning: a laboratory manual[M]. 2nd ed. Cold Spring Harbor, New York: Spring Harbor Laboratory Press, 1989.
|
| [14] |
ROZAS J, SÁNCHEZ-DELBARRIO J C, MESSEGUER X, et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics, 2003, 19(18): 2496-2497. DOI:10.1093/bioinformatics/btg359 |
| [15] |
EXCOFFIER L, LISCHER H E L. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10(3): 564-567. DOI:10.1111/j.1755-0998.2010.02847.x |
| [16] |
POSADA D. jModelTest: phylogenetic model averaging[J]. Molecular Biology and Evolution, 2008, 25(7): 1253-1256. DOI:10.1093/molbev/msn083 |
| [17] |
WILGENBUSCH J C, SWOFFORD D. Inferring evolutionary trees with PAUP[J]. Current Protocols in Bioinformatics, 2003(1): 4-6. |
| [18] |
EXCOFFIER L, SMOUSE P E, QUATTRO J M. Analysis of molecular variance inferred from metricdistances among DNA haplotypes: application to human mitochondrial DNA restriction data[J]. Genetics, 1992, 131: 479-491. DOI:10.1093/genetics/131.2.479 |
| [19] |
JENSEN J L, BOHONAK A J, KELLEY S T. Isolation by distance, web service[J]. BMC Genetics, 2005, 6: 13. |
| [20] |
SLATKIN M. Isolation by distance in equilibrium and non-equilibrium populations[J]. Evolution, 1993, 47(1): 264-279. DOI:10.2307/2410134 |
| [21] |
TAJIMA F. Statistical-method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123: 585-595. DOI:10.1093/genetics/123.3.585 |
| [22] |
FU Y X. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection[J]. Genetics, 1997, 147: 915-925. DOI:10.1093/genetics/147.2.915 |
| [23] |
ZHUANG X, DING S, WANG J, et al. A set of 16 consensus primer pairs amplifying the complete mitochondrial genomes of orange-spotted grouper (Epinephelus coioides) and Hong Kong grouper (Epinephelus akaara)[J]. Molecular Ecology Resources, 2009, 9(6): 1551-1553. DOI:10.1111/j.1755-0998.2009.02716.x |
| [24] |
余科, 安苗, 黄胜, 等. 基于Cytb和D-Loop基因对中国少鳞鳜种群遗传多样性的分析[J]. 水产科学, 2020, 39(6): 894-901. DOI:10.16378/j.cnki.1003-1111.2020.06.014 |
| [25] |
GRANT W, BOWEN B W. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation[J]. The Journal of Heredity, 1998, 89(5): 415-426. DOI:10.1093/jhered/89.5.415 |
| [26] |
SIGURGÍSLASON H, ÁRNASON E. Extent of mitochondrial DNA sequence variation in Atlantic cod from the Faroe Islands: a resolution of gene genealogy[J]. Heredity, 2003, 91(6): 557-564. DOI:10.1038/sj.hdy.6800361 |
| [27] |
吴仁协, 柳淑芳, 庄志猛, 等. 基于线粒体Cyt b基因的黄海、东海小黄鱼(Larimichthys polyactis)群体遗传结构[J]. 自然科学进展, 2009, 19(9): 924-930. |
| [28] |
王登玉, 丁少雄, 郭丰, 等. 中国东南近海斜带石斑鱼群体的地理分化和遗传结构研究[J]. 高技术通讯, 2008, 18(3): 324-329. |
| [29] |
HASSAN N C, AZIZ A H A, ALI N M, et al. Genetic diversity in farmed tiger grouper broodstock inferred by partial cytochrome b gene[J]. International Journal of Chemical, Environmental & Biological Sciences, 2015, 3(1): 25-28. |
| [30] |
COPPINGER C R, JAMES N, GOUWS G, et al. Assessing the genetic diversity of catface grouper Epinephelus andersoni in the subtropical Western Indian Ocean[J]. Fisheries Research, 2019, 218: 186-197. DOI:10.1016/j.fishres.2019.05.008 |
| [31] |
BALLOUX F, LUGON-MOULIN N. The estimation of population differentiation with microsatellite markers[J]. Molecular Ecology, 2002, 11(2): 155-165. DOI:10.1046/j.0962-1083.2001.01436.x |
| [32] |
PALUMBI S R. Genetic divergence, reproductive isolation, and marine speciation[J]. Annual Review of Ecology and Systematics, 1994, 25: 547-572. DOI:10.1146/annurev.es.25.110194.002555 |
| [33] |
傅蒙娜, 王军, 丁少雄, 等. 基于线粒体DNA细胞色素b序列分析3个条纹斑竹鲨群体的遗传结构和遗传分化[J]. 热带海洋学报, 2010, 29(6): 86-91. |
| [34] |
RICHARDSON L R, GOLD J R. Mitochondrial DNA variation in an red grouper (Epinephelus morio) and greater amberjack (Seriola dumerili) from the Gulf of Mexico[J]. ICES Journal of Marine Science, 1993, 50: 53-62. |
| [35] |
GAITHER M R, JONES S A, KELLEY C, et al. High connectivity in the deepwater snapper Pristipomoides filamentosus (Lutjanidae) across the Indo-Pacific with isolation of the Hawaiian Archipelago[J]. PLoS One, 2011, 6(12): e28913. |
| [36] |
CHEN S P, LIU T, LI Z F, et al. Genetic population structuring and demographic history of red spotted grouper (Epinephelus akaara) in South and East China Sea[J]. African Journal of Biotechnology, 2008, 7(20): 3554-3562. |
| [37] |
LAMBECK K, ESAT T M, POTTER E K. Links between climate and sea levels for the past three million years[J]. Nature, 2002, 419: 199-206. |



