2. 云南大学生命科学学院, 云南昆明 650500;
3. 中国科学院昆明植物研究所, 云南省极小种群野生植物综合保护重点实验室, 云南昆明 650201;
4. 云南大学生态学与环境学院, 云南省植物繁殖适应与进化生态学重点实验室, 云南昆明 650500
2. School of Life Sciences, Yunnan University, Kunming, Yunnan, 650500, China;
3. Yunnan Key Laboratory for Integrative Conservation of Plant Species with Extremely Small Populations, Kunming Institute of Botany, Chinese Academy of Sciences, Kunming, Yunnan, 650201, China;
4. Yunnan Key Laboratory of Plant Reproductive Adaptation and Evolutionary Ecology, School of Ecology and Environmental Science, Yunnan University, Kunming, Yunnan, 650500, China
玄参科(Scrophulariaceae)醉鱼草属(Buddleja)植物多为灌木或小乔木,全球有90余种,分布于美洲、非洲和亚洲的热带至温带地区[1]。本属植物醉鱼草(B.lindleyana)叶片中含有醉鱼草甙和醉鱼草酮甙,对鱼有麻醉作用,古时渔人将其叶片捣烂放入水中,鱼取食后如同醉酒般浮于水面,故而得名“醉鱼草”[2, 3]。醉鱼草属植物具有较高的园艺价值,如大叶醉鱼草(B.davidii)被誉为“蝴蝶灌木”(Butterfly bush),引种到欧美等地区后成为了流行的观赏植物[4, 5]。该属一些类群还具有药食价值,被用作传统药物和食品添加剂[6]。醉鱼草属植物普遍存在多倍化现象[7, 8],亚洲地区多倍化比例高,且种内和种间倍性变化丰富(2n=2x,4x,6x,8x,12x,16x,24x),多倍化可能促进该属物种对不同环境的适应[7, 9]。醉鱼草属植物通常具有较高的生态适应性,部分类群为广布种和先锋种,国外甚至有园艺品种逃逸发展为入侵物种[4, 10]。但醉鱼草属也存在一些狭域分布的受威胁类群,世界自然保护联盟(IUCN)濒危物种红色名录共收录了6种醉鱼草属植物[11];《中国高等植物受威胁物种名录》将3种醉鱼草属植物列为易危(Vulneralbe,VU)[12];《云南省极小种群野生植物保护名录(2021版)》收录了2种醉鱼草属植物[13]。
无柄醉鱼草(B. sessilifolia)曾为大花醉鱼草(B.colvilei)的同物异名,后经形态学和分子生物学研究厘清了无柄醉鱼草为一个独立的物种[14]。野外调查发现,该物种仅零星分布于高黎贡山北麓,部分居群受自然和人为因素干扰严重,近年来成年植株数量急剧下降,随时面临灭绝风险,根据《IUCN物种红色名录濒危等级和标准》,该物种目前受威胁等级为濒危(EN),是典型的极小种群野生植物(Plant Species with Extremely Small Populations,PSESP),亟待开展保护生物学研究[13-16]。群体遗传学被认为是研究物种致濒原因的关键,因为遗传变异决定了种群适应变化的能力[17]。狭域分布的小种群物种容易受遗传漂变和近亲交配的影响,导致遗传多样性水平降低,进而使其对环境变化的适应力减弱,灭绝风险更高[17-19]。Hamrick等[20]总结了653篇涉及449个植物物种的等位酶,发现狭域分布的濒危物种遗传多样性水平普遍低于广泛分布的物种。但也有研究表明,部分狭域分布的物种仍具有较高的遗传多样性水平[21, 22]。可见植物濒危程度和遗传变异之间并未有普遍联系[19, 23],而未在统一标准下比较可能会导致遗传多样性水平和物种濒危程度不相关,如评估物种受威胁程度的标准(基于当地丰度、地理范围和生境特异性等)和物种的生活史属性。理想情况下,应选择具有类似进化历史、繁育系统、传粉者及散布策略的物种进行比较,通常系统发育关系密切的物种可以满足以上条件[21, 24]。
亚洲醉鱼草属植物系统发育研究表明,无柄醉鱼草与其他9个多倍体物种聚为一支,与大叶醉鱼草同为四倍体,且二者亲缘关系较近[8]。有趣的是,这一分支中无柄醉鱼草是分布最为狭窄的极小种群野生植物,而大叶醉鱼草是分布最为广泛的物种[4, 16]。大叶醉鱼草原产于我国中部、中西部地区,花序硕大艳丽,花色丰富,气味芳香,极具观赏性,自19世纪引入欧美地区后,被广泛应用于园林造景,是醉鱼草属开发最成功的观赏物种。但由于管理不善且物种本身适应性较强,大叶醉鱼草在引入地逃逸至野外大量繁殖造成入侵,在多个国家和地区被划定为入侵物种[4, 10, 25]。大叶醉鱼草在中国原产地分布海拔范围广(1 000-3 000 m),分布生境多为撂荒地、河流和道路两旁以及受干扰严重的贫瘠土地上,可作为先锋物种占领新生境。目前关于无柄醉鱼草和大叶醉鱼草遗传多样性方面的研究较少。因此,本研究基于简单重复序列(Simple Sequence Repeats, SSR)分子标记技术,采用多倍体遗传数据分析方法,比较无柄醉鱼草和大叶醉鱼草的遗传多样性和遗传结构,旨在探讨这两个物种各居群遗传多样性的分布格局和历史成因,并解析地理分布范围、濒危程度与遗传多样性的关系,提出针对濒危物种无柄醉鱼草的保护建议。
1 材料与方法 1.1 材料无柄醉鱼草126个样品来自5个野生居群(表 1),大叶醉鱼草206个样品来自14个野生居群(表 2),采样地理位置见图 1。样品采集时样株间隔10-15 m,避免采集到同一植株,采集植株新鲜枝条上完整健康、无病虫害的嫩叶,放入分子袋后加入硅胶干燥,带回实验室常温保存。
| 居群 Population |
位置 Location |
采样数量 Sample size |
经度 Longitude |
纬度 Latitude |
海拔/m Altitude/m |
| S | Gongshan, Sandui | 30 | 98°25′24″E | 27°42′55″N | 2 620 |
| D | Gongshan, Danzhu | 33 | 98°35′51″E | 27°37′56″N | 2 850 |
| M | Junction of Myanmar and Gongshan | 30 | 98°33′53″E | 27°35′31″N | 2 780 |
| F | Junction of Myanmar and Fugong | 14 | 98°39′59″E | 27°14′28″N | 3 060 |
| K | Gongshan, Dulongjiang Crossing | 19 | 98°29′52″E | 27°46′48″N | 3 140 |
| 居群 Population |
位置 Location |
采样数量 Sample size |
经度 Longitude |
纬度 Latitude |
海拔/m Altitude/m |
| ZT | Zhaotong | 8 | 104°08′58″E | 28°23′38″N | 1 134 |
| GG | Gongga | 29 | 101°58′54″E | 29°34′06″N | 3 166 |
| SZ | Shizhu | 15 | 108°09′53″E | 30°02′15″N | 629 |
| SNJ | Shennongjia | 12 | 110°28′29″E | 31°24′19″N | 835 |
| WDS | Wudangshan | 12 | 111°07′12″E | 32°30′24″N | 189 |
| WG | Wugang | 12 | 110°35′05″E | 26°42′36″N | 364 |
| GZ | Guzhang | 15 | 109°50′50″E | 28°31′21″N | 565 |
| ZJJ | Zhangjiajie | 12 | 110°26′35″E | 29°20′50″N | 879 |
| DJ | Dejiang | 12 | 108°11′47″E | 28°23′37″N | 760 |
| ZY | Zunyi | 12 | 106°41′26″E | 26°59′23″N | 1 345 |
| LH | Lianghe | 12 | 104°35′58″E | 25°48′46″N | 1 741 |
| TBS | Taibaishan | 12 | 107°48′51″E | 34°00′55″N | 2 710 |
| ES | Enshi | 34 | 109°33′10″E | 30°25′10″N | 647 |
| SQ | Shiqian | 9 | 108°28′42′′E | 27°35′08′′N | 882 |
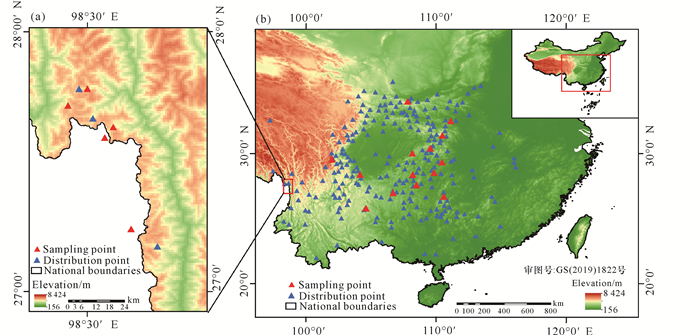
|
| 图 1 无柄醉鱼草(a)和大叶醉鱼草(b)采样点地理分布 Fig. 1 Geographic distribution of sampling point of B.sessilifolia (a) and B.davidii (b) |
1.2 方法 1.2.1 DNA提取及PCR扩增分析
采用改良的十六烷基三甲基溴化铵(Cetyltrimethylammonium Bromide,CTAB)法提取所有样品的总DNA,提取的DNA使用1%琼脂糖凝胶电泳和紫外分光光度计(Nandrop-1000)检测质量和浓度,检测合格的样品置于-20 ℃冰箱保存备用。无柄醉鱼草选用的引物来源于葛佳[16]筛选的10对SSR序列(表 3),大叶醉鱼草选用Schreiter等[25]筛选的8对SSR序列(表 4)。将确定好的两个物种引物序列提交给生工生物工程(上海)股份有限公司合成荧光引物,以提取后的样品DNA为模板,利用选取的引物序列进行PCR扩增。
| 引物编号 Primer number |
引物序列 Primer sequences |
重复单元 Repeat motif |
产物长度/bp Size range/bp |
| BS01 | F: CATTCCAACCGCGCAGAATC R: CGCGGAAGTCGTTCTTGTTG |
(AGC)5 | 136 |
| BS02 | F: GTTGTTGTCGGCGGATTGAG R: CATTCCAACCGCGCAGAATC |
(CTG)5 | 143 |
| BS03 | F: TCCTTGTCAGCTTGCTCCTG R: TGAAGAAGAGCGTGCTCAGG |
(CTT)5 | 103 |
| BS04 | F: CGTGCTCGTGCTTCTTCTTG R: ATCAAGGACAAGAGCCACGG |
(CTT)6 | 103 |
| BS05 | F: TCCAACGACTAGGAATCGCG R: GTGACGGAGCAGACGGAATC |
(GAA)5 | 115 |
| BS06 | F: AACTCCGATAAGCACACGGC R: TCATCAACTCGCTGCCGTAC |
(GCT)5 | 100 |
| BS07 | F: ATGACGACTACGACGATGGC R: CCTCTGCCGTACTCCTCATC |
(GGA)5 | 115 |
| BS08 | F: ATGGCGGCTATGGCTATGAC R: CGCGGAAGTCGTTCTTGTTG |
(TCC)5 | 106 |
| BS09 | F: GGAAGGAAGCTCCGGAAGAG R: GACGTCGTACTAGCAGACGC |
(CCG)6 | 101 |
| BS10 | F: AACTGCCAACAACAATCCGG R: AGTCGCAGTCGCAGATGTC |
(AAC)5 | 111 |
| 引物编号 Primer number |
引物序列 Primer sequences |
重复单元 Repeat motif |
产物长度/bp Size range/bp |
| Bud04 | F:GCTATTCATGGTAATTGAGTGAGG R:ATTGACGCCTCCTCTACCTG |
(GATA)7 | 145 |
| Bud09 | F: GCTCAACTGTCAGTACGTTGC R: CTCCTGCACTTCAGATTGTTTAC |
(CT)21 | 164 |
| Bud10 | F: TCCCTCTCATATTGGGATAACA R: GCATTTGGAACCGTTAAAGC |
(CT)26 T(CT)4 | 183 |
| Bud12 | F: ACATCCCTACCCGTGATAGTAG R: TTTTCGCTGTTTGTCCACTTAC |
(GA)19 | 143 |
| Bud03 | F: GCATGCGCTGACATTTTTC R: GTCTTCTCGACCCATGTGC |
(TATC)8(TA)5 | 111 |
| Bud06 | F: CGTCACATGTCGTTCGTAGG R: TTCCGTTATTCCCATTGTCC |
(CT)20CATA(CA)6 | 199 |
| Bud13 | F: CCTAACTGCGAATTGTATAGTTTCC R: TCTGATGCAGTCAGGTTTGC |
(CT)14 | 110 |
| Bud14 | F: CAAAACCAATGCCCAAAGAG R: AGCTTAGGAGTCCCCCACAC |
(GA)19 | 181 |
PCR扩增反应体系总体积25 μL,包括DNA模板1.0 μL、正向和反向引物各0.5 μL、10×PCR Buffer (含2.0 μL MgCl2) 2.5 μL、10 mmol·L-1 dNTP溶液0.5 μL、5 U·μL-1 Taq聚合酶0.2 μL,用无菌水补足至25 μL。PCR反应扩增程序:95 ℃预变性3 min;之后分两个阶段,第一阶段95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸30 s,10次循环;第二阶段95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,20次循环;最后于72 ℃修复延伸6 min。扩增后的PCR产物使用ABI PRISM 3730 XL(美国ABI公司)进行毛细管电泳测定片段长度。电泳结果通过软件GeneMarker 2.2.0读取每个扩增片段的长度,记录等位基因用于后续分析。
1.2.2 数据分析由于本研究的两个物种均为四倍体,多倍体的遗传变异具有多方面的复杂性,多个等位基因和复杂的分离模式会造成遗传数据分析结果产生偏差,选择适合的多倍体遗传数据分析方法可以避免或减少多倍化带来的偏差[26]。
本研究采用多倍体分析软件GenoDive 3.06[27]和Polygene 1.6[28]对两个物种的遗传数据进行分析,这两个软件同时考虑了基因型歧义和双倍减少,降低了多倍化可能造成的误差。利用GenoDive 3.06和Polygene 1.6软件计算两个物种各居群等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、多态信息指数(PIC)等遗传多样性参数;通过Polygene 1.6软件进行分子方差分析(Analysis of Molecular Variance, AMOVA),并计算居群间遗传分化系数(FST);基于Nei′s遗传距离进行主成分分析(PCoA);对各居群遗传关系构建非加权组平均法(Unweighted Pair Group Mean Analysis, UPGMA)聚类树,反映居群间遗传关系;并构建地理距离和遗传距离矩阵,采用R语言中的ade4包进行Mantel检验,分析群体遗传距离与地理距离的相关性。
通过软件Structure 2.3.4对来自不同居群的个体进行遗传结构分析,Structure分析适用于多倍体,当参数recessiveallelles被设置为1时,计算将忽略输入文件中所有等位基因的剂量信息,减少多倍体数据带来的误差[26]。依据居群数量设置运行参数,设置无柄醉鱼草K值为1-5,大叶醉鱼草K值为1-14,每个K值重复运算20次,马尔科夫链蒙特卡罗(MCMC)重复200 000次后进行100 000次burn-in。利用在线软件Structure Harvester(https://taylor0.biology.ucla.edu/struct_harvest/)根据最大似然值△K的最大lnP(D)值确定最佳聚类分组[29],然后通过在线软件Pophelper 2.3.1(http://pophelper.com/)绘制群体结构分析图[30]。
2 结果与分析 2.1 遗传多样性分析各位点遗传多样性指数见表 5。无柄醉鱼草在10个位点上共检测到102个等位基因,大叶醉鱼草在8个位点上共检测到109个等位基因。无柄醉鱼在各位点上观测杂合度、期望杂合度、Shannon′s多样性指数分别为0.503-0.696、0.518-0.780、0.889-1.751,平均值分别为0.621、0.663、1.332;大叶醉鱼草分别为0.730-0.993、0.761-0.893、1.485-2.375,平均值分别为0.820、0.824、1.983。无柄醉鱼草10个位点中,高度多态性位点(PIC>0.50)有8个,中度多态性位点(0.25<PIC<0.50)有2个;大叶醉鱼草8个位点均为高度多态性位点,表明这两个物种的引物都呈现出较高的多态性,能提供可靠的遗传信息。
| 物种 Species |
位点 Locus |
等位基因数 Na |
有效等位基因数 Ne |
观测杂合度 Ho |
期望杂合度 He |
Shannon′s 多样性指数I |
多态信息指数 PIC |
| B.sessilifolia | BS01 | 13 | 3.3 | 0.658 | 0.693 | 1.590 | 0.657 |
| BS02 | 9 | 2.1 | 0.547 | 0.518 | 0.889 | 0.425 | |
| BS03 | 6 | 3.7 | 0.689 | 0.730 | 1.410 | 0.686 | |
| BS04 | 12 | 3.6 | 0.696 | 0.725 | 1.515 | 0.678 | |
| BS05 | 9 | 3.8 | 0.683 | 0.738 | 1.548 | 0.698 | |
| BS06 | 7 | 2.8 | 0.585 | 0.640 | 1.205 | 0.584 | |
| BS07 | 12 | 2.4 | 0.546 | 0.583 | 1.084 | 0.508 | |
| BS08 | 16 | 2.3 | 0.503 | 0.564 | 1.055 | 0.472 | |
| BS09 | 10 | 4.5 | 0.647 | 0.780 | 1.751 | 0.755 | |
| BS10 | 8 | 2.9 | 0.661 | 0.655 | 1.276 | 0.597 | |
| Mean (S.E.) | 10.200(0.914) | 3.140(0.231) | 0.621(0.021) | 0.663(0.026) | 1.332(0.083) | 0.606(0.033) | |
| B.davidii | Bud04 | 17 | 6.4 | 0.783 | 0.844 | 2.137 | 0.828 |
| Bud09 | 12 | 4.7 | 0.735 | 0.788 | 1.720 | 0.757 | |
| Bud10 | 13 | 7.2 | 0.803 | 0.861 | 2.181 | 0.847 | |
| Bud12 | 21 | 7.4 | 0.886 | 0.865 | 2.238 | 0.851 | |
| Bud03 | 14 | 5.3 | 0.758 | 0.811 | 1.934 | 0.787 | |
| Bud06 | 10 | 4.3 | 0.730 | 0.769 | 1.798 | 0.747 | |
| Bud13 | 15 | 9.3 | 0.874 | 0.893 | 2.375 | 0.883 | |
| Bud14 | 7 | 4.2 | 0.993 | 0.761 | 1.485 | 0.718 | |
| Mean (S.E.) | 13.600(1.414) | 6.100(0.595) | 0.820(0.030) | 0.824(0.016) | 1.983(0.100) | 0.802(0.019) |
无柄醉鱼草5个居群和大叶醉鱼草14个居群的遗传多样性指数见表 6。无柄醉鱼草观测杂合度、期望杂合度、Shannon′s多样性指数分别为0.578(K)-0.654(D)、0.564(K)-0.648(S)、0.991(K)-1.258(S),平均值分别为0.614、0.606、1.162;大叶醉鱼草分别为0.795(WDS)-0.847(ZJJ)、0.749(SQ)-0.798(ZJJ)、1.612(SNJ)-1.800(LH),平均值分别为0.819、0.775、1.729。
| 物种 Species |
居群 Population |
等位基因数 Na |
有效等位基因数 Ne |
私有等位基因数 Np |
观测杂合度 Ho |
期望杂合度 He |
Shannon′s 多样性指数I |
多态信息指数 PIC |
近交系数 Fis |
| B.sessilifolia | D | 6.7 | 3.1 | 0.8 | 0.654 | 0.645 | 1.245 | 0.584 | 0.085 |
| M | 7.1 | 2.9 | 1.3 | 0.603 | 0.605 | 1.211 | 0.555 | 0.309 | |
| S | 6.8 | 3.2 | 1.0 | 0.652 | 0.648 | 1.258 | 0.586 | 0.148 | |
| F | 5.4 | 2.8 | 0.2 | 0.582 | 0.571 | 1.104 | 0.522 | 0.099 | |
| K | 4.3 | 2.6 | 0.2 | 0.578 | 0.564 | 0.991 | 0.496 | 0.198 | |
| Mean (S.E.) | 6.060 (0.472) | 2.900 (0.095) | 0.700 (0.196) | 0.614 (0.015) | 0.606 (0.016) | 1.162 (0.045) | 0.548 (0.016) | 0.168 (0.036) | |
| B.davidii | ZT | 7.6 | 4.7 | 0.0 | 0.833 | 0.766 | 1.661 | 0.733 | -0.089 |
| GG | 8.9 | 5.1 | 0.1 | 0.843 | 0.785 | 1.725 | 0.751 | -0.078 | |
| DJ | 9.3 | 4.8 | 0.3 | 0.804 | 0.769 | 1.757 | 0.742 | -0.047 | |
| SZ | 7.5 | 4.7 | 0.0 | 0.831 | 0.766 | 1.640 | 0.729 | -0.089 | |
| ES | 9.5 | 4.8 | 0.1 | 0.816 | 0.774 | 1.745 | 0.745 | -0.053 | |
| GZ | 8.4 | 5.2 | 0.0 | 0.829 | 0.788 | 1.776 | 0.761 | -0.054 | |
| SNJ | 7.5 | 4.5 | 0.0 | 0.814 | 0.755 | 1.612 | 0.717 | -0.083 | |
| WDS | 8.5 | 4.6 | 0.3 | 0.795 | 0.757 | 1.694 | 0.728 | -0.050 | |
| WG | 7.5 | 4.9 | 0.0 | 0.804 | 0.769 | 1.656 | 0.733 | -0.047 | |
| ZJJ | 8.1 | 5.2 | 0.0 | 0.847 | 0.798 | 1.792 | 0.771 | -0.062 | |
| ZY | 8.8 | 4.9 | 0.0 | 0.804 | 0.765 | 1.742 | 0.739 | -0.052 | |
| LH | 8.9 | 5.1 | 0.1 | 0.820 | 0.788 | 1.800 | 0.760 | -0.043 | |
| TBS | 8.8 | 4.6 | 0.9 | 0.814 | 0.769 | 1.713 | 0.739 | -0.059 | |
| SQ | 8.0 | 4.7 | 0.4 | 0.806 | 0.749 | 1.637 | 0.715 | -0.078 | |
| Mean (S.E.) | 8.720 (0.177) | 4.930 (0.060) | 0.200 (0.065) | 0.819 (0.004) | 0.775 (0.004) | 1.729 (0.016) | 0.744 (0.004) | -0.059 (0.004) |
综上,无柄醉鱼草和大叶醉鱼草在居群水平上具有较高的遗传多样性,且大叶醉鱼草高于无柄醉鱼草。两个物种各居群遗传参数变化不大,遗传多样性水平相似。无柄醉鱼草所有居群近交系数均大于零(表 6),群体中存在杂合子缺失现象,近交程度较高; 相反大叶醉鱼草所有居群近交系数均小于零(表 6),群体中杂合子过剩,异交程度较高。
2.2 遗传结构分析 2.2.1 分子方差分析通过分子方差分析研究两个物种的遗传变异情况,结果见表 7。无柄醉鱼草5个居群中95.684%的遗传变异来自居群内部,4.316%的遗传变异来自居群间,居群间遗传分化系数(FST)为0.043;大叶醉鱼草14个居群中97.639%的遗传变异来自居群内部,2.362%的遗传变异来自居群间,居群间遗传分化系数为0.024。以上结果表明两个物种绝大部分遗传变异存在于居群内部,居群间遗传分化程度极低,但无柄醉鱼草居群间遗传变异程度略高于大叶醉鱼草。
| 物种 Species |
变异来源 Source of variation |
自由度 df |
方差和 SS |
变异分组 Variation grouping |
占变异百分比/% Percentage of variation/% |
遗传分化系数 FST |
| B.sessilifolia | Among populations | 4 | 71.69 | 0.015 | 4.316 | 0.043 |
| Within populations | 125 | 1 547.33 | 0.329 | 95.684 | ||
| B.davidii | Among populations | 13 | 130.30 | 0.010 | 2.362 | 0.024 |
| Within populations | 205 | 2 648.21 | 0.415 | 97.639 |
2.2.2 Mantel检验
对无柄醉鱼草和大叶醉鱼草居群进行Mantel检验,检测地理距离与遗传距离之间的相关性,分析结果见图 2。结果显示,无柄醉鱼草P=0.875>0.05,表明无柄醉鱼草各居群间的遗传距离与地理距离不具有相关性,遗传分化不遵循距离隔离模式;大叶醉鱼草P=0.000 1<0.05,表明各居群间的遗传距离与地理距离间存在相关性,地理距离对大叶醉鱼草各居群间的遗传分化具有较强的塑造作用。
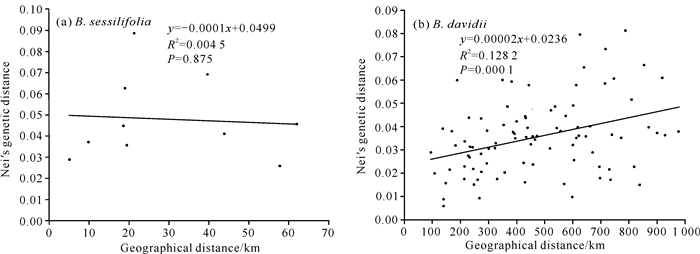
|
| 图 2 无柄醉鱼草和大叶醉鱼草遗传距离与地理距离相关性检验 Fig. 2 Correlation test of B.sessilifolia and B.davidii between genetic distance and geographical distance |
2.2.3 UPGMA聚类树
基于Nei′s遗传距离对无柄醉鱼草的5个居群和大叶醉鱼草的14个居群构建UPGMA聚类树进行聚类分析,结果如图 3所示。无柄醉鱼草被分为2支,居群D和M聚为一支(Clade Ⅰ),居群S、F和K聚为另一支(Clade Ⅱ);大叶醉鱼草的14个居群可分为3支,居群ZT、GG、SZ、ES、SNJ、SQ聚为一支(Clade Ⅰ),分支中居群SQ单独为一小支,与其他居群遗传距离相对较远;居群DJ、GZ、ZJJ、LH、WG、ZY、WDS为一支(Clade Ⅱ),分支中居群WDS单独为一小支,与其他居群遗传距离相对较远;剩余的居群TBS单独为一支(Clade Ⅲ)。
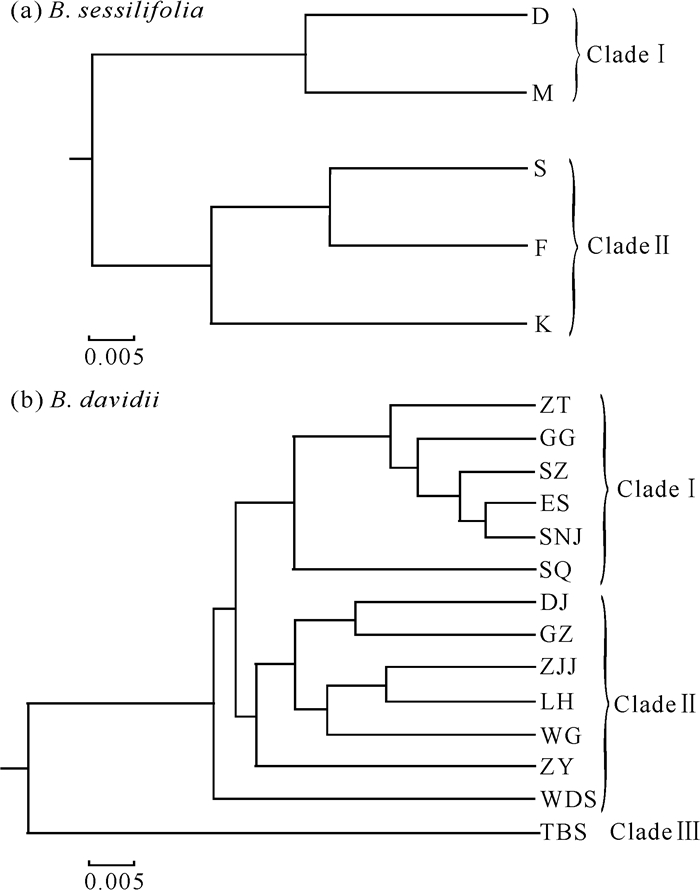
|
| 图 3 无柄醉鱼草和大叶醉鱼草UPGMA聚类树分析 Fig. 3 UPGMA clustering tree analysis of B.sessilifolia and B.davidii |
2.2.4 主坐标分析
通过主坐标分析两个物种的群体间遗传关系,分析结果见图 4。无柄醉鱼草126个个体中,主坐标1和主坐标2的贡献率分别为15.35%和24.74%;大叶醉鱼草206个个体中,主坐标1和主坐标2的贡献率分别为9.18%和15.58%。两个物种的PCoA结果与UPGMA聚类树分支基本一致。
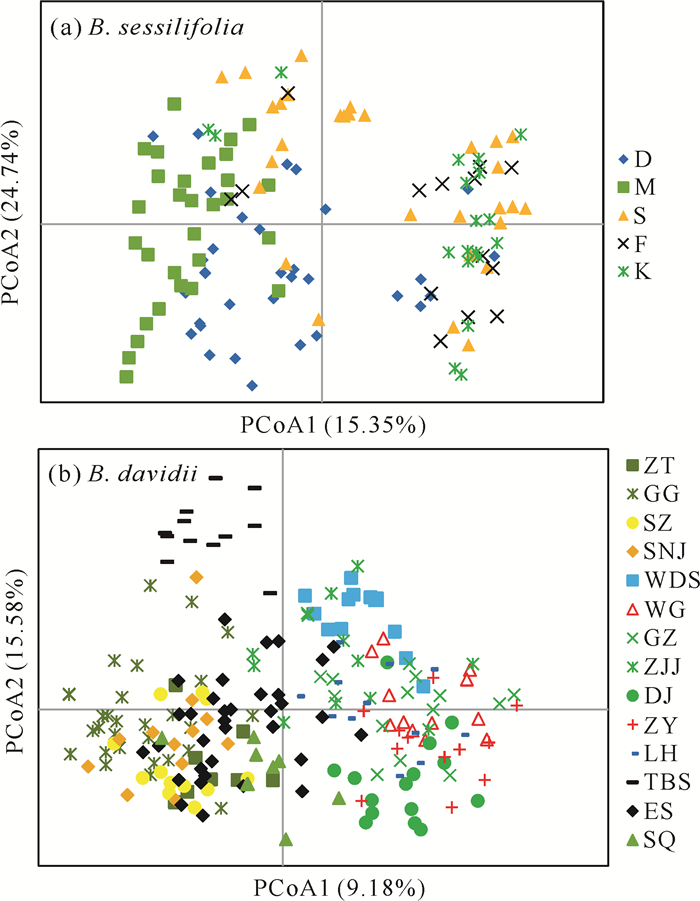
|
| 图 4 无柄醉鱼草和大叶醉鱼草主坐标分析 Fig. 4 PCoA of B.sessilifolia and B.davidii |
2.2.5 Structure聚类分析
采用Structure分析无柄醉鱼草5个居群与大叶醉鱼草14个居群的遗传结构,结果如图 5所示。根据Evanno等[31]的评判标准,即通过△K来确定最佳K值。无柄醉鱼草居群的最佳K值为2,大叶醉鱼草的最佳K值为3。无柄醉鱼草5个居群被归为2个起源类群(基因库),居群D和M组成一个分组,居群F和K组成另一个分组,居群S位于中间,混杂了两个基因库组成的个体。大叶醉鱼草14个居群被归为3个起源类群(基因库),居群ZT、GG、SZ、SNJ、ES、SQ被分为一组,居群DJ、WG、ZY、LH、GZ、ZJJ被分为另外一组,居群WDS、TBS为一个分组,每个居群都存在3种基因库不同程度的相互渗透。两个物种的Structure分析结果与UPGMA聚类分析、PCoA结果基本一致,相互印证。
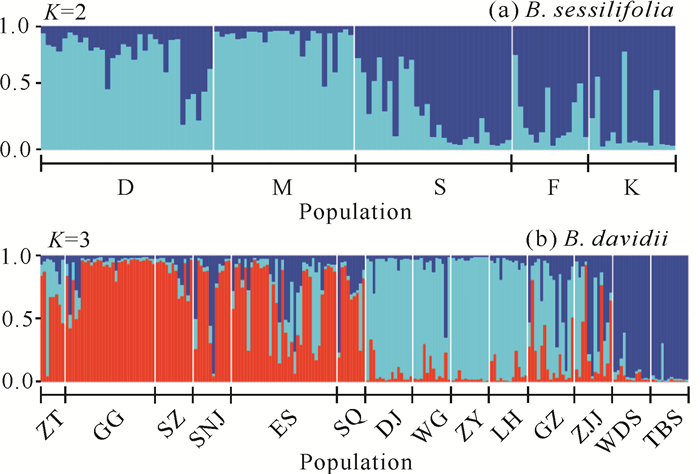
|
| 图 5 无柄醉鱼草和大叶醉鱼草Structure分析 Fig. 5 Structure analysis of B.sessilifolia and B.davidii |
3 讨论 3.1 无柄醉鱼草与大叶醉鱼草的遗传多样性
遗传多样性是物种进化潜力和适应环境变化能力的内在体现,比较分布狭窄的濒危物种与分布广泛的近缘种的遗传多样性,有助于了解濒危物种在演化历史和关键生活史中可能面临的挑战,以及其适应性和生存能力的变化。这对于揭示濒危物种的遗传特征、演化历史有重要意义,并可为制定濒危物种保护策略提供理论依据[21, 24]。遗传多样性参数He和I是衡量遗传多样性水平的重要指标。与醉鱼草属二倍体物种如互叶醉鱼草(B.alternifolia,He=0.380、I=0.691)、皱叶醉鱼草(B.crispa,He=0.551、I=1.085)和互对醉鱼草(B.wardii,He=0.513、I=0.951)相比[32],本研究中无柄醉鱼草(He=0.606, I=1.162)和大叶醉鱼草(He=0.775, I=1.729)的遗传多样性水平更高。与Nybom[23]报道的通过SSR分子标记评估的地区特有种(He=0.42)和狭域分布种(He=0.56)相比,本研究中无柄醉鱼草遗传多样性水平更高,接近区域分布种(He=0.65)和广泛分布种(He=0.62)的平均水平,而广布种大叶醉鱼草的遗传多样性平均水平(He=0.78)高于广泛分布种(He=0.62)。本研究中两个物种都维持了较高的遗传多样性水平,可能与其多倍体属性有关,多倍体中基因数量整倍增加,具有更多的基因副本,这意味着在进化过程中可以积累更多的突变和遗传变异,相较于二倍体,近缘物种通常表现出更高水平的杂合度[33, 34]。高水平的遗传变异赋予了物种更高的灵活性,也缓冲了居群因遗传漂变造成的遗传侵蚀[35]。
本研究中两个醉鱼草属物种都维持了较高的遗传多样性水平,但广布种大叶醉鱼草在物种水平和居群水平的遗传多样性均高于狭域种无柄醉鱼草,这一结果验证了广泛分布的物种比狭域分布的近缘种具有更高水平的遗传多样性的普遍观点[20]。物种的遗传多样性受到多种内外因素的影响,理论上认为小种群、分布范围狭窄的物种中,遗传漂变会成为主要的进化力量,遗传漂变可能会造成一些等位基因的丢失或固定,导致居群内遗传多样性减少和杂合性降低;而有害基因的固定则加剧了居群的脆弱性,进而降低物种适应性和遗传多样性水平[18]。近年来由于多种因素干扰,无柄醉鱼草多个居群植株数量持续减少,可能会导致其遗传多样性进一步丧失,从而降低该物种适应环境变化的潜力,加剧灭绝风险。因此,为实现该物种长期生存的目标,需尽快采取有效的保护措施。广布种的生存环境差异大、适应性幅度更广、居群间基因交流频繁、种群间分化小,能更好地维持遗传变异,也更可能产生新变异[21],这可能是无柄醉鱼草遗传多样性水平低于大叶醉鱼草的原因之一。物种的遗传多样性水平不仅受地理分布范围和居群大小等因素影响,还与物种的交配系统特征有关[36]。通常认为异交物种比自交物种具有更高的遗传多样性,自交会增加居群内的纯合性,造成遗传变异丧失,遗传多样性锐减;近交会导致后代适应能力下降,即表现为近交衰退,且小种群会加剧近亲繁殖[37]。传粉生物学研究表明,无柄醉鱼草的交配系统是以异交为主的兼性自交[16],大叶醉鱼草为异交[26]。本研究中无柄醉鱼草所有居群的近交系数均大于零,表明每个居群都存在自交或近交现象;而大叶醉鱼草所有居群的近交系数均小于零,表明异交程度较高,符合两个物种的交配系统特征。交配系统由异交向自交的转变会降低物种的遗传多样性水平[38],交配系统上的差异也可能是造成无柄醉鱼草遗传多样性水平低于大叶醉鱼草的原因之一。
3.2 无柄醉鱼草与大叶醉鱼草的遗传结构物种的遗传结构是遗传变异在群体内、群体间的分布以及在时间上的变化。本研究通过UPGMA聚类、PCoA和Structure分析对无柄醉鱼草和大叶醉鱼草进行遗传结构解析,3种方法得到的结果基本一致。
遗传分化指数(FST)通常用于评价群体间的遗传分化程度,当FST ≥0.25时,群体遗传分化程度极高;当0.15≤FST<0.25时,遗传分化程度高;当0.05≤FST<0.15时,遗传分化程度中等;当0<FST<0.05时,遗传分化程度低[39]。本研究中,无柄醉鱼草遗传分化指数为0.043,大叶醉鱼草为0.024,表明两个物种居群间遗传分化程度均较低。造成该现象的原因可能与两个物种的多倍体属性有关:遗传分化的主要因素——遗传突变和基因迁移在多倍体中的影响更弱,基因迁移会携带更多的基因拷贝数量,导致居群间的同质化程度更高,从而降低多倍体的FST值,使遗传分化程度低于二倍体[26, 40]。本研究还发现狭域种无柄醉鱼草相对广布种大叶醉鱼草具有较高的分化程度。物种的遗传结构受到基因流、突变、选择和遗传漂变等多种因素的共同影响,其中基因流起重要作用[41],植物居群内和居群间的基因流主要依靠花粉扩散和种子传播,无柄醉鱼草交配系统为以异交为主的兼性自交,大叶醉鱼草交配系统为异交。自交在一定程度上限制了种群间的花粉流动,导致自交物种的遗传分化大于异交物种[20, 23]。此外,无柄醉鱼草由于分布区狭窄,生境破碎化以及分布区高海拔多雨的气候条件造成了传粉者限制,其主要传粉昆虫白背熊峰(Bombus festivus)的访问多发生于同株异花之间(占比98%)[16],跨居群间基因流较弱;大叶醉鱼草的传粉者更为丰富,多为鳞翅目(Lepidoptera)且能长距离飞行的蝶类[42, 43],居群间基因交流相对更强。同时,无柄醉鱼草存在种子取食者,果实被蛾类幼虫寄生严重,被寄生的果实种子减产近一半,寄生还改变了果实裂开的拓扑结构,影响残存种子的传播[16];大叶醉鱼草单株花序量大,果实众多,种子结实量巨大,尘埃型种子借助风和水流传播,种子具长翅,更易于远距离传播,且具有强适应性[42]。综上,无柄醉鱼草花粉流和种子流受到多重限制,这可能是导致无柄醉鱼草居群间遗传分化程度高于大叶醉鱼草的原因之一。
3.3 无柄醉鱼草保护建议本研究结果表明濒危物种无柄醉鱼草野外居群数量稀少,遗传多样性水平低于广布种大叶醉鱼草,居群间分化程度较低。无柄醉鱼草5个居群中,居群D、M、S遗传多样性较高且是现存植株数量较多的居群,拥有丰富的特有等位基因,遗传结构分析中居群M和D被分为一组,且距居群S较近,这3个居群遗传差异较小,可作为一个遗传单元,建议将居群D、M、S划分为保护单元1;由于居群F和K地理距离相隔较远,建议各自划分为一个保护单元,即保护单元2、3。保护单元1中,居群M在缅甸境内,居群S较大且受干扰程度低,而居群D近年来因修路遭严重破坏,植株数量大幅下降,建议重点保护;保护单元2中居群K也因修路受到严重破坏,残余植株极少,建议重点保护。由于无柄醉鱼草遗传多样性水平相对较高,遗传变异主要存在于居群内部,并且该物种对生境要求较为严苛,对活体植株直接进行迁地保护存在较大困难,因此建议对该物种优先实施就地保护,加强对现有居群的保护。
无柄醉鱼草分布于我国的居群均位于自然保护区内,建议与保护区合作,划定保护范围,减少在划定范围内的人为活动干扰。对于居群中潜在可能受塌方、泥石流等自然灾害影响的植株,在现有分布点附近选择较为安全的相似生境建立近地保护点,尽快将植株移栽至安全区域。同时应加强宣传教育,提高当地居民对该物种的保护意识和关注度。该物种的迁地保护建议以种质资源(种子、花粉)收集和保存为主,考虑到无柄醉鱼草居群内个体间的遗传变异较高,收集应尽可能覆盖所有居群,对居群内的每个个体进行收集,保证最大程度地保存该物种居群间的遗传变异,选择遗传多样性高的居群(如居群D、M、S)收集种子,开展种子萌发、育苗组织培养以及扦插等人工扩繁相关技术研究,培育后代于原居群内开展回归,扩大现有居群的数量和规模。
4 结论本研究对无柄醉鱼草5个野外居群遗传多样性进行研究,与同倍性的近缘广布种大叶醉鱼草相比,无柄醉鱼草遗传多样性较低,且其生活史多个环节存在障碍,限制了其花粉流和种子流,进而导致无柄醉鱼草遗传分化程度高于大叶醉鱼草。而人为干扰和自然灾害导致无柄醉鱼草植株数量持续下降,可能会进一步引发遗传多样性丧失,从而加剧其灭绝风险,无柄醉鱼草的抢救性保护刻不容缓。建议将无柄醉鱼草5个居群划分为3个保护单元,重点对丹珠和独龙江隧道口居群开展就地保护等工作。考虑到无柄醉鱼草繁殖过程存在一定的限制,在今后的研究中应考虑如何通过遗传拯救重建该种的传粉网络,进而打破花粉流限制。另外,无柄醉鱼草遗传多样性水平接近广布种平均水平,不符合传统的狭窄分布的小种群物种遗传变异水平低的假设,鉴于多倍体植物遗传多样性水平普遍较高,而多数遗传多样性研究主要针对二倍体植物,因此今后在研究多倍体遗传多样性水平时建议与同倍性的物种进行比较。
致谢:
感谢中国科学院昆明植物研究所孙卫邦研究员对本研究的大力支持,感谢李迎春、陶丽丹、魏薇、李云猛、蔡杰、郭永杰、亚吉东(中国科学院昆明植物研究所)、徐昊(秦岭国家植物园)、公维昌(曲阜师范大学)、张鑫(云南林业职业技术学院)、肖永福(贡山县民政局)、熊云、和毅(高黎贡山国家级自然保护区贡山管护分局)等人协助样品采集,感谢杨丰懋、肖斯悦(中国科学院昆明植物研究所)对本研究分析方法的建议。
| [1] |
CHAU J H, O'LEARY N, SUN W B, et al. Phylogenetic relationships in tribe Buddlejeae (Scrophulariaceae) based on multiple nuclear and plastid markers[J]. Botanical Journal of the Linnean Society, 2017, 184(2): 137-166. DOI:10.1093/botlinnean/box018 |
| [2] |
HOUGHTON P J. Ethnopharmacology of some Buddleja species[J]. Journal of Ethnopharmacology, 1984, 11(3): 293-308. DOI:10.1016/0378-8741(84)90075-8 |
| [3] |
李秉滔. 中国植物志: 第61卷[M]. 北京: 科学出版社, 1992.
|
| [4] |
TALLENT-HALSELL N G, WATT M S. The invasive Buddleja davidii (butterfly bush)[J]. The Botanical Review, 2009, 75(3): 292-325. DOI:10.1007/s12229-009-9033-0 |
| [5] |
STUART D D. Buddlejas[M]. Portland: Timber Press, 2006.
|
| [6] |
张虎翼, 潘竞先, 陈雅妍. 醉鱼草属植物化学成分及生物活性研究进展[J]. 国外医药(植物药分册), 1995, 10(5): 195-201. |
| [7] |
CHEN G, SUN W B, SUN H. Ploidy variation in Buddleja L.(Buddlejaceae) in the Sino-Himalayan region and its biogeographical implications[J]. Botanical Journal of the Linnean Society, 2007, 154(3): 305-312. DOI:10.1111/j.1095-8339.2007.00650.x |
| [8] |
YANG F M, GE J, GUO Y J, et al. Deciphering complex reticulate evolution of Asian Buddleja (Scrophulariaceae): insights into the taxonomy and speciation of polyploid taxa in the Sino-Himalayan region[J]. Annals of Botany, 2023, 132(1): 15-28. DOI:10.1093/aob/mcad022 |
| [9] |
WU S D, WANG Y, WANG Z F, et al. Species divergence with gene flow and hybrid speciation on the Qinghai-Tibet Plateau[J]. New Phytologist, 2022, 234(2): 392-404. DOI:10.1111/nph.17956 |
| [10] |
GASPERINI C, CARRARI E, SELVI F. Invasion of riparian habitats by Buddleja davidii: a case study from the Northern Apennines[J]. Annali Di Botanica, 2020, 10: 21-32. |
| [11] |
IUCN. The IUCN red list of threatened species[Z/OL]. (2018-01-01)[2023-08-10]. https://www.iucnredlist.org/search?query=Buddleja&searchType=species.
|
| [12] |
覃海宁, 杨永, 董仕勇, 等. 中国高等植物受威胁物种名录[J]. 生物多样性, 2017, 25(7): 696-744. |
| [13] |
孙卫邦. 云南省极小种群野生植物保护名录: 2021版[M]. 昆明: 云南科技出版社, 2021.
|
| [14] |
GE J, CAI L, BI G Q, et al. Characterization of the complete chloroplast genomes of Buddleja colvilei and B. sessilifolia: implications for the taxonomy of Buddleja L[J]. Molecules, 2018, 23(6): 1248. DOI:10.3390/molecules23061248 |
| [15] |
云南林业和草原局. 云南省极小种群野生植物保护名录(2022年版)[Z/OL]. (2023-01-13)[2023-08-10]. https://lcj.yn.gov.cn/html/2023/zdlyxxgkqt_0113/67989.html.
|
| [16] |
葛佳. 无柄醉鱼草分类界定及保护生物学研究[D]. 北京: 中国科学院大学, 2018.
|
| [17] |
SUPPLE M A, SHAPIRO B. Conservation of biodiversity in the genomics era[J]. Genome Biology, 2018, 19(1): 131. DOI:10.1186/s13059-018-1520-3 |
| [18] |
RALLS K, BALLOU J D, DUDASH M R, et al. Call for a paradigm shift in the genetic management of fragmented populations[J]. Conservation Letters, 2018, 11(2). |
| [19] |
GITZENDANNER M A, SOLTIS P S. Patterns of genetic variation in rare and widespread plant congeners[J]. American Journal of Botany, 2000, 87(6): 783-792. DOI:10.2307/2656886 |
| [20] |
HAMRICK J L, GODT M J W. Allozyme diversity in plant species[M]//BROWN H D, CLEGG M T, KAHLER A L(eds). Plant population genetics, breeding, and genetic resources. Sunderland, Massachusetts: Sinauer Associates Inc, 1989.
|
| [21] |
GE S, WANG K Q, HONG D Y, et al. Comparisons of genetic diversity in the endangered Adenophora lobophylla and its widespread congener, A.potaninii[J]. Conservation Biology, 1999, 13(3): 509-513. DOI:10.1046/j.1523-1739.1999.98136.x |
| [22] |
SILVA G A R, KHAN G, RIBEIRO-SILVA S, et al. Extreme genetic structure in a relict cactus genus from campo rupestre landscapes: implications for conservation[J]. Biodiversity and Conservation, 2020, 29(4): 1263-1281. DOI:10.1007/s10531-020-01934-6 |
| [23] |
NYBOM H. Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants[J]. Molecular Ecology, 2004, 13(5): 1143-1155. DOI:10.1111/j.1365-294X.2004.02141.x |
| [24] |
HE T H, RAO G Y, YOU R L, et al. Genetic diversity of widespread Ophiopogon intermedius (Liliaceae s.l.): a comparison with endangered O.xylorrhizus[J]. Biological Conservation, 2000, 96(2): 253-257. DOI:10.1016/S0006-3207(00)00068-9 |
| [25] |
SCHREITER S, EBELING S K, DURKA W. Polymorphic microsatellite markers in the invasive shrub Buddleja davidii (Scrophulariaceae)[J]. American Journal of Botany, 2011, 98(2): e39-e40. |
| [26] |
CHEN G, SUN H, SUN W B, et al. Buddleja davidii and Buddleja yunnanensis: exploring features associated with commonness and rarity in Buddleja[J]. Flora-Morphology, Distribution, Functional Ecology of Plants, 2011, 206(10): 892-895. DOI:10.1016/j.flora.2011.05.006 |
| [27] |
MEIRMANS P G. Genodive version 3.0:easy-to-use software for the analysis of genetic data of diploids and polyploids[J]. Molecular Ecology Resources, 2020, 20(4): 1126-1131. DOI:10.1111/1755-0998.13145 |
| [28] |
HUANG K, DUNN D W, RITLAND K, et al. Polygene: population genetics analyses for autopolyploids based on allelic phenotypes[J]. Methods in Ecology and Evolution, 2020, 11(3): 448-456. DOI:10.1111/2041-210X.13338 |
| [29] |
EARL D A, VONHOLDT B M. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method[J]. Conservation Genetics Resources, 2012, 4(2): 359-361. DOI:10.1007/s12686-011-9548-7 |
| [30] |
FRANCIS R M. Pophelper: an R package and web app to analyse and visualize population structure[J]. Molecular Ecology Resources, 2017, 17(1): 27-32. DOI:10.1111/1755-0998.12509 |
| [31] |
EVANNO G, REGNAUT S, GOUDET J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study[J]. Molecular Ecology, 2005, 14(8): 2611-2620. DOI:10.1111/j.1365-294X.2005.02553.x |
| [32] |
廖荣丽. 互对醉鱼草的自然杂交起源与历史变迁研究[D]. 北京: 中国科学院大学(中国科学院昆明植物研究所), 2020.
|
| [33] |
ABBOTT R J, BROCHMANN C. History and evolution of the Arctic flora: in the footsteps of Eric Hultén[J]. Molecular Ecology, 2003, 12(2): 299-313. DOI:10.1046/j.1365-294X.2003.01731.x |
| [34] |
LUTTIKHUIZEN P C, STIFT M, KUPERUS P, et al. Genetic diversity in diploid vs.tetraploid Rorippa amphibia (Brassicaceae)[J]. Molecular Ecology, 2007, 16(17): 3544-3553. DOI:10.1111/j.1365-294X.2007.03411.x |
| [35] |
BROCHMANN C, BRYSTING A K, ALSOS I G, et al. Polyploidy in Arctic plants[J]. Biological Journal of the Linnean Society, 2004, 82(4): 521-536. DOI:10.1111/j.1095-8312.2004.00337.x |
| [36] |
FRANKHAM R, RALLS K. Conservation biology: inbreeding leads to extinction[J]. Nature, 1998, 392: 441-442. DOI:10.1038/33022 |
| [37] |
FRANKHAM R, BRADSHAW C J A, BROOK B W. Genetics in conservation management: revised recommendations for the 50/500 rules, red list criteria and population viability analyses[J]. Biological Conservation, 2014, 170: 56-63. DOI:10.1016/j.biocon.2013.12.036 |
| [38] |
CHARLESWORTH B. Effective population size and patterns of molecular evolution and variation[J]. Nature Reviews Genetics, 2009, 10(3): 195-205. DOI:10.1038/nrg2526 |
| [39] |
WRIGHT S. Evolution and the genetics of populations, volume 3:experimental results and evolutionary deductions[M]. Chicago: University of Chicago Press, 1984.
|
| [40] |
MEIRMANS P G, VAN TIENDEREN P H. The effects of inheritance in tetraploids on genetic diversity and population divergence[J]. Heredity, 2013, 110(2): 131-137. DOI:10.1038/hdy.2012.80 |
| [41] |
ORSINI L, VANOVERBEKE J, SWILLEN I, et al. Drivers of population genetic differentiation in the wild: isolation by dispersal limitation, isolation by adaptation and isolation by colonization[J]. Molecular Ecology, 2013, 22(24): 5983-5999. DOI:10.1111/mec.12561 |
| [42] |
陈高, 孙航, 孙卫邦. 大叶醉鱼草居群、种子萌发特征及适应性的初步研究[J]. 武汉植物学研究, 2007, 25(5): 527-530. |
| [43] |
邵长芬, 关文灵, 李得发, 等. 大叶醉鱼草访花昆虫行为与活动规律[J]. 亚热带植物科学, 2014, 43(3): 227-231. |



