中华圆田螺(Cipangopaludina cathayensis)是全国水产十大优异种质资源之一,隶属于腹足纲(Gastropoda)田螺科(Viviparidae)圆田螺属(Cipangopaludina),广泛分布于中国湖泊、水库、沼泽及稻田等环境,因其肉味鲜美、风味独特、营养丰富而成为实体店热销的美食,年产值超百亿元[1-3]。同时,田螺也是青鱼(Mylopharyngodon piceus)、中华绒螯蟹(Eriocheir sinensis)、家禽等经济物种的重要饵料来源,在维持水生生态系统平衡中发挥着至关重要的作用,具有重要的经济价值和生态效益[4, 5]。目前对中华圆田螺的研究主要集中在精子形态[6]、养殖技术[7, 8]、水体修复[9]、生物活性功能[10, 11]、形态特征[12]等方面,而对中华圆田螺种群遗传多样性和遗传分化的研究仍较为薄弱。
微卫星(Microsatellite)又称为简单重复序列(Sample Sequence Repeats,SSR),普遍存在于动物的基因组中,因其具有随机分布性和高水平的等位基因变异性,使其作为遗传标记具有科学有效的应用价值[13, 14]。目前,该技术已广泛应用于梨形环棱螺(Bellamya purificata)[15]、厚壳贻贝(Mytilus unguiculatus)[16]、方斑东风螺(Babylonia areolata)[17]等经济腹足类动物种群遗传结构的研究中。近年来,本课题组进行了走访调研,发现中国的田螺养殖长期采用“养繁一体”的模式,使种质趋于小型化,优质性状退化严重,导致养殖产量低,经济效益差,加之福寿螺(Pomacea canaliculata)入侵严重,侵占了田螺的生态位,进而改变了生境中田螺的生物多样性[18]。因此,为探究广西地区中华圆田螺的遗传多样性和种群结构,本研究采用微卫星分子标记技术分析其10个不同地理种群的遗传资源信息,并筛选出适合广西当地气候条件的养殖种群,为有效开发优异种质资源,开展田螺大规模养殖和选育工作提供科学资料。
1 材料与方法 1.1 样本采集中华圆田螺样本于2022年4-6月分别采集于河池都安(HCDA)、贵港桂平(GGGP)、玉林陆川(YLLC)、梧州藤县(WZTX)、柳州融水(LZRS)、桂林龙胜(GLLS)、贺州八步(HZBB)、南宁横州(NNHZ)、百色田林(BSTL)和百色那坡(BSNP)等10个不同的地理种群,每个种群30个个体(表 1)。将采集到的300个活体野生田螺采用泡沫盒包装加冰袋的方式保持低温,运回实验室后进行肌肉DNA样本采集,并加入适量无水乙醇置于-20 ℃保存备用。采样点地理位置如图 1所示。
| 种群 Population |
采集点 Sampling site |
经纬度 Longitude and latitude |
样本数/个 Number of sample/ind. |
采集日期 Sampling date |
| HCDA | Du′an Yao Autonomous County | 23.93°N,108.10°E | 30 | April 2022 |
| GGGP | Guiping City | 23.39°N,110.07°E | 30 | May 2022 |
| GLLS | Longsheng Various Nationalities Autonomous County | 25.79°N,110.01°E | 30 | May 2022 |
| HZBB | Babu District, Hezhou City | 24.41°N,111.55°E | 30 | June 2022 |
| LZRS | Rongshui Miao Autonomous County | 25.06°N,109.25°E | 30 | May 2022 |
| WZTX | Teng County | 23.37°N,110.91°E | 30 | April 2022 |
| YLLC | Luchuan County | 22.32°N,110.26°E | 30 | June 2022 |
| NNHZ | Hengzhou City | 22.68°N,109.26°E | 30 | May 2022 |
| BSNP | Napo County | 23.38°N,105.83°E | 30 | June 2022 |
| BSTL | Tianlin County | 24.29°N,106.22°E | 30 | June 2022 |
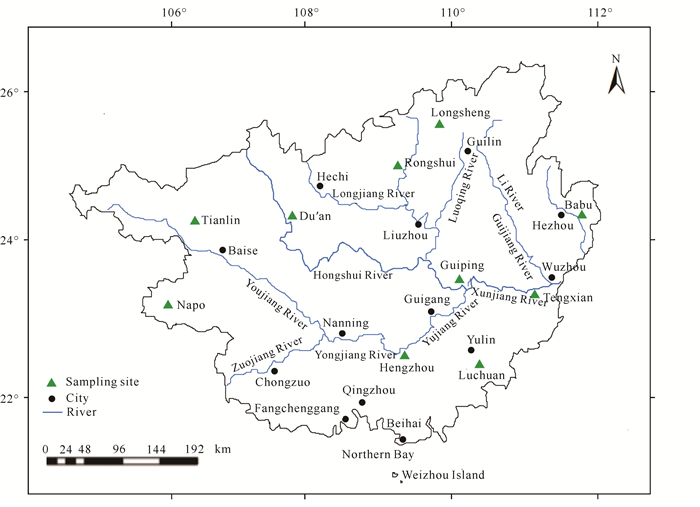
|
| 图 1 采样点地理位置 Fig. 1 Geographic location of sampling points |
1.2 基因组DNA提取、PCR扩增和测序
取20 mg新鲜腹足肌肉,采用磁珠法基因组DNA提取试剂盒[天根生化科技(北京)有限公司]提取基因组DNA,经凝胶成像系统(BIO-RAD,Gel DocTM XR+)和1%琼脂糖凝胶电泳检测合格后,于-20 ℃保存备用。本课题组前期对中国圆田螺的全长转录组数据[美国国家生物技术信息中心(NCBI)登录号为PRJNA817831]进行筛选验证,获得了22对中高多态性微卫星引物,多态信息含量(PIC)为0.247-0.848,本研究从中选择12对PIC大于0.50的微卫星引物进行分析。12对微卫星引物序列使用M13通用接头序列(TGTAAAACGACGGCCAGT)在上游引物5′方向进行修饰,并合成带不同荧光基团的M13接头序列(表 2)。PCR反应体系总体积为15 μL,包含2×Taq PCR Master Mix 7.5 μL,Mix primer 2.0 μL,DNA模板1 μL和ddH2O 4.5 μL。PCR扩增程序:96 ℃预变性3 min;94 ℃变性30 s,62-52 ℃退火30 s,72 ℃延伸1 min,共30个循环;72 ℃延伸10 min,4 ℃保存。取3 μL荧光PCR产物经琼脂糖凝胶电泳鉴定合格后,再经ABI 3730xl DNA测序仪(Applied Biosystem, Inc., USA)进行荧光电泳检测,并使用软件AGeneMarker V2.2.0对原始数据进行条带分型。
| 位点 Locus |
重复单元 Repeat motif |
引物序列(5′→3′) Primer sequence (5′→3′) |
产物大小/bp Size of product/bp |
荧光标记(5′) Fluorescence labeling (5′) |
| CMS063 | (TA)6 | F: CGTCCTGCTTCTTCTATCGC R: GATGCCAGACAACAGAATGC |
160-169 | FAM |
| CMS046 | (AG)10 | F: AAAGGGCAGCATAGGATTCA R: TTGTGGAACGCGATAATTGA |
209-221 | FAM |
| CMS085 | (TTG)6 | F: TCCTTGCTACTGCGGGTACT R: GCATGCACAACAACGAACTT |
252-325 | FAM |
| CMS143 | (GA)10 | F: GGCCTTAAGTGTCGGAATCA R: TGCCTACTGTCCCAGGTTTC |
176-192 | HEX |
| CMS072 | (AC)14 | F: CCCGCCTGAATAGAATGATG R: GAGGAGGAGTGGCATTTCTG |
233-262 | HEX |
| CMS042 | (CA)8 | F: TGCAATCGACTTGATTCTGC R: TCACTTTCAGATGGGAGCCT |
195-201 | TAMRA |
| CMS058 | (GA)7 | F: CAATCATGCAGAGCCTGAAA R: ATGCGTTTACCCATGCCTAT |
273-281 | TAMRA |
| CMS016 | (AG)6 | F: ATTGCATGCATGTGGTCATT R: AAGGGAAAGTCATCATCCCA |
196-209 | FAM |
| CMS190 | (AT)6 | F: GTCACCACTGAATGACACCG R: AGACTTTGGCAATTGGTGCT |
242-246 | FAM |
| CMS055 | (AT)7 | F: ATTACACATGTGGCGAGTGC R: ATCGGTAGCAACAATCCGAA |
285-289 | FAM |
| CMS066 | (CT)9 | F: ATGATCGACGAGGTGGAAAG R: GATCGATACGGACAGACGGT |
212-305 | HEX |
| CMS092 | (AT)7 | F: TAAATCGTTCCACGTGCTCA R: GCAATTTCATGCCTAGCTCA |
237-257 | TAMRA |
1.3 数据分析
使用GenAIex v6.501软件[19]计算12个微卫星位点在10个地理种群中的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He),并分析种群间的Nei′s遗传距离[20]、两两种群间的遗传分化指数(Fst)和香农信息指数(I)以及进行主坐标分析(PCoA)。各种群间的基因流(Nm)计算公式为Nm=0.25(1-Fst)/Fst。采用固定指数(F)衡量观测杂合度偏离哈代-温伯格(Hardy-weinberg)平衡的程度,计算公式为F=(He-Ho)/He。运用Cervus 3.0软件计算各个位点的多态信息含量,采用分子方差分析(AMOVA)计算种群间的遗传结构[21]。种群间的聚类分析采用Phylip 3.6.6软件的非加权组平均法(UPGMA)构建系统进化树。采用Arcmap v10.8.1软件记录各中华圆田螺种群的位置信息,用R软件“vegan”包对遗传距离与地理距离进行Mantel分析。
2 结果与分析 2.1 微卫星位点的多态性12对微卫星引物在10个地理种群中共检测到140个等位基因,等位基因数(Na)为5-30,平均值为12,其中CMS092位点的Na最多(30个),CMS016位点最少(5个);有效等位基因数(Ne)为1.674-13.871,平均值为4.170(表 3)。香农信息指数(I)为0.822-2.869,观测杂合度(Ho)为0.253-0.651,期望杂合度(He)为0.403-0.928。固定指数(F)为0.191-0.639,12个位点的F均大于0,表明各位点均偏离哈代-温伯格(Hardy-weinberg)平衡。12个位点的多态信息含量(PIC)为0.373-0.923,平均值为0.632,其中有9个处于高度多态位点(PIC≥0.5),有3个处于中度多态性位点(0.25≤PIC<0.5)。
| 位点 Locus |
等位基因数(Na) Number of allele types (Na) |
有效等位基因数(Ne) Number of effective alleles (Ne) |
香农信息指数(I) Shannon′s information index (I) |
观测杂合度(Ho) Observed heterozygosity(Ho) |
期望杂合度(He) Expected heterozygosity(He) |
固定指数(F) Fixation index (F) |
多态信息含量(PIC) Polymorphic information content (PIC) |
| CMS016 | 5 | 2.146 | 0.846 | 0.318 | 0.534 | 0.404 | 0.428 |
| CMS190 | 11 | 5.674 | 1.931 | 0.483 | 0.824 | 0.414 | 0.801 |
| CMS055 | 8 | 3.320 | 1.399 | 0.253 | 0.699 | 0.639 | 0.655 |
| CMS066 | 15 | 4.575 | 1.816 | 0.498 | 0.781 | 0.362 | 0.755 |
| CMS092 | 30 | 13.871 | 2.869 | 0.393 | 0.928 | 0.576 | 0.923 |
| CMS063 | 6 | 1.674 | 0.822 | 0.323 | 0.403 | 0.198 | 0.373 |
| CMS046 | 10 | 1.931 | 1.023 | 0.388 | 0.482 | 0.196 | 0.453 |
| CMS085 | 6 | 2.681 | 1.165 | 0.476 | 0.627 | 0.241 | 0.556 |
| CMS143 | 19 | 3.005 | 1.664 | 0.522 | 0.667 | 0.217 | 0.640 |
| CMS072 | 17 | 5.098 | 2.029 | 0.651 | 0.804 | 0.191 | 0.781 |
| CMS042 | 7 | 3.325 | 1.420 | 0.469 | 0.699 | 0.329 | 0.654 |
| CMS058 | 6 | 2.735 | 1.172 | 0.433 | 0.634 | 0.317 | 0.565 |
| Average value | 12 | 4.170 | 1.513 | 0.434 | 0.674 | 0.340 | 0.632 |
2.2 中华圆田螺10个地理种群的遗传多样性
中华圆田螺10个地理种群的遗传多样性如表 4所示。中华圆田螺10个地理种群的Na为2.0-6.0,Ne为1.474-3.467,I为0.415-1.226,Ho为0.231-0.540,He为0.239-0.608,表明不同地理种群的遗传多样性较为丰富;F为0.045-0.210,各种群均偏离Hardy-weinberg平衡,表明种群内的纯合体数量过多。其中,NNHZ种群的遗传多样性水平最高,BSNP种群的遗传多样性水平最低。
| 种群 Population |
数量 Number |
等位基因数(Na) Number of allele types (Na) |
有效等位基因数(Ne) Number of effective alleles (Ne) |
香农信息指数(I) Shannon′s information index (I) |
观测杂合度(Ho) Observed heterozygosity(Ho) |
期望杂合度(He) Expected heterozygosity(He) |
固定指数(F) Fixation index (F) |
| BSNP | 30 | 2.0 | 1.474 | 0.415 | 0.236 | 0.239 | 0.053 |
| NNHZ | 30 | 6.0 | 3.467 | 1.226 | 0.459 | 0.592 | 0.210 |
| BSTL | 30 | 6.0 | 2.893 | 1.121 | 0.471 | 0.560 | 0.124 |
| HCDA | 30 | 3.0 | 1.675 | 0.531 | 0.231 | 0.300 | 0.199 |
| GGGP | 30 | 5.0 | 2.554 | 1.022 | 0.415 | 0.530 | 0.182 |
| GLLS | 30 | 3.0 | 2.229 | 0.903 | 0.498 | 0.524 | 0.045 |
| HZBB | 30 | 6.0 | 3.276 | 1.194 | 0.540 | 0.581 | 0.035 |
| LZRS | 30 | 6.0 | 3.301 | 1.184 | 0.499 | 0.608 | 0.161 |
| WZTX | 30 | 6.0 | 3.132 | 1.172 | 0.486 | 0.558 | 0.109 |
| YLLC | 30 | 5.0 | 2.710 | 1.046 | 0.475 | 0.562 | 0.124 |
| Average value | 30 | 4.8 | 2.671 | 0.981 | 0.431 | 0.505 | 0.124 |
2.3 遗传距离和聚类分析
中华圆田螺10个地理种群间的遗传距离(D)和遗传一致性(DI)分别为0.032-0.961和0.383-0.969,其中HZBB种群与NNHZ种群的遗传距离最远(D=0.032),遗传一致性最大(DI=0.969);HCDA种群与GLLS种群的遗传距离最近(D=0.961),遗传一致性最小(DI=0.383)(表 5)。种群聚类结果显示,HCDA种群和BSNP种群各单独为一支;而GLLS、GGGP、WZTX、HZBB、YLLC、BSTL、LZRS和NNHZ种群聚为一支,表明8个种群亲缘关系较近(图 2)。PCoA结果表明,HCDA种群与BSNP种群各聚为一类,其余种群则相互汇聚在一起,这与UPGMA聚类结果一致(图 3)。
| BSNP | NNHZ | BSTL | HCDA | GGGP | GLLS | HZBB | LZRS | WZTX | YLLC | |
| BSNP | 0.527 | 0.555 | 0.413 | 0.464 | 0.445 | 0.519 | 0.621 | 0.405 | 0.491 | |
| NNHZ | 0.641 | 0.856 | 0.505 | 0.797 | 0.565 | 0.969 | 0.835 | 0.946 | 0.854 | |
| BSTL | 0.589 | 0.156 | 0.480 | 0.691 | 0.675 | 0.841 | 0.839 | 0.832 | 0.954 | |
| HCDA | 0.885 | 0.684 | 0.733 | 0.409 | 0.383 | 0.523 | 0.506 | 0.477 | 0.461 | |
| GGGP | 0.768 | 0.227 | 0.369 | 0.895 | 0.497 | 0.769 | 0.658 | 0.747 | 0.674 | |
| GLLS | 0.810 | 0.571 | 0.394 | 0.961 | 0.700 | 0.553 | 0.594 | 0.553 | 0.601 | |
| HZBB | 0.656 | 0.032 | 0.173 | 0.648 | 0.263 | 0.593 | 0.831 | 0.911 | 0.841 | |
| LZRS | 0.477 | 0.180 | 0.176 | 0.681 | 0.418 | 0.520 | 0.185 | 0.754 | 0.847 | |
| WZTX | 0.905 | 0.055 | 0.184 | 0.740 | 0.291 | 0.593 | 0.094 | 0.283 | 0.819 | |
| YLLC | 0.712 | 0.158 | 0.047 | 0.775 | 0.394 | 0.509 | 0.173 | 0.166 | 0.200 |
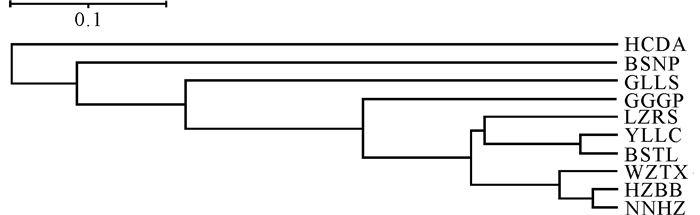
|
| 图 2 UPGMA聚类法构建的分支图 Fig. 2 Branch diagram constructed by UPGMA cluster method |
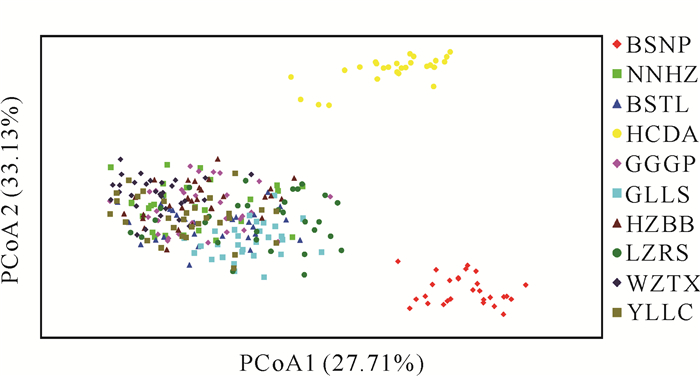
|
| 图 3 中华圆田螺10个地理种群的主坐标分析 Fig. 3 PCoA of 10 geographical populations of C.cathayensis |
2.4 种群间的遗传变异
中华圆田螺10个地理种群间的遗传分化指数(Fst)和基因流(Nm)结果见表 6。各种群间的Fst为0.011-0.390,其中HZBB种群与NNHZ种群的遗传分化程度最小(Fst=0.011),HCDA种群与BSNP种群的遗传分化程度最大(Fst=0.390),且这两个种群与其他种群的Fst均高于0.200。10个地理种群间的Nm为0.391-21.807,其中HCDA种群、BSNP种群与其他8个种群的Nm均小于1.000。分子方差分析结果(表 7)表明,75%的遗传变异来自种群内,25%的遗传变异来自种群间,种群间呈现出高程度的分化(Fst=0.248,P<0.001),其中HCDA种群、BSNP种群与其他种群的遗传分化贡献程度较高、基因流水平低。由Mantel分析结果可知,r值为0.191 7,P值为0.187(图 4),表明地理距离对遗传距离的影响较小,并不是主要的影响因素。
| BSNP | NNHZ | BSTL | HCDA | GGGP | GLLS | HZBB | LZRS | WZTX | YLLC | |
| BSNP | 0.747 | 0.782 | 0.391 | 0.604 | 0.635 | 0.750 | 0.971 | 0.549 | 0.653 | |
| NNHZ | 0.251 | 5.231 | 0.838 | 3.227 | 1.543 | 21.807 | 4.020 | 11.513 | 4.509 | |
| BSTL | 0.242 | 0.046 | 0.762 | 2.049 | 2.007 | 4.719 | 4.289 | 3.843 | 13.819 | |
| HCDA | 0.390 | 0.230 | 0.247 | 0.588 | 0.584 | 0.907 | 0.852 | 0.748 | 0.734 | |
| GGGP | 0.293 | 0.072 | 0.109 | 0.298 | 1.138 | 2.692 | 1.691 | 2.390 | 1.856 | |
| GLLS | 0.282 | 0.139 | 0.111 | 0.300 | 0.180 | 1.500 | 1.629 | 1.379 | 1.550 | |
| HZBB | 0.250 | 0.011 | 0.050 | 0.216 | 0.085 | 0.143 | 4.244 | 6.778 | 4.355 | |
| LZRS | 0.205 | 0.059 | 0.055 | 0.227 | 0.129 | 0.133 | 0.056 | 2.445 | 4.494 | |
| WZTX | 0.313 | 0.021 | 0.061 | 0.251 | 0.095 | 0.153 | 0.036 | 0.093 | 3.387 | |
| YLLC | 0.277 | 0.053 | 0.018 | 0.254 | 0.119 | 0.139 | 0.054 | 0.053 | 0.069 |
| 差异来源 Source of differences |
自由度 Degree of freedom |
总方差 Sum of squares |
均方差 Mean square |
估算的差异值 Estimated variance value |
差异值的百分比/% Percentage of variation/% |
遗传分化指数(Fst) Genetic differentiation index (Fst) |
P值 P value |
| Among populations | 9 | 578.068 | 64.230 | 1.036 | 25 | 0.248 | <0.001 |
| Within populations | 574 | 1 800.723 | 6.294 | 3.147 | 75 | ||
| Total | 583 | 2 378.791 | 4.183 | 100 |
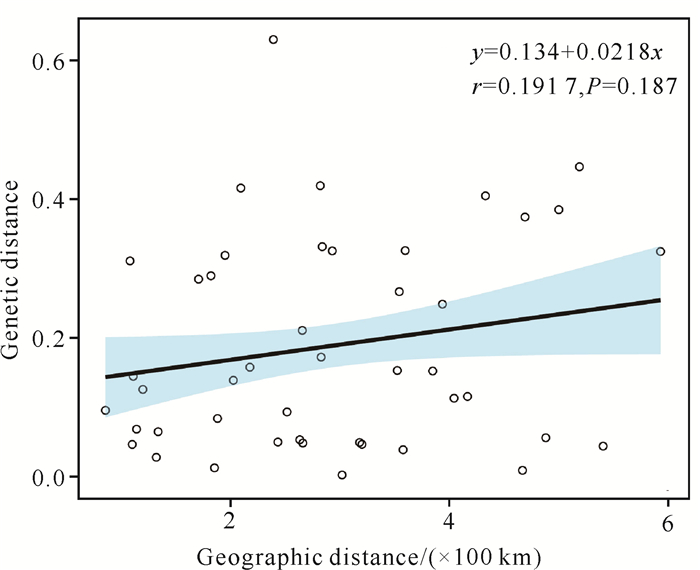
|
| 图 4 地理距离与遗传距离的Mantel分析 Fig. 4 Mantel analysis between geographical distance and genetic distance |
3 讨论 3.1 微卫星引物多样性
近年来,高通量测序技术逐渐发展成熟,为DNA分子标记的开发提供了一种高效便捷的科学方法。王忠良等[22]基于马氏珠母贝(Pinctada martensi)转录组数据挖掘到17对高多态性的EST-SSR引物,为其良种培育奠定了良好基础。熊钢[23]从泥东风螺(Babylonia lutosa)的转录组数据中筛选得到13 962个EST-SSR序列并发掘到7个含EST-SSR序列的生长相关基因,并将其成功应用于野生种群和人工养殖种群的遗传多样性研究中。卢玮筱等[24]通过RNA-seq测序技术获得扁玉螺(Neverita didyma)SSR文库,并从中筛选获得一批高多态性的微卫星分子标记。本研究选用的12对微卫星引物的多态信息含量(PIC)平均值为0.632,其中处于高度多态性位点数量为9个(PIC≥0.5),中度多态性位点数量为3个(0.25≤PIC<0.5)[25, 26]。因此,本研究首次基于近源物种中华圆田螺全长转录组数据获得多态性微卫星标记,并成功应用到中华圆田螺的种群遗传多样性分析中且取得较好的效果,对于后续的研究具有一定参考价值。
固定指数(F)是评价自然选择和遗传漂变作用下种群内的杂合度与总种群杂合度比较后的减少数值[27]。本研究中中华圆田螺10个种群的F值均大于0,均出现偏离Hardy-weinberg平衡的现象,说明这10个种群中的纯合子出现过量的情况,可能是受无效等位基因扩增、杂合子缺少等多种因素的影响,类似的结果在梨形环棱螺[15]和铜锈环棱螺(Bellamya aeruginosa)[28]种群多样性研究中也有发现。
3.2 种群遗传多样性一般认为有效等位基因(Ne)越接近等位基因数(Na)的绝对值,则说明种群中的等位基因分布越均匀。本研究中中华圆田螺10个种群的Na均大于Ne(约2倍),表明各种群间均出现等位基因数量分布不均匀的现象,其中在WZTX种群表现的最为突出(Na/Ne=6.0/3.132),而这种现象在罗氏沼虾(Macrobrachium rosenbergii)[29]、花鲈(Lateolabrax maculatus)[30]等其他水产物种中也出现过。
香农信息指数(I)是评估种群分布均匀度和种群内遗传分化程度的一个重要指标,其数值越大表明种群的遗传多样性越大,种群分化程度越高[31]。邬思荣[32]采用8对微卫星引物对4个壳色马氏珠母贝选育种群进行分析,其种群的I为1.233 8-1.275 0,表明这些种群具有较高的遗传多样性和较大的遗传选育潜力。阮惠婷等[33]对广西主要流域的飘鱼(Pseudolaubuca sinensis)种群进行遗传多样性分析,发现飘鱼I的平均值为0.596 9,说明该种群是随机交配和分布的。本研究中中华圆田螺种群I的平均值为0.981,其中NNHZ种群的I最高(I=1.226),BSNP种群最低(I=0.415),说明广西地区中华圆田螺种群的整体遗传多样性水平较高。
杂合度是评估种群遗传多样性高低的一个重要指标。期望杂合度能根据每个等位基因的基因频率进行计算,不易受样本量的影响,能更好地反映种群遗传多样性水平[34]。本研究结果显示,中华圆田螺10个种群He的平均值为0.505,其中BSNP种群表现出较低的多态性(He=0.239),这与香农信息指数分析所得的结果一致。此外,本研究还发现观测杂合度均小于期望杂合度,推测这些种群可能出现了杂合子缺失的情况[33]。目前大部分研究认为种群的遗传多样性受多种因素的影响,包括人为活动、地理分布、自然灾害、生活习性、进化地位等[35]。虽然人为捕捞、农药滥用、工程改造、福寿螺入侵等对渔业生态造成了严重的影响,但是本研究结果显示广西地区的中华圆田螺仍具有较高的遗传多样性。
3.3 种群间遗传分化基于Nei′s方法计算群体间的遗传距离对探索品种起源、分析种群间的亲缘关系、绘制系统发育树、指导亲本选配等方面具有重要作用[36]。本研究结果显示,中华圆田螺10个地理种群的遗传距离(D)为0.032-0.961,遗传一致性(DI)为0.383-0.969,其中HCDA种群与GLLS种群的D值最大(D=0.961),DI值最小(DI=0.383),说明这两个种群的分化时间长、遗传关系远、遗传变异大。聚类分析和PCoA结果显示,HCDA种群和BSNP种群各单独为一支,而其他8个种群聚为一支,说明这两个种群可能受地理隔离的影响导致与外界交流水平低,人为干扰程度少。
AMOVA结果显示,中华圆田螺以种群内遗传变异(75%)为主,这与梨形环棱螺、铜锈环棱螺等淡水螺的研究结果相似[15, 28]。遗传分化指数(Fst)是评价种群遗传分化程度的重要指标,与基因流(Nm)呈负相关关系[37]。本研究发现BSNP种群和HCDA种群的Fst最大(0.390),Nm最小(0.391),且BSNP种群、HCDA种群与其他种群的Fst均大于0.200,Nm均小于1.000。研究表明,当0.15≤Fst<0.25时,种群间有较大的遗传分化;Fst≥0.25时,种群间存在极大的遗传分化[26]。综上结果可知,BSNP种群和HCDA种群与其他8个种群遗传分化较大,基因交流较少,可能已经发生由遗传漂变引发的实质性遗传分化现象,这与Nei′s遗传距离的分析结果一致。另外,除BSNP种群和HCDA种群外,其余8个地理种群间的Fst为0.011-0.180,Nm均大于1.000,说明这些种群间的遗传分化程度水平较低,基因交流频繁,能够有效地抑制遗传漂变的作用,降低种群间出现遗传分化的风险[38]。Mantel分析结果显示,中华圆田螺各种群间遗传距离与地理距离相关性较低,且NNHZ、GLLS、GGGP、WZTX、HZBB、YLLC、BSTL和LZRS种群的基因交流频繁,受地理隔离影响小,无明显分化现象,保持着较高的遗传多样性水平。因此,笔者推测出现该结果的原因可能有以下两个方面:(1)随着广西地区稻螺人工养殖规模逐步扩大,人为耕作和引种活动频繁,本研究采集的野生种群中可能有一部分为人工养殖逃逸的样本;(2)近年来广西湘江流域、西江流域等地区洪涝灾害频发,而田螺幼仔偏好吸附于枯枝烂叶、塑料薄膜等漂浮物表面,从而促进了中华圆田螺种群的扩散和交流。
4 结论开展广西地区中华圆田螺种群遗传多样性研究,掌握分子系统数据有利于该物种的开发利用。本研究选取的12对微卫星引物为中高多态性位点,适用于田螺种群的遗传学研究与种质资源评估。广西地区中华圆田螺10个地理种群的遗传多样性较高,现有种质资源良好,尤其是HCDA种群和BSNP种群具有较好的开发与利用价值。但随着全国稻螺综合养殖模式的不断发展和福寿螺的入侵,野生田螺的种质资源可能会出现逐步减少的趋势,因此后续还需要对中华圆田螺自然资源进行全面而深入的研究,为田螺优异种质资源的挖掘和良种选育提供科学数据。
| [1] |
程果, 但小琴, 王卫民, 等. 中华圆田螺和铜锈环棱螺的主要风味物质差异性分析[J]. 食品科技, 2021, 46(5): 238-245. |
| [2] |
解婧媛, 孙涛, 廖芳, 等. 柳州地区中华圆田螺和方形环棱螺的含肉率及营养分析[J]. 现代食品, 2021(17): 203-205. |
| [3] |
薛飞, 黄凯, 宿志健, 等. 中华圆田螺氨基酸、脂肪酸营养价值与重金属安全性评价[J]. 渔业科学进展, 2022, 43(1): 180-187. |
| [4] |
YI X, GAO J, LI L, et al. Effects of fattening diets on the nutritional quality and flavor of the adult female Chinese mitten crab (Eriocheir sinensis)[J]. Aquaculture Reports, 2022, 25: 101223. DOI:10.1016/j.aqrep.2022.101223 |
| [5] |
樊荟慧, 杜雪松, 文衍红, 等. 不同饲料对中华圆田螺仔螺生长及存活的影响[J]. 中国饲料, 2020(7): 86-89. |
| [6] |
黄家锐, 郭青松, 但小琴, 等. 两种圆田螺精子的超微形态结构与活力比较研究[J]. 淡水渔业, 2021, 51(6): 11-18. DOI:10.3969/j.issn.1000-6907.2021.06.002 |
| [7] |
解婧媛. 中华圆田螺稻田养殖的研究进展[J]. 农村经济与科技, 2021, 32(11): 56-57, 70. DOI:10.3969/j.issn.1007-7103.2021.11.021 |
| [8] |
黄恒章. 草鱼主养池塘尾水养殖中华圆田螺试验[J]. 中国水产, 2021(7): 75-77. |
| [9] |
邢浩春, 李志亮, 陈梅兰, 等. 中华圆田螺+水培花卉协同净化水质的效果[J]. 贵州农业科学, 2016, 44(9): 148-151. DOI:10.3969/j.issn.1001-3601.2016.09.038 |
| [10] |
陈剑杰, 曹谨玲, 李潇, 等. 氟对中华圆田螺肝胰脏抗氧化酶活性和MDA含量的影响[J]. 生态毒理学报, 2018, 13(1): 268-273. |
| [11] |
周妍英, 罗正明. 镉对中华圆田螺抗氧化酶活性和脂质过氧化的影响[J]. 食品工业科技, 2018, 39(15): 43-47. |
| [12] |
邱齐骏, 颜冬, 龚守贺, 等. 丘陵山区养殖中华圆田螺主要形态性状对螺重的相关性及通径分析[J]. 河北渔业, 2021(11): 3-6, 12. DOI:10.3969/j.issn.1004-6755.2021.11.002 |
| [13] |
PARK Y J, LEE M N, KANG J H, et al. Population genetic structure of Semisulcospira gottschei: simultaneous examination of mtDNA and microsatellite markers[J]. Molecular Biology Reports, 2021, 48(1): 97-104. DOI:10.1007/s11033-020-05821-9 |
| [14] |
PARK Y J, LEE M N, KIM E M, et al. Development and characterization of novel polymorphic microsatellite markers for the Korean freshwater snail Semisulcospira coreana and cross-species amplification using next-generation sequencing[J]. Journal of Oceanology and Limnology, 2020, 38(2): 503-508. DOI:10.1007/s00343-019-9058-0 |
| [15] |
金武, 曹静越, 马骋, 等. 梨形环棱螺11个地理种群遗传多样性和遗传分化[J]. 淡水渔业, 2022, 52(2): 16-21. DOI:10.3969/j.issn.1000-6907.2022.02.003 |
| [16] |
傅泽钦. 我国沿海厚壳贻贝Mytilus unguiculatus群体遗传结构与种群地理分布格局[D]. 舟山: 浙江海洋大学, 2019.
|
| [17] |
朱丹丽. 基于线粒体和微卫星标记的方斑东风螺遗传多样性研究[D]. 长沙: 湖南农业大学, 2016.
|
| [18] |
张春霞. 福寿螺与本地田螺在不同生境胁迫下的响应差异与作用机制研究[D]. 广州: 华南农业大学, 2016.
|
| [19] |
EXCOFFIER L P, SMOUSE P E, QUATTRO J M V. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data[J]. Genetics, 1992, 131(2): 479-491. DOI:10.1093/genetics/131.2.479 |
| [20] |
HATTEMER H H. Genetic distance between populations[J]. Theoretical and Applied Genetics, 1982, 62(3): 219-223. DOI:10.1007/BF00276242 |
| [21] |
MEIRMANS P G. Using the AMOVA framework to estimate a standardized genetic differentiation measure[J]. Evolution: International Journal of Organic Evolution, 2006, 60(11): 2399-2402. DOI:10.1111/j.0014-3820.2006.tb01874.x |
| [22] |
王忠良, 丁燏, 许尤厚, 等. 基于转录组数据的马氏珠母贝EST-SSR位点的信息分析及其多态性检测[J]. 海洋与湖沼, 2015, 46(3): 687-693. |
| [23] |
熊钢. 基于转录组的泥东风螺EST-SSR标记开发及遗传多样性分析[D]. 长沙: 湖南农业大学, 2015.
|
| [24] |
卢玮筱, 陈永霞, 祁鹏志. 扁玉螺转录组SSR信息分析[J]. 浙江海洋大学学报(自然科学版), 2018, 37(3): 215-220. DOI:10.3969/j.issn.1008-830X.2018.03.005 |
| [25] |
KUMAR S P J, SUSMITA C, SRIPATHY K V, et al. Molecular characterization and genetic diversity studies of Indian soybean (Glycine max (L.) Merr.) cultivars using SSR markers[J]. Molecular Biology Reports, 2022, 49(3): 2129-2140. DOI:10.1007/s11033-021-07030-4 |
| [26] |
ALI A, PAN Y B, WANG Q N, et al. Genetic diversity and population structure analysis of Saccharum and Erianthus genera using microsatellite (SSR) markers[J]. Scientific Reports, 2019, 9(1): 395. DOI:10.1038/s41598-018-36630-7 |
| [27] |
熊钢, 王晓清, 王佩, 等. 泥东风螺EST-SSR开发及其群体遗传多样性分析[J]. 渔业科学进展, 2020, 41(4): 117-124. |
| [28] |
顾钱洪, 熊邦喜, 陈洁, 等. 微卫星标记技术在淡水腹足类种群遗传学研究中的应用[J]. 应用与环境生物学报, 2011, 17(2): 280-286. |
| [29] |
李景芬, 夏正龙, 栾生, 等. 五个罗氏沼虾群体遗传多样性的微卫星分析[J]. 水生生物学报, 2020, 44(6): 1208-1214. |
| [30] |
黄皓, 范嗣刚, 王鹏飞, 等. 基于微卫星标记对6个花鲈群体的遗传多样性分析[J]. 南方水产科学, 2022, 18(1): 99-106. |
| [31] |
张民照. 用多态位点率和香农指数分析的飞蝗地理种群遗传多样性[J]. 中国农学通报, 2008, 24(9): 376-381. |
| [32] |
邬思荣. 湛江北部湾深水海域马氏珠母贝四种壳色选育系F5的生长速度、生长模型及其遗传多样性的SSR分析[D]. 湛江: 广东海洋大学, 2011.
|
| [33] |
阮惠婷, 徐姗楠, 李敏, 等. 飘鱼微卫星位点的筛选及珠江流域5个地理群体的遗传多样性分析[J]. 水生生物学报, 2020, 44(3): 501-508. |
| [34] |
QIN Y, SUN D Q, XU T J, et al. Genetic diversity and population genetic structure of the miiuy croaker, Miichthys miiuy, in the East China Sea by microsatellite markers[J]. Genetics and Molecular Research: GMR, 2014, 13(4): 10600-10606. DOI:10.4238/2014.December.18.1 |
| [35] |
HOQUE A, FIEDLER J D, RAHMAN M. Genetic diversity analysis of a flax (Linum usitatissimum L.) global collection[J]. BMC Genomics, 2020, 21(1): 557. DOI:10.1186/s12864-020-06922-2 |
| [36] |
JO K R, CHO S, CHO J H, et al. Analysis of genetic diversity and population structure among cultivated potato clones from Korea and global breeding programs[J]. Scientific Reports, 2022, 12(1): 10462. DOI:10.1038/s41598-022-12874-2 |
| [37] |
李强勇, 李旻, 曾地刚, 等. 凡纳滨对虾微卫星分子标记的开发及不同养殖家系遗传多态性分析[J]. 南方农业学报, 2020, 51(2): 429-436. DOI:10.3969/j.issn.2095-1191.2020.02.024 |
| [38] |
SLATKIN M. Gene flow in natural populations[J]. Annual Review of Ecology and Systematics, 1985, 16: 393-430. DOI:10.1146/annurev.es.16.110185.002141 |



