2. 西华师范大学环境科学与工程学院,四川南充 637009
2. College of Environmental Science and Engineering, China West Normal University, Nanchong, Sichuan, 637009, China
泛素是真核生物中由76个氨基酸构成的高度保守蛋白质[1]。泛素化是一种蛋白质共价修饰过程,通过该过程可以识别特定蛋白质,实现蛋白质功能的激活或抑制[1, 2]。泛素化过程要经过三级级联反应,首先由E1泛素激活酶激活泛素,其次E2泛素结合酶与泛素结合,最后与E3泛素蛋白连接酶共同作用,使底物与泛素相结合,实现泛素化[3]。泛素化在蛋白质调控[4, 5]、病毒入侵[2, 6]、免疫调节[7]、细胞凋亡[8]和肿瘤侵袭[9]等过程中起着重要作用。
ubr1基因编码E3泛素蛋白连接酶N端识别蛋白1(Ubr1),该蛋白含有一个典型的UBR型锌指结构。在球孢白僵菌(Beauveria bassiana)中,ubr1基因缺失会抑制精氨酸代谢,影响孢子萌发[10]。白色念珠菌(Candida albicans)中Ubr1蛋白可通过Cup9蛋白调控菌丝生长[11]。同样地,粟酒裂殖酵母(Schizosaccharomyces pombe)中ubr1基因亦广泛影响其生命活动。ubr1基因缺失会抑制粟酒裂殖酵母在以葡萄糖、麦芽糖、蔗糖、木糖或乙醇为碳源条件下的生长[12, 13],使菌株对多菌灵和雷帕霉素耐药[13, 14],并对伊曲康唑[13]、5′-氟尿嘧啶[15]、米卡芬净和衣霉素[16]敏感。Hayles等[17]的研究发现ubr1基因缺失后,粟酒裂殖酵母细胞形态会变短变粗。人类E3泛素蛋白连接酶包括Ubr1、E3泛素蛋白连接酶N端识别蛋白2(Ubr2)在内的多个连接酶[18]。人类Ubr1蛋白参与N端规则,清除体内受损、错误折叠等异常蛋白质[19],该基因突变会导致Johanson-Blizzard综合征,造成患儿发育迟缓、营养不良及鼻翼发育不全、牙齿畸形的特殊面容[20]。
粟酒裂殖酵母是一种与人类细胞有着相似细胞周期且易于基因编辑的单细胞生物,常被用作研究细胞有丝分裂的模式生物[21]。目前,ubr1基因缺失后粟酒裂殖酵母有丝分裂动力学变化未见报道,因此,本研究以粟酒裂殖酵母细胞为模型,通过在微管蛋白、动粒蛋白和肌动蛋白上构建荧光标记,结合活细胞成像技术分析细胞有丝分裂动力学变化,为进一步明确ubr1基因在细胞有丝分裂中的功能和分子机制提供科学依据。
1 材料与方法 1.1 菌株粟酒裂殖酵母菌株保存于西华师范大学西南野生动植物资源保护教育部重点实验室,菌株信息如表 1所示。
| 菌株编号 Strain number |
基因型 Genotype |
来源 Source |
| PT.286 | WT ade6-210 leu1-32 ura4-D18 h- | Laboratory preservation |
| PT.287 | WT ade6-210 leu1-32 ura4-D18 h+ | Laboratory preservation |
| YL15 | WT GFP-Atb2: : HygR Pact1-LifeAct-mC: : leu1+ ade6-210 ura4-D18 h+ | Laboratory preservation |
| PT2514 | WT Mis12-GFP: : leu1+ mC-Atb2: : HygR ade6-210 ura4-D18 h- | Laboratory preservation |
| HY 2338-1 | ubr1Δ: : KanR ade6.-210 leu1-32 ura4-D18 h+ | Laboratory preservation |
| HY 2338-2 | ubr1Δ: : KanR ade6-210 leu1-32 ura4-D18 h- | Laboratory preservation |
| HY 2338-3 | ubr1Δ: : KanR GFP-Atb2: : HygR Pact1-LifeAct-mC: : leu1+ ade6-210 ura4-D18 h? | This study produce |
| HY 2338-4 | ubr1Δ: : KanR Mis12-GFP: : leu1+ mC-Atb2: : HygR ade6-210 ura4-D18 h? | This study produce |
| Note:WT means wild-type strain, and ubr1Δ means ubr1 -deleted strain. | ||
1.2 培养基
YE5S培养基:葡萄糖15 g,酵母提取物2.5 g,腺嘌呤0.112 5 g,亮氨酸0.112 5 g,尿嘧啶0.112 5 g,组氨酸0.112 5 g,赖氨酸0.112 5 g,无菌水500 mL,若为固体培养基则加入琼脂粉8.5 g。
EMM-N培养基:EMM-N粉末3.66 g,葡萄糖10 g,腺嘌呤0.112 5 g,亮氨酸0.112 5 g,尿嘧啶0.112 5 g,组氨酸0.112 5 g,赖氨酸0.112 5 g,无菌水500 mL,若为固体培养基则加入琼脂粉30 g。
EMM2-Leu培养基:EMM2粉末16.165 g,腺嘌呤0.112 5 g,尿嘧啶0.112 5 g,组氨酸0.112 5 g,赖氨酸0.112 5 g,无菌水500 mL,琼脂粉8.5 g。
YE5S-Hyg培养基:YE5S固体培养基灭菌后加入潮霉素B,使其浓度达到0.3 mg/mL。
1.3 生长曲线测定挑取少量活化后的野生型和ubr1Δ菌株,接种于YE5S液体培养基,于摇床中25 ℃过夜培养,测定菌液在595 nm波长条件下的吸光度值(OD595)。将所得菌液稀释至OD595=0.1,置于25 ℃培养,每2 h检测1次OD595,共测量12 h,绘制生长曲线。
1.4 菌株构建ubr1缺失菌株构建:在PomBase数据库(https://www.pombase.org/)上查找基因序列,取外显子区域两端80 bp设计同源臂,以pFA6a-KanMX6为模板质粒,质粒引物序列为F1:5′-CGGATCCCCCGGGTTAATTAA-3′、R1:5′-GAATT-CGAGCTCGTTTAAAC-3′,以同源重组的方式获得缺失菌株。
单荧光标记菌株构建:以Mis12-GFP为例,在PomBase数据库上查找Mis12终止密码子TAA前后80 bp序列来设计同源臂,以pFA6a-GFP: LEU1为模板质粒,质粒引物序列为F5:5′-GGTGACGGTGCTGGTTTA-3′、R3:5′-TCGATGAATTCGAGC-TCG-3′,扩增得到带同源臂的荧光蛋白序列,同源重组后在EMM2-Leu培养基上筛选获得表达Mis12-GFP融合蛋白菌株。
双荧光标记菌株构建:将构建的两个单标记菌株在EMM-N培养基上诱导产孢,添加0.01 g/mL蜗牛酶于金属浴37 ℃处理2 h,将孢子涂布在EMM2-Leu和YE5S-Hyg培养基上进行筛选,结合奥林巴斯DP72显微镜观察得到带有两种荧光标记的子代菌株[22]。
带荧光标记的缺失菌株:将敲除菌株与荧光标记菌株在EMM-N培养基上诱导产孢后,添加0.01 g/mL蜗牛酶于金属浴37 ℃处理2 h,将孢子涂布在筛选培养基上进行筛选,结合奥林巴斯DP72显微镜观察得到带有相应荧光标记的子代缺失菌株[22]。
1.5 活细胞延时成像在25 ℃下,使用徕卡激光共聚焦显微系统(leica TCS-SP8)对已构建荧光标记的菌株进行活细胞成像。设置参数为曝光时间500 ms,2 min/帧,1帧扫描7层,合计90 min,共45帧[23]。
1.6 数据统计与分析使用绿色荧光蛋白(GFP)标记纺锤体,观察纺锤体的动力学变化,并根据纺锤体的行为划分有丝分裂时期。使用GFP标记动粒、mCherry标记纺锤体,观察动粒蛋白动力学变化。使用mCherry标记肌动蛋白环,以此检测肌动蛋白环动力学差异。使用Image J图像处理软件对荧光标记进行处理分析和数据统计。所有图片采用GraphPad Prism 8作图,用SPSS 26.0软件进行单因素方差分析和t检验,P < 0.05表示差异显著,P < 0.01表示差异极显著。
2 结果与分析 2.1 ubr1基因缺失对粟酒裂殖酵母细胞增殖、细胞形态和微管束的影响粟酒裂殖酵母细胞生长曲线结果显示,ubr1Δ菌株在前4 h内与野生型菌株相比无显著性差异(P>0.05),但培养12 h时ubr1Δ菌株的OD595值为0.239±0.005,野生型菌株的OD595值为0.760±0.006[图 1(a)],表明ubr1基因缺失后酵母细胞增殖受抑制。
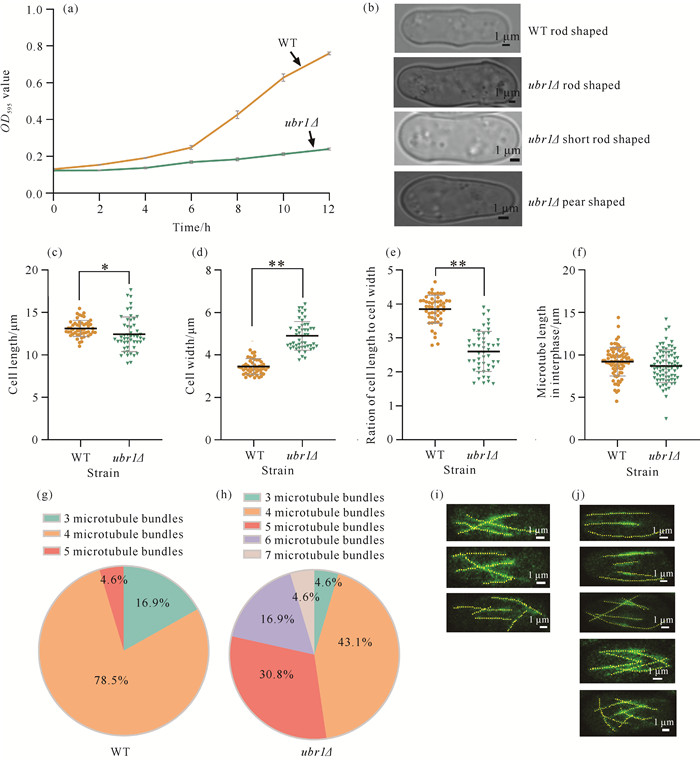
|
| *indicates significant difference(P < 0.05), **indicates extremely significant difference (P < 0.01). 图 1 ubr1基因缺失对细胞增殖、细胞形态和微管束的影响 Fig. 1 Effects of ubr1 deletion on cell proliferation, cell morphology, and microtubule bundles |
细胞形态分析结果显示,野生型菌株细胞呈棒状形态,细胞长度为(13.09±0.94) μm,宽度为(3.44±0.37) μm,长宽比为3.84±0.41;ubr1Δ菌株细胞形态从棒状、短棒状到梨形,细胞长度为(12.41±2.01) μm,宽度为(4.89±0.67) μm,长宽比为2.60±0.58,与野生型菌株相比具有显著性差异(P < 0.05,P < 0.01)[图 1:(b)-(e)]。
有丝分裂间期细胞微管束的长度统计结果表明,野生型菌株和ubr1Δ菌株的微管束长度分别为(9.20±1.67)、(8.69±1.93) μm,两者间无显著差异[P>0.05,图 1(f)]。有丝分裂间期细胞微管束数目的统计结果表明,野生型菌株能产生3、4和5根微管束,其中4根微管束的占比最大,为78.5%[图 1(g)];ubr1Δ菌株能产生3、4、5、6和7根微管束,其比例分别为4.6%,43.1%,30.8%,16.9%和4.6%[图 1(h)],提示ubr1基因缺失会导致粟酒裂殖酵母细胞的微管束增加。图 1(i)和(j)分别为野生型和ubr1Δ菌株的微管模式图。
2.2 ubr1基因缺失对纺锤体动力学的影响ubr1Δ菌株存在两种纺锤体形成形式,ubr1Δ (ⅰ)表示正常形成纺锤体,ubr1Δ (ⅱ)表示异常形成两条纺锤体[图 2(a)]。ubr1Δ菌株中错误形成两条纺锤体的个体占比为18.5%,各自延伸进入两个子细胞中并完成断裂。以有丝分裂间期微管束完全解聚的时刻作为有丝分裂开始的“0”时刻,对纺锤体的伸长长度进行统计分析,结果显示野生型菌株和ubr1Δ菌株分别在5.8、5.9 min进入有丝分裂中期,在13.8、13.1 min进入有丝分裂后期,两种菌株间无显著性差异[图 2(b)]。对细胞有丝分裂各个时期的时长和纺锤体伸长长度进行统计分析发现,ubr1Δ菌株有丝分裂后期的持续时间为(14.8±3.25) min,较野生型菌株显著延长(3.00±1.36) min,且两者间具有显著性差异[P < 0.05,图 2(c)]。在有丝分裂中期野生型菌株纺锤体伸长长度为(1.52±0.48) μm,ubr1Δ菌株为(1.02±0.38) μm,ubr1Δ菌株较野生型菌株显著缩短(0.50±0.10) μm (P < 0.05);而在有丝分裂后期野生型菌株纺锤体伸长长度为(9.05±1.08) μm,ubr1Δ菌株为(10.63±1.84) μm,ubr1Δ菌株较野生型菌株显著增加(1.58±0.76) μm [P < 0.05,图 2(d)]。ubr1基因缺失对有丝分裂各时期纺锤体伸长速度并无显著影响[图 2(e)]。野生型菌株纺锤体断裂方式主要为直线型断裂(63.3%),而ubr1Δ菌株主要为拱型断裂(70.4%),而且还出现了鱼钩型和S型断裂[图 2:(f)-(h)]。以上结果表明ubr1基因缺失会显著缩短有丝分裂中期纺锤体伸长长度,显著增加有丝分裂后期时长和纺锤体伸长长度,细胞纺锤体的主要断裂类型由直线型转为拱型。
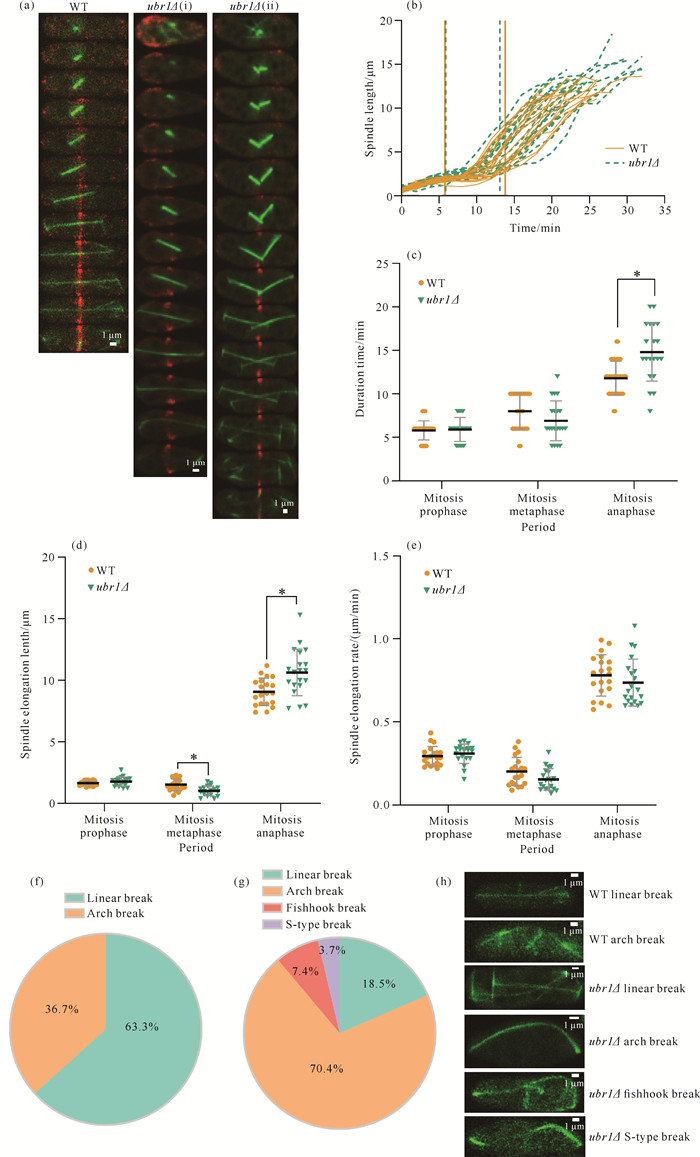
|
| *indicates significant difference (P < 0.05). 图 2 ubr1基因缺失对纺锤体动力学的影响 Fig. 2 Effects of ubr1 deletion on spindle dynamics |
2.3 ubr1基因缺失对动粒动力学的影响
ubr1Δ菌株存在两种动粒形成形式,ubr1Δ (i)表示正常的动粒分裂过程,ubr1Δ (ii)表示动粒错误加倍,占比18.5%[图 3(a)]。有丝分裂开始前,两个动粒聚集在细胞中央;有丝分裂开始后,各自附着一条纺锤体并分裂成两个动粒,最终每个子细胞分得两个动粒[图 3(a)]。以有丝分裂间期微管束完全解聚的时刻为有丝分裂开始的“0”时刻,对有丝分裂过程中动粒间的距离进行分析,结果显示野生型菌株和ubr1Δ菌株的动粒分别在9.4、10.2 min定位到纺锤体的两端,两者间并无显著性差异[图 3(b)]。对动粒定位在纺锤体两端的时间和距离进行统计分析,发现野生型菌株和ubr1Δ菌株亦无显著性差异[图 3:(c)、(d)]。以上结果表明ubr1基因缺失会导致动粒数量出现异常,但对动粒蛋白动力学并无影响。
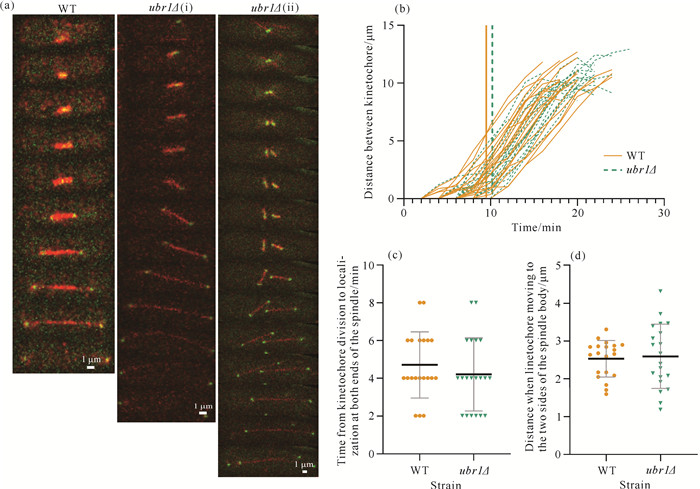
|
| 图 3 ubr1基因缺失对动粒动力学的影响 Fig. 3 Effects of ubr1 deletion on kinetosomal dynamics |
2.4 ubr1缺失对肌动蛋白环动力学的影响
肌动蛋白环在细胞分裂过程中的变化如图 4(a)所示,肌动蛋白环直径随时间变化的统计分析结果如图 4(b)所示。野生型菌株肌动蛋白环的初始直径为(3.69±0.25) μm,而ubr1Δ菌株为(4.81±0.44) μm,两者具有极显著差异[P < 0.01,图 4(c)]。野生型菌株肌动蛋白环的形成时间为(9.80±1.54) min,ubr1Δ菌株为(13.40±4.39) min,ubr1基因缺失后肌动蛋白环的形成时间显著延长(3.60±2.85) min[P < 0.05,图 4(d)]。野生型菌株和ubr1Δ菌株肌动蛋白环的收缩时间分别为(19.60±3.07)、(24.50±3.28) min [图 4(e)];野生型菌株和ubr1Δ菌株肌动蛋白环的收缩速度分别为(0.20±0.03)、(0.20±0.02) μm/min[图 4(f)],表明ubr1基因缺失会导致肌动蛋白环的收缩时间延长,但对肌动蛋白环的收缩速度并无明显影响。
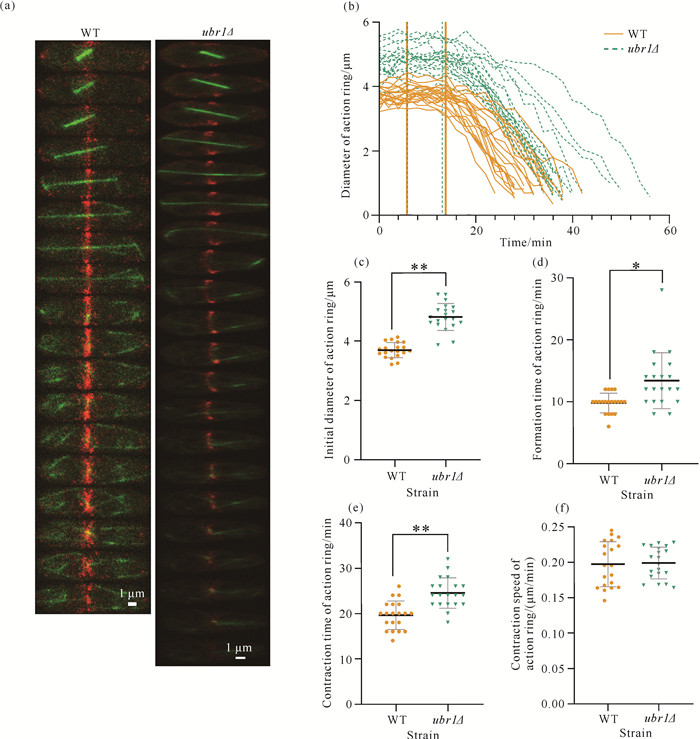
|
| * and ** indicate significant differences at the P<0.05 and P < 0.01 levels, respectively. 图 4 ubr1基因缺失对肌动蛋白环动力学的影响 Fig. 4 Effects of ubr1 deletion on actin ring dynamics |
3 讨论
微管蛋白作为细胞骨架,由多种蛋白因子调控,并参与维持细胞形态[24]。for3基因缺失会导致裂殖酵母细胞微管束数目增加[25],同时细胞变短[26]。Rho型GTP酶激活蛋白4(Rga4)的碳端GAP结构域介导For3蛋白定位到细胞尖端,维持细胞直径[27]。本研究发现ubr1基因缺失后细胞内微管束数目增加,细胞形态异常,呈短棒状和梨形,与Hayles等[17]的初筛结果一致,表明Ubr1蛋白与For3蛋白都有维持有丝分裂间期微管束数目正常的功能,ubr1基因可通过调控有丝分裂间期微管束的数目来维持细胞形态正常。
动粒与纺锤体、动粒与染色质的物理连接,决定了遗传物质的分配情况[28]。动粒蛋白不足或过度会导致微管与染色质的结合过少或者过多[29]。许多肿瘤细胞都存在动粒蛋白失调的现象[30],动粒正成为癌症基因组不稳定的参与者[31]。在裂殖酵母细胞中,动粒蛋白附着在由微管构成的纺锤体上,随着纺锤体的延伸分向两极[32]。Rabitsch等[33]发现裂殖酵母细胞的染色体分离Pcs1蛋白在减数第二次分裂期将动粒的微管结合位点整合到一起,以确保姐妹染色单体的正确分离。Gregan等[34]发现与Pcs1蛋白有互作的Mde4蛋白缺失后,裂殖酵母细胞的动粒会分裂成多个,导致染色体分离滞后,部分细胞染色体错误分离。本研究中ubr1Δ菌株也出现多个动粒,表明Ubr1蛋白亦参与动粒分裂的调控。
根据纺锤体的伸长状态,可区分细胞有丝分裂所处的具体时期[35]。纺锤体的持续时间亦决定了子细胞核的空间位置[36]。纺锤体正确且适度的延伸,是染色体分离的物质基础。在调控纺锤体延伸过程中,cid12基因缺失的裂殖酵母细胞的有丝分裂后期时间变长,纺锤体过度延伸[37],染色体错误分离[17]。spo5基因被敲除后,裂殖酵母细胞减数第二次分裂期纺锤体过度延长,原本位于同一侧的两个孢子被推到另一侧[36]。裂殖酵母细胞中的E3泛素连接酶Dma1主要是确保染色体正确附着在纺锤体上,如果附着错误,Dma1则会推迟细胞质分裂以修正错误[38],Dma1基因缺失会导致纺锤体组装检查点功能受损[39]。本研究中ubr1Δ菌株相比于野生型菌株在有丝分裂后期持续时间延长(3.00±1.36) min、纺锤体伸长长度增加(1.58±0.76) μm,甚至出现鱼钩型和S型断裂,提示Ubr1蛋白主要参与后期纺锤体的伸长调控,但并未参与纺锤体组装检查点的调控, 因此ubr1基因缺失菌株可以正常形成纺锤体并行使功能。
肌动蛋白在细胞分裂中参与收缩环形成,经过收缩环的收缩缢裂,母细胞最终分裂成两个子细胞,完成有丝分裂[40]。在肌动蛋白形成收缩环的过程中,肌动蛋白互作蛋白1(Aip1)与肌动蛋白解聚因子1(Adf1)共同作用,可加强Adf1切割肌动蛋白丝的能力,aip1基因缺失导致裂殖酵母的收缩环更早开始收缩[41]。在调控收缩环收缩的蛋白中,myo52基因编码Ⅴ型肌球蛋白,该基因缺失导致收缩环的收缩时间显著延长[42];myp2基因编码肌球蛋白Ⅱ型重链,该基因缺失虽然可形成形态正常的收缩环,但是收缩环的收缩时间延长[43]。本研究发现ubr1Δ菌株肌动蛋白环直径增加,形成时间和收缩时间延长,但是收缩速度与野生型菌株并无差异,提示Ubr1蛋白参与调控肌动蛋白环直径的大小,并由于肌动蛋白环直径增加导致形成时间和收缩时间延长,与肌动蛋白环收缩速度无关。
4 结论本研究采用荧光蛋白标记和活细胞成像技术分析ubr1Δ菌株和野生型菌株在细胞形态和有丝分裂期的细胞动力学差异,结果表明ubr1基因缺失会导致粟酒裂殖酵母细胞增殖缓慢,细胞内微管束数目增多,细胞形态由棒状向短棒状、梨形变化。ubr1Δ菌株在有丝分裂期出现多个动粒,中期纺锤体伸长长度缩短,后期纺锤体伸长长度和持续时间增加。同时,纺锤体出现鱼钩型和S型的断裂方式。ubr1Δ菌株肌动蛋白环较野生型更宽,且形成时间和收缩时间均延长,但收缩速度与野生型并无差异。本研究揭示了ubr1基因缺失对粟酒裂殖酵母细胞有丝分裂动力学的影响,为明确ubr1基因在有丝分裂中的功能及分子机制提供了科学依据,但导致细胞动力学变化的上下游蛋白还有待进一步研究。
| [1] |
ZHENG N, SHABEK N. Ubiquitin ligases: structure, function, and regulation[J]. Annual Review of Biochemistry, 2017, 86: 129-157. DOI:10.1146/annurev-biochem-060815-014922 |
| [2] |
吴玉湖, 胡欣妍, 杨宣叶, 等. 蛋白质泛素化修饰对流感病毒增殖和致病性的影响[J]. 病毒学报, 2023, 39(4): 1152-1160. |
| [3] |
PICKART C M. Mechanisms underlying ubiquitination[J]. Annual Review of Biochemistry, 2001, 70: 503-533. DOI:10.1146/annurev.biochem.70.1.503 |
| [4] |
任宇, 燕文荃, 丁玥竹, 等. VPS28通过泛素化信号通路调控奶牛乳腺上皮细胞中乳蛋白的合成[J]. 畜牧兽医学报, 2022, 53(8): 2558-2567. DOI:10.11843/j.issn.0366-6964.2022.08.014 |
| [5] |
FOOT N, HENSHALL T, KUMAR S. Ubiquitination and the regulation of membrane proteins[J]. Physiological Reviews, 2017, 97(1): 253-281. DOI:10.1152/physrev.00012.2016 |
| [6] |
BARRADO-GIL L, GALINDO I, MARTÍNEZ-ALONSO D, et al. The ubiquitin-proteasome system is required for African swine fever replication[J]. PLoS One, 2017, 12(12): e0189741. DOI:10.1371/journal.pone.0189741 |
| [7] |
LIU X, WANG Q, CHEN W, et al. Dynamic regulation of innate immunity by ubiquitin and ubiquitin-like proteins[J]. Cytokine & Growth Factor Reviews, 2013, 24(6): 559-570. |
| [8] |
NEUTZNER A, LI S, XU S, et al. The ubiquitin/proteasome system-dependent control of mitochondrial steps in apoptosis[J]. Seminars in Cell & Developmental Biology, 2012, 23(5): 499-508. |
| [9] |
SEO H G, KIM H B, YOON J Y, et al. Mutual regulation between OGT and XIAP to control colon cancer cell growth and invasion[J]. Cell Death & Disease, 2020, 11(9): 815. |
| [10] |
郭鸿蓉, 林晓凤, 朱玲, 等. 转录组和蛋白组联合分析揭示Ubr1介导的白僵菌萌发和极性生长[J]. 微生物学报, 2022, 62(8): 3176-3189. |
| [11] |
LU Y, SU C, UNOJE O, et al. Quorum sensing controls hyphal initiation in Candida albicans through Ubr1-mediated protein degradation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(5): 1975-1980. |
| [12] |
BURR R, STEWART E V, SHAO W, et al. Mga2 transcription factor regulates an oxygen-responsive lipid homeostasis pathway in fission yeast[J]. The Journal of Biological Chemistry, 2016, 291(23): 12171-12183. DOI:10.1074/jbc.M116.723650 |
| [13] |
RODRÍGUEZ-LÓPEZ M, BORDIN N, LEES J, et al. Broad functional profiling of fission yeast proteins using phenomics and machine learning[J]. eLife, 2023, 12: RP88229. DOI:10.7554/eLife.88229.3 |
| [14] |
PENNEY M, SAMEJIMA I, WILKINSON C R, et al. Fission yeast 26S proteasome mutants are multi-drug resistant due to stabilization of the Pap1 transcription factor[J]. PLoS One, 2012, 7(11): e50796. DOI:10.1371/journal.pone.0050796 |
| [15] |
MOJARDÍN L, BOTET J, MORENO S, et al. Chromosome segregation and organization are targets of 5′-Fluorouracil in eukaryotic cells[J]. Cell Cycle, 2015, 14(2): 206-218. DOI:10.4161/15384101.2014.974425 |
| [16] |
MA Y, JIANG W, LIU Q, et al. Genome-wide screening for genes associated with FK506 sensitivity in fission yeast[J]. PLoS One, 2011, 6(8): e23422. DOI:10.1371/journal.pone.0023422 |
| [17] |
HAYLES J, WOOD V, JEFFERY L, et al. A genome-wide resource of cell cycle and cell shape genes of fission yeast[J]. Open Biology, 2013, 3(5): 130053. DOI:10.1098/rsob.130053 |
| [18] |
VU T T M, VARSHAVSKY A. The ATF3 transcription factor is a short-lived substrate of the arg/N-degron pathway[J]. Biochemistry, 2020, 59(30): 2796-2812. |
| [19] |
VARSHAVSKY A. The ubiquitin system, an immense realm[J]. Annual Review of Biochemistry, 2012, 81: 167-176. DOI:10.1146/annurev-biochem-051910-094049 |
| [20] |
符晓莉, 张丽, 魏绪霞, 等. 一例UBR1基因新变异所致Johanson-Blizzard综合征患儿的分析[J]. 中华医学遗传学杂志, 2022, 39(12): 1379-1384. |
| [21] |
RUSTICI G, MATA J, KIVINEN K, et al. Periodic gene expression program of the fission yeast cell cycle[J]. Nature Genetics, 2004, 36(8): 809-817. |
| [22] |
袁荣美, 丁祥, 谭秀梅, 等. sgf73基因敲除对粟酒裂殖酵母细胞有丝分裂的影响[J]. 微生物学杂志, 2022, 42(6): 10-24. |
| [23] |
白鑫, 丁祥, 蒲帝宏, 等. 粟酒裂殖酵母SPAC27E2.11c基因缺失对有丝分裂期微管动力学的影响[J]. 厦门大学学报(自然科学版), 2023, 62(5): 848-857. |
| [24] |
KAPITEIN L C, HOOGENRAAD C C. Building the neuronal microtubule cytoskeleton[J]. Neuron, 2015, 87(3): 492-506. |
| [25] |
FEIERBACH B, CHANG F. Roles of the fission yeast formin for3p in cell polarity, actin cable formation and symmetric cell division[J]. Current Biology, 2001, 11(21): 1656-1665. |
| [26] |
PRESTI L L, MARTIN S G. Shaping fission yeast cells by rerouting actin-based transport on microtubules[J]. Current Biology, 2011, 21(24): 2064-2069. |
| [27] |
DAS M, WILEY D J, MEDINA S, et al. Regulation of cell diameter, For3p localization, and cell symmetry by fission yeast Rho-GAP Rga4p[J]. Molecular Biology of the Cell, 2007, 18(6): 2090-2101. |
| [28] |
PAVIN N, TOLIC I M. Self-organization and forces in the mitotic spindle[J]. Annual Review of Biophysics, 2016, 45: 279-298. |
| [29] |
THANGAVELU P U, LIN C Y, VAIDYANATHAN S, et al. Overexpression of the E2F target gene CENPI promotes chromosome instability and predicts poor prognosis in estrogen receptor-positive breast cancer[J]. Oncotarget, 2017, 8(37): 62167-62182. |
| [30] |
ZHANG W G, MAO J H, ZHU W, et al. Centromere and kinetochore gene misexpression predicts cancer patient survival and response to radiotherapy and chemotherapy[J]. Nature Communications, 2016, 7: 12619. |
| [31] |
PESENTI M E, WEIR J R, MUSACCHIO A. Progress in the structural and functional characterization of kinetochores[J]. Current Opinion in Structural Biology, 2016, 37: 152-163. |
| [32] |
DING R, MCDONALD K L, MCINTOSH J R. Three-dimensional reconstruction and analysis of mitotic spindles from the yeast, Schizosaccharomyces pombe[J]. The Journal of Cell Biology, 1993, 120(1): 141-151. |
| [33] |
RABITSCH K P, PETRONCZKI M, JAVERZAT J P, et al. Kinetochore recruitment of two nucleolar proteins is required for homolog segregation in meiosis Ⅰ[J]. Developmental Cell, 2003, 4(4): 535-548. |
| [34] |
GREGAN J, RIEDEL C G, PIDOUX A L, et al. The kinetochore proteins Pcs1 and Mde4 and heterochromatin are required to prevent merotelic orientation[J]. Current Biology, 2007, 17(14): 1190-1200. |
| [35] |
YUAN R M, DING X, TAN X M, et al. Loss of FZO1 gene results in changes of cell dynamics in fission yeast[J]. International Journal of Molecular Medicine, 2020, 46(6): 2194-2206. |
| [36] |
FLOR-PARRA I, IGLESIAS-ROMERO A B, SALAS-PINO S, et al. Importin α and vNEBD control meiotic spindle disassembly in fission yeast[J]. Cell Reports, 2018, 23(4): 933-941. |
| [37] |
WIN T Z, STEVENSON A L, WANG S W. Fission yeast Cid12 has dual functions in chromosome segregation and checkpoint control[J]. Molecular and Cellular Biology, 2006, 26(12): 4435-4447. |
| [38] |
JOHNSON A E, COLLIER S E, OHI M D, et al. Fission yeast Dma1 requires RING domain dimerization for its ubiquitin ligase activity and mitotic checkpoint function[J]. The Journal of Biological Chemistry, 2012, 287(31): 25741-25748. |
| [39] |
MURONE M, SIMANIS V. The fission yeast dma1 gene is a component of the spindle assembly checkpoint, required to prevent septum formation and premature exit from mitosis if spindle function is compromised[J]. The EMBO Journal, 1996, 15(23): 6605-6616. |
| [40] |
POLLARD T D, WU J Q. Understanding cytokinesis: lessons from fission yeast[J]. Nature Reviews Molecular Cell Biology, 2010, 11(2): 149-155. |
| [41] |
CHEN Q, COURTEMANCHE N, POLLARD T D. Aip1 promotes actin filament severing by cofilin and regulates constriction of the cytokinetic contractile ring[J]. The Journal of Biological Chemistry, 2015, 290(4): 2289-2300. |
| [42] |
WANG N, LEE I J, RASK G, et al. Roles of the TRAPP-Ⅱ complex and the exocyst in membrane deposition during fission yeast cytokinesis[J]. PLoS Biology, 2016, 14(4): e1002437. |
| [43] |
OKADA H, WLOKA C, WU J Q, et al. Distinct roles of myosin-Ⅱ isoforms in cytokinesis under normal and stressed conditions[J]. iScience, 2019, 14: 69-87. |



