2. 广西科学院生物科学与技术研究所, 非粮生物质能技术全国重点实验室, 国家非粮生物质能源工程技术研究中心, 广西南宁 530007;
3. 南宁学院食品与质量工程学院, 广西南宁 541699
2. National Key Laboratory of Non-food Biomass Energy Technology, National Engineering Research Center for Non-Food Biorefinery, Institute of Biological Sciences and Technology, Guangxi Academy of Sciences, Nanning, Guangxi, 530007, China;
3. College of Food and Quality Engineering, Nanning University, Nanning, Guangxi, 541699, China
几丁质又名甲壳质、甲壳素、甲壳胺等,是由N-乙酰-D-葡萄糖胺通过β-1, 4糖苷键连接而成的线性糖类多聚物,是自然界中含量仅次于纤维素的第二大天然有机化合物,是甲壳类动物外壳、昆虫和节肢动物的外骨骼、真菌和藻类细胞壁的组成部分[1-3]。据统计,几丁质全球每年的产量为1×1010-1×1011 t[4, 5]。由于难溶于水、稀酸、碱等溶液中,每年加工虾蟹等甲壳类动物产生的大量几丁质难以得到有效开发利用,造成严重的资源浪费及环境危害。然而,几丁质的降解产物N-乙酰-D-氨基寡糖具有良好的水溶性与生物相容性,以及抗菌、抗病毒、抗肿瘤、降低胆固醇、促进伤口愈合、促进生长以及增强免疫力等多种生物活性,在生物医药、食品、农业和环保等领域具有较好的应用价值[3]。
几丁质酶是专一催化分解几丁质生成几丁寡糖和单糖的一类糖苷水解酶[6],广泛分布于各种生物体中,如细菌、真菌、植物、动物以及人类等[4, 7]。根据作用方式,几丁质酶可分为内切几丁质酶和外切几丁质酶[8]。内切几丁质酶在几丁质内部随机切割β-1, 4糖苷键,生成N-乙酰-D-氨基葡萄糖寡聚体或几丁质二糖,主要来源于糖苷水解酶18(GH18)家族和糖苷水解酶19(GH19)家族;外切几丁质酶从非还原几丁质末端切割几丁二糖单位,生成几丁二糖或N-乙酰氨基葡萄糖单体,主要来源于GH18和糖苷水解酶20(GH20)家族[9-12]。
根据氨基酸序列相似性,在碳水化合物活性酶数据库(http://www.cazy.org/)中,几丁质酶主要属于GH18和GH19家族。细菌来源的几丁质酶大部分属于GH18家族[13]。GH18家族几丁质酶的催化结构为经典的(β/α)8 TIM桶状结构,包括8个平行β-折叠和外侧围绕的8个α-螺旋。除催化结构(CD)外,GH18家族几丁质酶通常还包含其他多个结构域,例如独立的碳水化合物结合模块(CBM)、富含丝氨酸/苏氨酸的连接区以及基质辅助结合模块(例如第三型纤维连接蛋白FN-Ⅲ)[14]。根据催化结构域的序列和结构特征,细菌GH18家族几丁质酶又可分为A、B和C 3个亚家族[15]。A亚家族存在几丁质插入结构域(CID),即在原有的(β/α)8结构域中插入(α+β)结构域,增加了底物结合口袋的深度,有助于促进酶与长链底物的结合以及提升酶沿着几丁质长链移动的持续性,多为外切几丁质酶[16];B亚家族在loop6上存在发夹结构,多为内切几丁质酶;C亚家族则不存在插入结构,且loop7区域较长[15, 17]。到目前为止,已从细菌中分离出数百种几丁质酶。然而,由于大多数的几丁质酶为中温或中性酶,不能满足恶劣工业环境的多样化需求,因此,性能更好的新型几丁质酶在几丁质的工业生产中将具有较大的应用前景。与陆地细菌来源的几丁质酶相比,海洋细菌来源的几丁质酶往往具有较高的盐度耐受性、pH值稳定性和热稳定性,以及较多样的结构特征和功能等,是几丁质产业化利用的主要酶制剂[18]。
微泡菌(Microbulbifer)为革兰氏阴性菌,1997年首次被命名,目前有30个成员(www.bacterio.net/genus/Microbulbifer),广泛分布于海洋或盐碱环境中,包括深海沉积物、潮间带滩涂、红树林沉积物以及海洋藻类等[19]。本课题组前期从文昌青澜半岛逸龙湾海滩马尾藻样品中筛选到一株具有较强几丁质降解能力的耐热微泡菌M.thermotolerans YLW106(菌种保藏号:CGMCC 27855),本研究将对来自该菌株的几丁质酶Chi2375进行基因克隆、表达和初步的酶学性质分析,拟为几丁质废弃物高值化利用提供基础研究数据。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒耐热微泡菌M.thermotolerans YLW106、表达载体pET30a、感受态细胞Escherichia.coli DH5α、Escherichia.coli BL21(DE3)等均由本实验室保存。
1.1.2 主要试剂和仪器PrimerSTART Maxmix、In-Fusion试剂盒、限制性内切酶Nde I、Not I等购自Takara(宝)生物有限公司;胶回收试剂盒、DNA产物纯化试剂盒、质粒DNA快速小提试剂盒等购自天根生化科技(北京)有限公司;蛋白纯化用Ni-Fast填料购自Qiagen公司,阴离子柱(Hiload 16/10 Q Sepharose High Performance)和分子筛(Superdex 75 Increase 10/300 GL、Sperdex Peptide 10/30 GL)购自Cytiva公司,引物由武汉金开瑞生物工程有限公司合成,所使用的所有其他化学品和试剂均为分析级试剂。梯度PCR仪TPro fessional Thermocycler购自德国Biometra公司,凝胶成像仪Gel DocTM XR+购自美国Bio-Rad公司,蛋白层析仪AKTA purifier购自美国GE公司,高效液相色谱仪(HPLC)Ultimate 3000购自美国DIONEX公司。
1.2 方法 1.2.1 胶体几丁质的制备参考文献[20]方法制备胶体几丁质。将5 g粉状几丁质加入100 mL浓盐酸,缓慢搅拌至无颗粒黏稠液体状态,密封然后将其置于4 ℃冷藏24 h。然后向经浓盐酸处理的液体中加入超纯水,离心后弃上清液,多次超纯水离心洗涤至pH值为中性,取出冻干为粉末状,过150目筛备用。
1.2.2 几丁质酶基因的克隆从耐热微泡菌M.thermotolerans YLW106的基因测序结果中筛选出含有完整序列的几丁质酶基因Chi2375。根据Chi2375的基因序列,利用Takara网站(https://www.takarabio.com/learning-centers/cloning/primer-design-and-other-tools),设计含有限制性内切酶NdeⅠ识别序列(CATATG)和NotⅠ识别序列(GCGGCCGC)的引物Chi2375 -F和Chi2375 -R(表 1)。
| 无缝克隆引物 Infusion cloning primers |
引物顺序5′到3′ Primer sequence 5′ to 3′ |
| Chi2375-F | AAGGAGATATACATATGGTGGACTGCAGAAGTCTGCCC |
| Chi2375-R | TGCTCGAGTGCGGCCGCGGGCAGATTGTGTACATGGCTGC |
PCR程序:98 ℃ 3 min;98 ℃ 15 s,65 ℃ 15 s,72 ℃ 20 s,36个循环;72 ℃ 10 min。将PCR扩增得到的DNA片段进行胶回收纯化,与经NdeⅠ和NotⅠ双酶切处理后纯化的pET30a质粒,在In-Fusion连接酶作用下进行连接,然后将连接产物转化到E.coli DH5α感受态细胞中,提取质粒送深圳华大基因科技有限公司测序。将构建成功的重组质粒命名为pET30a- Chi2375。
1.2.3 序列分析和结构预测采用NCBI的BLAST程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行氨基酸序列分析,采用SignalIP 6.0工具(https://services.healthtech.dtu.dk/services/SignalP-6.0/)进行信号肽序列分析,采用CLUSTALW程序(https://www.genome.jp/tools-bin/clustalw)和ESPript3程序(https://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)进行氨基酸多序列比对,采用NCBI的CD-search工具(https://www.ncbi.nlm.nih.gov/cdd/)进行保守域的分析,使用InterProScan工具(https://www.ebi.ac.uk/interpro/search/sequence/)进行结构域分析,采用ExPASy的ProtParam工具(http://web.expasy.org/protparam/)进行理论分子量和等电点的预测,采用MEGA 7.0软件对序列进行进化树构建。采用AlphaFold2模型生成序列的三维(3D)结构模型,采用PyMOL程序(SchrÖdinger LLC, Cambridge, MA, USA)对3D图形进行分析。
1.2.4 重组几丁质酶基因在大肠杆菌的诱导表达及纯化将测序正确的重组质粒转入E.coli BLR(DE3)感受态细胞中,于37 ℃、220 r·min-1条件下培养至菌液OD600为0.4-0.8时,加入终浓度为0.5 mmol·L-1的异丙基-β-D-硫代半乳糖苷(IPTG),在16 ℃、150 r·min-1条件下诱导24 h,离心收集菌体。超声波破胞后用镍亲和层析(Ni-Fast)、阴离子柱(Hiload 16/10 Q Sepharose High Performance)及分子筛(Superdex 75 Increase 10/300 GL)纯化重组酶Chi2375,并对纯化后的重组酶进行变性聚丙烯酰胺凝胶电泳(SDS-PAGE)分析。
1.2.5 重组酶的酶学性质分析重组酶的酶活力测定:采用二硝基水杨酸(DNS)法测定还原糖含量,计算几丁质酶活力[21]。反应混合物体积为300 μL,由10 μL适当稀释的酶液,以及290 μL含50 mmol·L-1 Tris-HCl(pH值为8.0)缓冲液的1% (W/V)胶体几丁质组成。反应混合物在55 ℃下反应30 min后,加入300 μL DNS溶液终止反应,沸水浴10 min进行显色反应,冰浴冷却10 min后,12 000 r·min-1离心5 min,在540 nm处测定上清液的吸光值。以沸水浴10 min后的灭活酶液作为空白对照,每组实验设置3组平行及1组空白对照,取平均值,根据N-乙酰D-氨基葡萄糖标准曲线计算酶活力。酶活力单位(U)定义:标准测定条件下,每分钟水解胶体几丁质释放1 μmol N-乙酰D-氨基葡萄糖所需的酶量。
(1) pH值对重组酶的影响
取稀释的酶液,以1% (W/V)的胶体几丁质为底物,在37 ℃条件下,测定50 mmol·L-1不同pH值的缓冲液(醋酸-醋酸钠缓冲液:pH值为4.0-6.0;磷酸氢二钠-磷酸二氢钠缓冲液:pH值为6.0-8.0;Tris-HCl缓冲液:pH值为8.0-9.0;甘氨酸-NaOH缓冲液:pH值为9.0-10.0)对重组酶酶活力的影响,得到重组酶的最适pH值。将重组酶保存在不同pH值的缓冲液中(甘氨酸-盐酸:pH值为2.5-3.5;醋酸-醋酸钠缓冲液:pH值为3.5-6.0;磷酸盐缓冲液:pH值为6.0-8.0;Tris-HCl缓冲液:pH值为8.0-9.0;甘氨酸-NaOH缓冲液:pH值为8.5-12.0),于4 ℃冰箱放置24 h;然后,根据酶活力测定方法,将经不同pH值缓冲液处理过的酶液加入反应体系中,测定重组酶的pH稳定性。每组3个平行,计算不同pH值下重组酶的酶活力。
(2) 温度对重组酶的影响
在最适pH值条件下,测定不同反应温度(35-70 ℃,间隔5 ℃)对重组酶酶活力的影响,以测得的最高酶活力为100%,计算不同温度下的相对酶活力,确定最适反应温度。将重组酶置于不同温度(35-65 ℃,间隔5 ℃)孵育30 min,然后在最适反应条件下测定重组酶残余酶活力,以未处理酶液的酶活力为100%,计算重组酶在各温度下保温30 min的残余酶活力,分析重组酶的热稳定性。将重组酶分别放置在50和55 ℃下,测定随时间变化重组酶的酶活力,分析重组酶的半衰期t1/2值;在梯度PCR仪上,取稀释的重组酶在一系列温度(48、50、52、54、56、58和60 ℃)上热激30 min,然后在最适反应温度和pH值条件下测定残余酶活力,分析重组酶的热半失活温度T5030值;以未经温度处理的重组酶的酶活力为100%,每组3个平行,计算不同温度条件下重组酶的酶活力。
(3) 底物特异性、Km和Vmax的测定
分别以1% (W/V)的胶体几丁质、虾壳细粉几丁质、α-几丁质、β-几丁质、壳聚糖、羧甲基纤维素钠(CMC-Na)为底物,在最适条件下测定重组酶酶活力。
在最适pH值和最适温度条件下,测定以不同浓度(10-70 mg·mL-1)胶体几丁质为底物时重组酶的酶活力,使用软件Origin绘制1/V-1/[S]双倒数曲线,得出重组酶的Km、Vmax。
(4) 金属离子和化学试剂对重组酶的影响
取经过稀释的重组酶与终浓度分别为1和5 mmol·L-1的金属离子(Na+、Zn2+、Mn2+、Ba2+、Ni2+、Ca2+、Mg2+、K+、Co2+、Cu2+、Fe2+、Fe3+),以及终浓度分别为0.5%和1.0%的有机试剂[Triton X-100、乙醇、异丙醇、二甲基亚砜(DMSO)、吐温-20、吐温-80、乙二胺四乙酸(EDTA)、十二烷基硫酸钠(SDS)、二硫苏糖醇(DTT)]混匀后在4 ℃孵育24 h,在最适条件下测定它们对重组酶酶活力的影响。以不添加影响因子的酶活力为100%,计算各种试剂作用下重组酶的残余酶活力。
(5) 盐离子浓度对重组酶的影响
取经过稀释的重组酶与终浓度为0.5-3.5 mol·L-1的NaCl溶液混匀后在4 ℃孵育24 h,在最适反应条件下测定它们对重组酶酶活力的影响。以不添加NaCl溶液的酶活力为100%,计算不同浓度NaCl溶液作用下重组酶的残余酶活力。
(6) 水解产物分析
以几丁单糖、几丁二糖、几丁三糖、几丁四糖为标准样,测定重组酶的水解产物。在290 μL含0.5% (W/V)胶体几丁质的体系中,加入浓度为2 mg·mL-1的重组酶10 μL,于50 ℃反应0.5、2.0、4.0、8.0、24.0、48.0、72.0 h,沸水浴10 min终止反应,室温冷却,12 000 r·min-1离心10 min后取上清液。采用高效液相色谱仪和分子排阻色谱法(SEC)检测重组酶Chi2375降解胶体几丁质生成的产物,使用示差折光检测器,分离柱为分子筛Sperdex Peptide 10/30 GL(Cytiva,USA);流动相为超纯水,流速为0.3 mL·min-1;柱温为常温。
2 结果与分析 2.1 几丁质酶生物信息学分析根据耐热微泡菌M.thermotolerans YLW106的基因组序列分析,获得几丁质酶的基因序列Chi2375 (NCBI登录号:OR979724)。该基因序列与M.thermotolerans HB226069菌株的几丁质酶基因(NCBI登录号:CP130317)、M.thermotolerans DAU221菌株的几丁质酶基因(NCBI登录号:CP014864)以及Microbulbifer sp. YPW1菌株的几丁质酶基因(NCBI登录号:CP055157)的序列一致性分别为99.54%、99.41%和78.86%。Chi2375的氨基酸序列与来源于Microbulbifer sp. GX H0434的几丁质酶(NCBI登录号:WP_250460025)、M.hydrolyticus的几丁质酶(NCBI登录号:AAT81213.1)以及M.pacificus的几丁质酶(NCBI登录号:WP_233998789.1)的一致性分别为80.93%、60.41%和77.89%。使用ProtParam工具分析,Chi2375基因全长1 521 bp,编码507个氨基酸,理论分子量为53.79 kD,等电点为4.732。使用SignalIP 6.0工具分析信号肽序列,显示Chi2375的第18和19位氨基酸残基为信号肽酶切割位点。使用InterProScan工具分析,Chi2375含有1个Glyco_18结构域(位于196-507位氨基酸残基)、2个碳水化合物结合结构域(CBM,分别位于第19-74位氨基酸残基,第109-156位氨基酸残基),以及连接不同结构域之间的、由多个丝氨酸(Ser)残基组成的长链。通过BLAST程序分析,采用CLUSTAW程序和MEGA 7.0软件对Chi2375进行序列比对和进化树构建[图 1(a)],Chi2375属于GH18家族的B亚家族。利用AlphaFold2模型构建Chi2375的三维结构,显示Chi2375的催化结构域为典型的(β/α)8 TIM结构,在β6与α6之间具有1个由2个β折叠和1个α螺旋组成的插入结构域,呈现GH18家族B亚家族的结构特征[图 1(b)]。保守域分析发现,Chi2375具有GH18家族几丁质酶中保守的几丁质结合序列SXGG和催化序列DXXDXDXE[图 1(c)]。
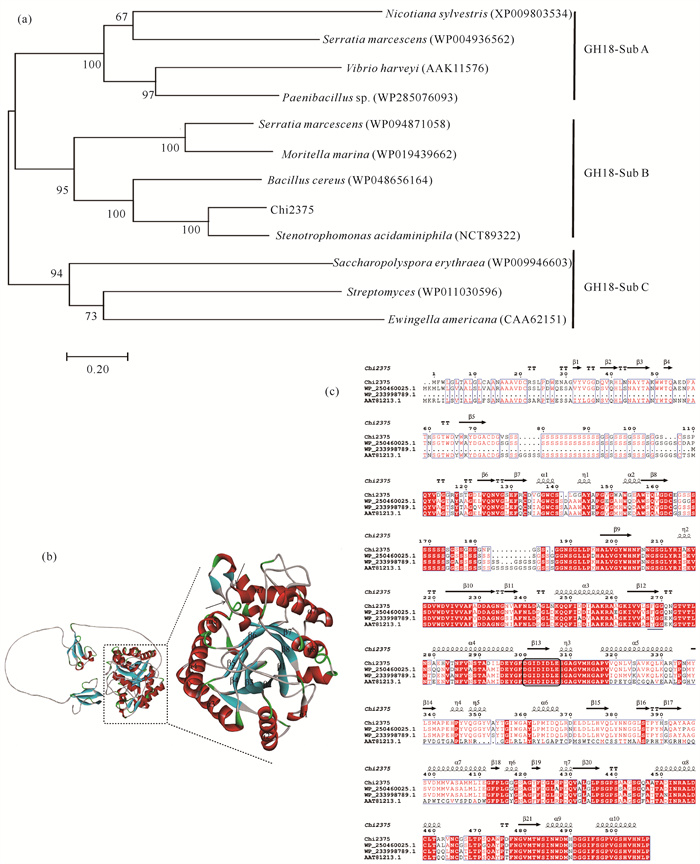
|
| (a) Molecular phylogenetic tree of chitinase Chi2375;(b) Protein structure of Chi2375;red indicates α-helix, blue indicates β-fold; (c) Alignment of putative chitinase sequences from Microbulbifer sp. GX H0434 (WP_250460025), M.hydrolyticus (AAT81213.1), M.pacificus (WP_233998789.1) and Chi2375 图 1 几丁质酶生物信息学分析 Fig. 1 Bioinformatics analysis of chitinase |
2.2 重组酶的诱导表达与纯化
将目的基因与表达载体连接构建pET30a- Chi2375重组表达质粒,并导入大肠杆菌表达,经镍柱纯化后,SDS-PAGE电泳结果表明,重组蛋白为单一条带,分子量大小约53 kDa,与理论分子量一致,可以满足下一步酶学性质的研究分析(图 2)。
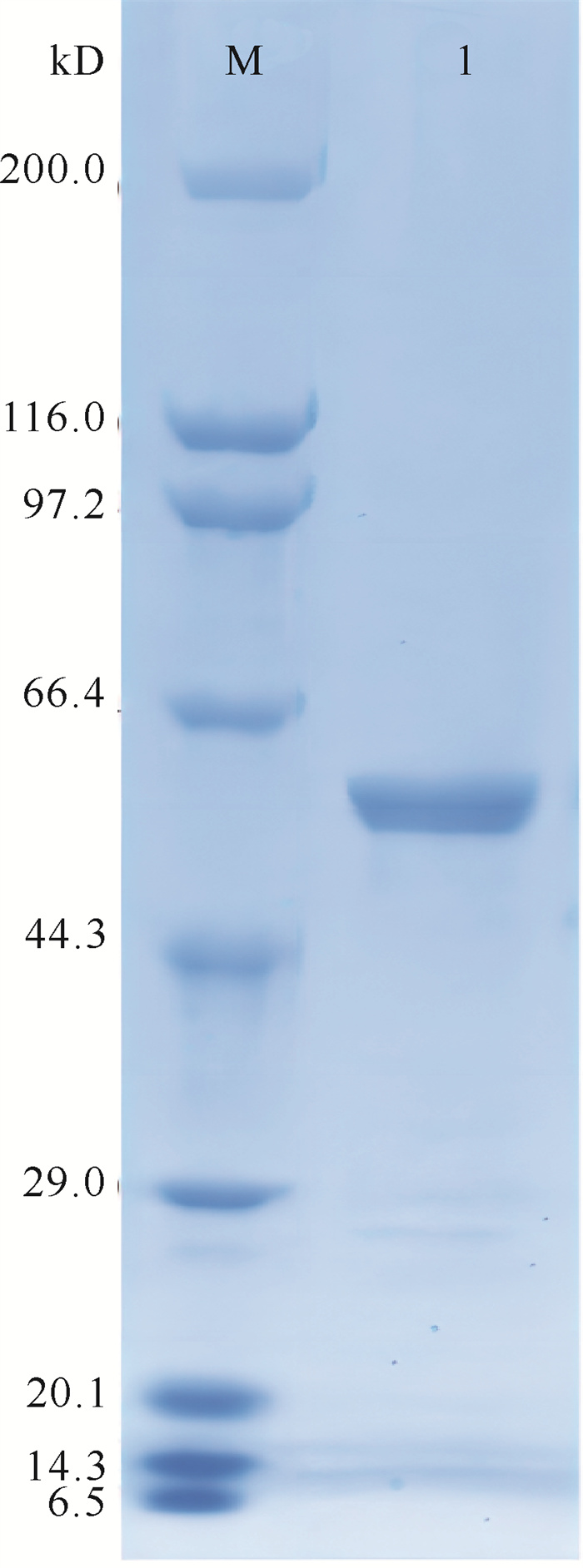
|
| M: molecular mass marker; 1:purified Chi2375. 图 2 重组酶Chi2375的SDS-PAGE分析 Fig. 2 SDS-PAGE analysis of recombinant Chi2375 |
2.3 重组酶的酶学性质分析 2.3.1 pH值和温度对重组酶Chi2375酶活力的影响
如图 3(a)所示,重组酶Chi2375的最适pH值为8.0;在pH值为6.5-9.0时,相对酶活力保持在80%以上;当pH值小于6.0或大于9.0时,相对酶活力下降至40%以下,表明重组酶Chi2375是一种偏碱性的酶。在pH值为3.5-12.0条件下,4 ℃保温24 h后重组酶Chi2375的相对酶活力保持在85%以上,表明重组酶Chi2375具有极高的pH耐受性。
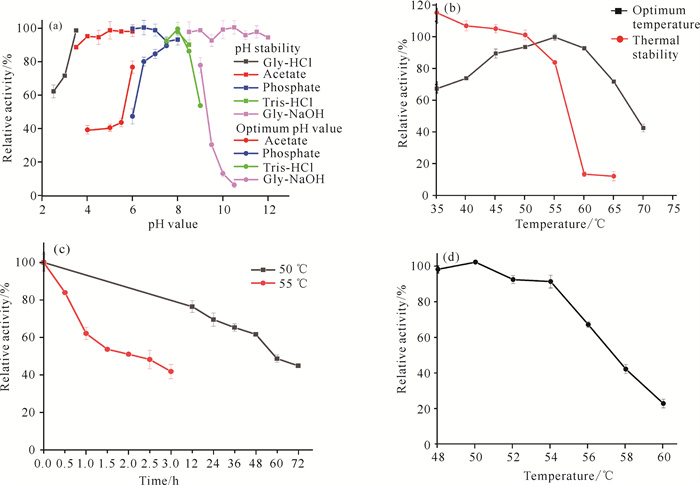
|
| (a) Optimum pH value and pH stability of recombinant Chi2375;(b) Optimum temperature and thermal stability of recombinant Chi2375;(c) Half-life of recombinant Chi2375 at 50 and 55℃; (d) Thermal stability of recombinant Chi2375 图 3 pH值和温度对重组酶Chi2375的影响 Fig. 3 Effects of pH value and temperature on recombinant Chi2375 |
如图 3(b)所示,重组酶Chi2375的最适温度为55 ℃;在45-60 ℃时,相对酶活力在80%以上;重组酶Chi2375在35-55 ℃温度范围孵育30 min后,相对酶活力在80%以上;但当温度为60 ℃时,相对酶活力则急剧下降至10%以下。如图 3(c)和图 3(d)所示,重组酶Chi2375在50、55 ℃的半衰期t1/2值分别为60和2 h,热半失活温度T5030为57 ℃,属于中等耐温酶。
2.3.2 重组酶Chi2375的底物特异性、Km和Vmax重组酶Chi2375对胶体几丁质的水解活力最高,对β-几丁质的水解活力是胶体几丁质的85%;以α-几丁质为底物时,重组酶酶活力很低,仅为胶体几丁质的1.5%;以壳聚糖为底物时,重组酶酶活力极低,约为胶体几丁质的0.5%;重组酶Chi2375对细粉几丁质和CMC-Na无水解能力(图 4)。以胶体几丁质为底物,利用软件Origin 2019,通过双倒数作图法拟合出重组酶Chi2375的Km和Vmax分别为34.19 mg·mL-1,7.491 U·mg-1(图 5)。
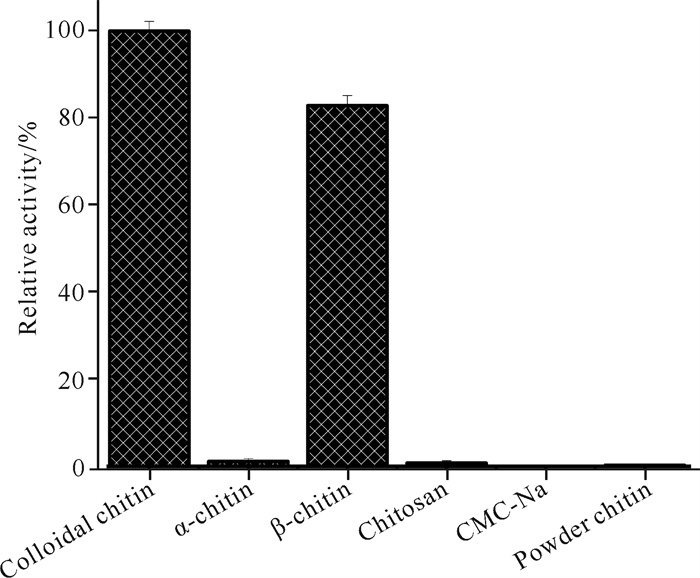
|
| 图 4 重组酶Chi2375的底物特异性测定 Fig. 4 Substrate specificity of recombinant Chi2375 |
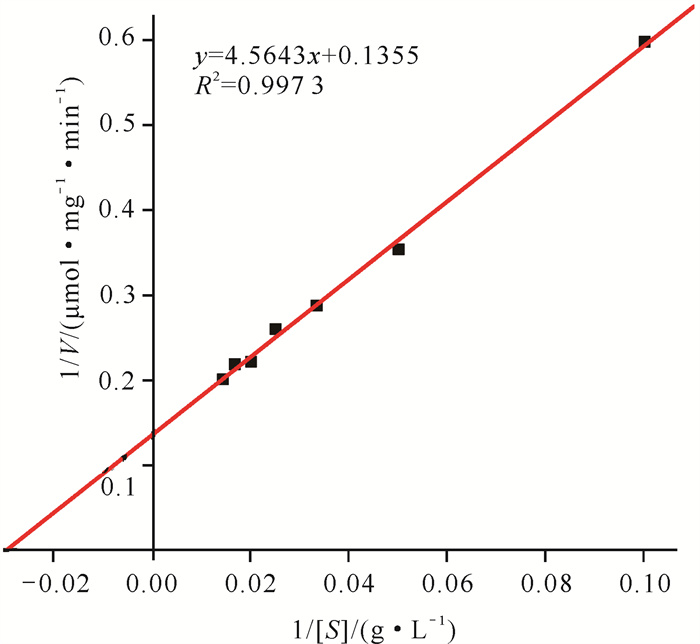
|
| 图 5 重组酶Chi2375的Km和Vmax Fig. 5 Km and Vmax of recombinant Chi2375 |
2.3.3 金属离子和有机试剂对重组酶Chi2375酶活力的影响
K +、Na+、Ca2+、Cu2+等金属离子对重组酶Chi2375有激活作用,Zn2+、Mn2+、Ni2+、Co2+、Fe2+、Fe3+等金属离子对重组酶Chi2375具有不同程度的抑制作用,Ba2+和Mg2+对重组酶Chi2375酶活力影响不明显[图 6(a)]。在离子浓度为1 mmol·L-1时,Cu2+和Ca2+对重组酶Chi2375的激活作用明显,相对酶活力均约为116%。当离子浓度为5 mmol·L-1时,Cu2+对Chi2375的激活作用增强,相对酶活力为140%;Ni2+和Fe3+对重组酶Chi2375的抑制作用明显,相对酶活力下降至20%以下。
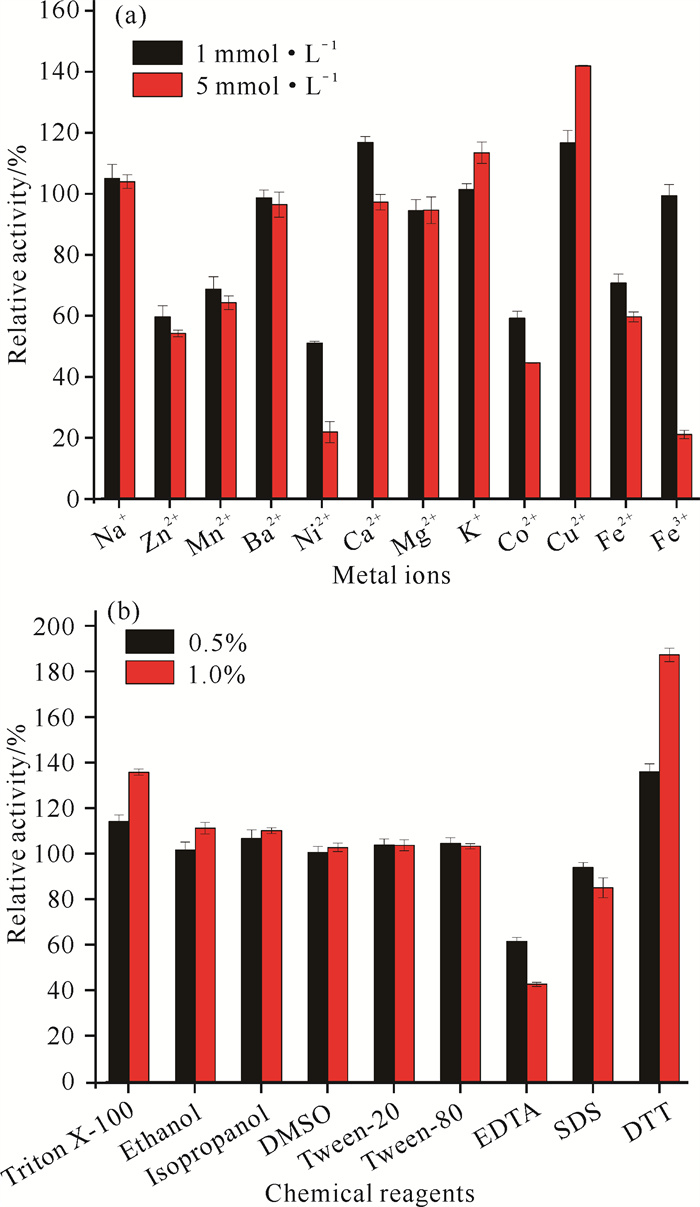
|
| (a) Effects of 1 and 5 mmol·L-1metal ions on the activity of recombinant Chi2375;(b) Effects of 0.5% and 1.0% chemical reagents on the activity of recombinant Chi2375 图 6 金属离子和有机试剂对重组酶Chi2375活力的影响 Fig. 6 Effects of metal ions and chemical reagents on recombinant Chi2375 |
如图 6(b)所示,0.5%的EDTA对重组酶Chi2375具有明显抑制效果,相对酶活力为59.4%;1.0%的EDTA和SDS抑制重组酶Chi2375的酶活力,相对酶活力分别为42.9%和85.2%;1.0%的Triton X-100和DTT对重组酶Chi2375有明显的激活作用,相对酶活力分别为135.7%和186.9%;乙醇、异丙醇、DMSO、吐温-20和吐温-80对重组酶Chi2375酶活力影响不大,相对酶活力维持在100%-110%。由此可见,重组酶Chi2375是一种对有机溶剂耐受的几丁质酶。
2.3.4 盐离子浓度对重组酶Chi2375酶活力的影响如图 7所示,0.5 mol·L-1 NaCl溶液对重组酶Chi2375酶活力无影响。在1.0 mol·L-1NaCl溶液处理时,重组酶Chi2375的相对酶活力在90%以上。在1.5-3.5 mol·L-1 NaCl处理下,重组酶Chi2375的相对酶活力仍保持在75%以上。
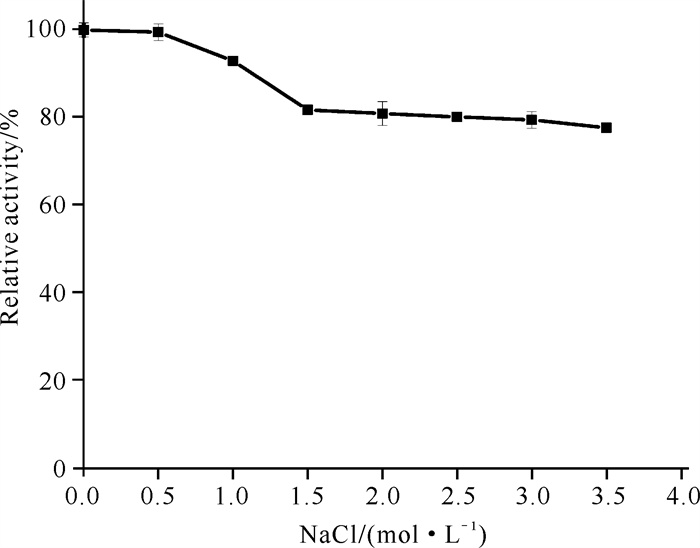
|
| 图 7 盐离子对重组酶Chi2375活力的影响 Fig. 7 Effects of NaCl concentration on stability of recombinant Chi2375 |
2.3.5 水解产物分析
采用分子排阻色谱法检测重组酶Chi2375降解胶体几丁质生成的产物,结果如图 8所示。不同反应时间(0.5-72 h)的终产物均以几丁二糖[(GlcNAc)2]为主(>90.0%),以及少量的几丁单糖(GlcNAc)(< 10.0%),说明重组酶Chi2375是一种外切几丁质酶。
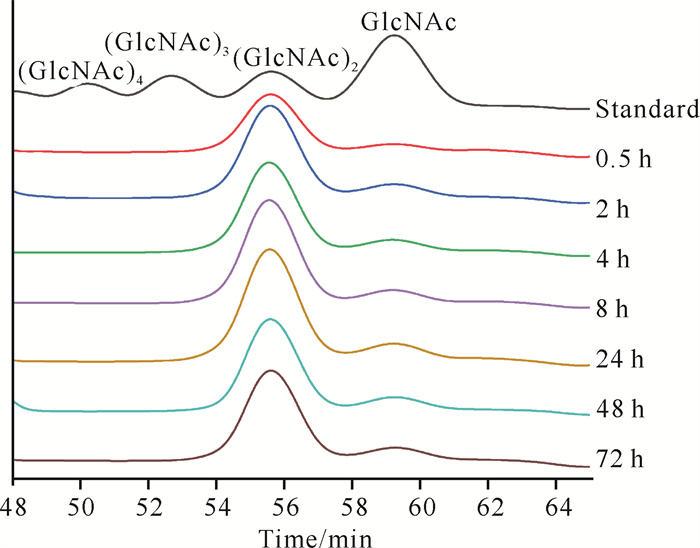
|
| 图 8 重组酶Chi2375水解胶体几丁质产物的HPLC图谱 Fig. 8 HPLC chromatograms of hydrolysis products of colloidal chitin by recombinant Chi2375 |
3 讨论
如今,随着几丁质酶的应用愈加广泛,对于其性能的研究需求也越来越大。重组酶Chi2375具有两个独立碳水化合物结合结构域(CBM),CBM有助于酶与几丁质底物的结合,增加酶与底物的亲和力,提高催化效率。
3.1 温度和pH值重组酶Chi2375为碱性几丁质酶,目前,有关碱性几丁质酶的报道较少,其中大多数几丁质酶为中性酶,最适pH值为7.0-8.0。例如,来自Aeromonas sp.SK10的几丁质酶Chi18A的最适pH值为8.0,来自Aeromonas sp.SK15的几丁质酶Chi18C最适pH值为7.5[22]。重组酶Chi2375具有较高的pH耐受性,在pH值为3.5-12.0的缓冲液中,于4 ℃条件下孵育24 h仍有90%以上的残余酶活力,与大部分的海洋几丁质酶在pH值为4.0-9.0中表现出较高稳定性的特性相一致[23, 24]。重组酶Chi2375的最适温度低于来自A.fumigatus的几丁质酶YJ-407(60 ℃)[25],但高于来自A.griseoaurantiacus的几丁质酶KX010988(40 ℃)[26]。细菌来源的几丁质酶在20-55 ℃具有较宽泛的温度耐受性。例如,来自转棘孢木霉Trichoderma asperellumd的几丁质酶Tachi1在35-50 ℃保温1 h后具有50%以上的酶活力[27];来自白长链霉菌Streptomyces albolongus ATCC 27414的几丁质酶SaChiA4在35-55 ℃保温24 h仍保持60%以上的酶活力[3];来自家蚕Bombyx mori的几丁质酶Silkworm在25-55 ℃保温1 h仍具有50%以上酶活力[28]。重组酶Chi2375在35-50 ℃孵育30 min具有100%以上的相对酶活力,在55 ℃孵育30 min仍具有约80%的相对酶活力,符合这一特性。
3.2 底物特异性与多数来源于GH18家族的几丁质酶不同,重组酶Chi2375除了能降解胶体几丁质外,对β-几丁质也具有较强的降解能力(酶活力是胶体几丁质的82.90%)。目前报道的几丁质酶主要是能降解α-几丁质,能降解β-几丁质的较少。
3.3 金属离子和有机试剂金属离子与底物活化或静电稳定有关,并通过稳定底物-酶复合物在酶催化中发挥作用[29]。5 mmol·L-1的Cu2+对重组酶Chi2375具有促进作用,酶活力提高了约1.4倍;而Zn2+、Mn2+、Ni2+、Co2+、Fe2+和Fe3+则对重组酶Chi2375的酶活力具有抑制作用,相对酶活力均不足65%;Na+、Ca2+、Ba2+、Mg2+和K+对Chi2375活力影响不明显。与重组酶Chi2375不同的是来自海洋弧菌Vibrio的几丁质酶Chi4668,在10 mmol·L-1的Ca2+和K+的作用下,其相对酶活力分别在140%和120%以上[30]。来自海洋烟曲霉Aspergillus fumigatus df673的几丁质酶Δ30AfChiJ,在10 mmol·L-1的Ca2+、Na+和K+的作用下,相对酶活力在120%左右[31]。来自耐热微泡菌M.thermotolerans DAU221的几丁质酶MtCh509(氨基酸序列未公开)对己烷、异戊醇和异丙醇等有机溶剂具有较强的耐受性[32],与重组酶Chi2375相似,但与作为碱性酶的重组酶Chi2375不同,MtCh509是酸性酶,其最适pH值为4.6-5.0。
3.4 盐离子浓度重组酶Chi2375对高盐离子浓度具有较好的耐受性,是一种耐盐的几丁质酶。耐盐酶对需要高盐条件的工业过程至关重要。海洋来源的酶对高盐浓度具有一定的耐受性,来自海洋细菌Pseudoalteromonas aurantia DSM6057的几丁质酶Chi23在4.5 mol·L-1 NaCl溶液中0 ℃孵育1 h后残余酶活力仍保留80%以上[33];来自海洋Aspergillus fumigatus df673的几丁质酶Δ30AfChiJ,在9%(浓度约为1.5 mol·L-1)的NaCl溶液中30 ℃孵育3 h后残余酶活力仍保留70%以上[31]。
3.5 水解产物分析重组酶Chi2375的降解产物主要为(GlcNAc)2,且产物组成不随反应时间的延长而变化,这一产物特征与几丁质外切酶以(GlcNAc)2为一个单元对底物进行切割的特性一致。与GH18家族A亚家族中的其他几丁质酶相似,如来自猪粪便环境的几丁质酶Echi47[34]、来自哈维弧菌Vibrio harveyi的几丁质酶Chi4668[30]作为典型的GH18家族A亚家族几丁质酶可以将几丁质降解为(GlcNAc)2。而有关GH18家族B亚家族的几丁质酶,目前报道的主要为内切几丁质酶,如来自海洋细菌Pseudoalteromonas aurantia DSM6057的几丁质酶Chi23,其水解胶体几丁质120 min的产物为(GlcNAc)2和(GlcNAc)3[33];来自海洋细菌Paenicibacillus barengoltzii的几丁质酶PbChi70,其水解胶体几丁质的产物最初为(GlcNAc)2、(GlcNAc)3和(GlcNAc)4,水解4 h后为GlcNAc和(GlcNAc)2[24]。与大多数GH18家族B亚家族几丁质酶不同的是,重组酶Chi2375属于外切酶。一些具有免疫活性的细胞表面存在(GlcNAc)2的残基受体,因此(GlcNAc)2可用于抗肿瘤及提高机体免疫力;此外,(GlcNAc)2还具有抑制α-糖苷酶的作用,可用于二型糖尿病的治疗。重组酶Chi2375可降解胶体几丁质得到纯度90%以上的(GlcNAc)2,因此重组酶Chi2375在医药领域具有广阔的应用前景。
4 结论本研究获得的重组酶Chi2375,基因全长1 521 bp,编码507个氨基酸,属于GH18家族,具有几丁质酶的保守催化序列DXXDXDXE。重组酶Chi2375的最适反应温度为55 ℃,T5030值约为57 ℃,最适反应pH值为8.0,pH值为6.5-9.0时相对酶活力在80%以上,为中等耐温碱性几丁质酶。金属离子Ca2+、K+、Cu2+及有机试剂Triton X-100、DTT对该酶活力具有促进作用,而Zn2+、Mn2+、Ni2+、Co2+、Fe2+、Fe3+和EDTA对该酶活力具有明显抑制作用。用胶体几丁质作为底物时重组酶Chi2375的酶活力最高,其水解产物主要为(GlcNAc)2。重组酶Chi2375属于GH18家族B亚家族外切几丁质酶,且对β-几丁质具有降解活力,是一种新型的几丁质酶。在后续的研究中,本课题组将开展重组酶Chi2375的结构功能研究,阐明其作用机制,进一步挖掘其潜能,为提高该酶对海洋甲壳类废弃物的综合利用水平奠定理论基础。
| [1] |
RAY L, PANDA A N, MISHRA S R, et al. Purification and characterization of an extracellular thermo-alkali stable, metal tolerant chitinase from Streptomyces chilikensis RC1830 isolated from a brackish water lake sediment[J]. Biotechnology Reports, 2019, 21: e00311. DOI:10.1016/j.btre.2019.e00311 |
| [2] |
PAN M Y, LI J H, LV X Q, et al. Molecular engineering of chitinase from Bacillus sp.DAU101 for enzymatic production of chitooligosaccharides[J]. Enzyme and Microbial Technology, 2019, 124: 54-62. DOI:10.1016/j.enzmictec.2019.01.012 |
| [3] |
GAO L, SUN J N, SECUNDO F, et al. Cloning, characterization and substrate degradation mode of a novel chitinase from Streptomyces albolongus ATCC 27414[J]. Food Chemistry, 2018, 261: 329-336. DOI:10.1016/j.foodchem.2018.04.068 |
| [4] |
LV J R, ZHANG Y M, MA M H, et al. Characterization of chitinase from Exiguobacterium antarcticum and its bioconversion of crayfish shell into chitin oligosaccharides[J]. Food Research International, 2022, 158: 111517. DOI:10.1016/j.foodres.2022.111517 |
| [5] |
HOWARD M B, EKBORG N A, WEINER R M, et al. Detection and characterization of chitinases and other chitin-modifying enzymes[J]. Journal of Industrial Microbiology & Biotechnology, 2003, 30(11): 627-635. |
| [6] |
刘邮洲, 罗楚平, 刘永锋, 等. 黏质沙雷氏菌C8-8几丁质酶基因的克隆与表达[J]. 江苏农业学报, 2012, 28(3): 487-491. DOI:10.3969/j.issn.1000-4440.2012.03.006 |
| [7] |
高兆建, 丁飞鸿, 陈欢, 等. 产黄青霉抗真菌几丁质酶的纯化及特性分析[J]. 食品科学, 2021, 42(14): 129-136. |
| [8] |
王琳, 陈雅如, 程湄婕, 等. 微生物几丁质酶研究进展及应用[J]. 中国生物工程杂志, 2022, 42(12): 101-110. |
| [9] |
ZHOU J, CHEN J H, XU N, et al. The broad-specificity chitinases: their origin, characterization, and potential application[J]. Applied Microbiology and Biotechnology, 2019, 103(8): 3289-3295. DOI:10.1007/s00253-019-09718-x |
| [10] |
LI Z K, WANG T, LUO X, et al. Biocontrol potential of Myxococcus sp. strain BS against bacterial soft rot of calla lily caused by Pectobacterium carotovorum[J]. Biological Control, 2018, 126: 63-44. |
| [11] |
郑家敏. 几丁质酶高产菌株选育及酶学性质研究[D]. 福州: 福州大学, 2018.
|
| [12] |
刘朋, 叶波平. 微生物几丁质酶研究进展及应用现状[J]. 科学技术创新, 2018(12): 52-53. |
| [13] |
OYELEYE A, NORMI Y M. Chitinase: diversity, limitations, and trends in engineering for suitable applications[J]. Bioscience Reports, 2018, 38(4): BSR2018032300. |
| [14] |
赵沙, 颜子娟, 张舒, 等. 细菌几丁质酶结构、功能及分子设计的研究进展[J]. 生物化学与生物物理进展, 2022, 49(7): 1179-1191. |
| [15] |
刘力睿, 潘杰, 李猛. 微生物几丁质酶的研究进展、应用及展望[J]. 生物资源, 2020, 42(5): 494-504. |
| [16] |
LI H, GREENE L H. Sequence and structural analysis of the chitinase insertion domain reveals two conserved motifs involved in chitin-binding[J]. PLoS One, 2010, 5(1): e8654. |
| [17] |
SUN X M, LI Y J, TIAN Z N, et al. A novel thermostable chitinolytic machinery of Streptomyces sp. F-3 consisting of chitinases with different action modes[J]. Biotechnology for Biofuels, 2019, 12: 136. |
| [18] |
DAS S, DEY P, ROY D, et al. N-acetyl-D-glucosamine production by a chitinase of marine fungal origin: a case study of potential industrial significance for valorization of waste chitins[J]. Applied Biochemistry and Biotechnology, 2019, 187(1): 407-423. |
| [19] |
LIU C, SHEN N K, WU J F, et al. Cloning, expression and characterization of a chitinase from Paenibacillus chitinolyticus strain UMBR0002[J]. PeerJ, 2020, 8: e8964. |
| [20] |
郝之奎. Chitinolyticbacter meiyuanensis的筛选鉴定及其发酵产几丁质酶研究[D]. 无锡: 江南大学, 2011.
|
| [21] |
MILLER G L. Use of dinitrosalicylic acid reagent for determination of reducing sugar[J]. Analytical Chemistry, 1959, 31(3): 426-428. |
| [22] |
KIM S, PARK J, OH J, et al. Molecular characteriza-tion of four alkaline chitinases from three chitinolytic bacteria isolated from a mudflat[J]. International Journal of Molecular Sciences, 2021, 22(23): 12822. |
| [23] |
LI R K, HU Y J, HE Y J, et al. A thermophilic chitinase 1602 from the marine bacterium Microbulbifer sp. BN3 and its high-level expression in Pichia pastoris[J]. Biotechnology and Applied Biochemistry, 2021, 68(5): 1076-1085. |
| [24] |
YANG S Q, FU X, YAN Q J, et al. Cloning, expres-sion, purification and application of a novel chitinase from a thermophilic marine bacterium Paenibacillus barengoltzii[J]. Food Chemistry, 2016, 192: 1041-1048. |
| [25] |
XIA G Q, JIN C S, ZHOU J, et al. A novel chitinase having a unique mode of action from Aspergillus fumigatus YJ-407[J]. European Journal of Biochemistry, 2001, 268(14): 4079-4085. |
| [26] |
SHEHATA A N, ABD EL ATY A A, DARWISH D A, et al. Purification, physicochemical and thermodynamic studies of antifungal chitinase with production of bioactive chitosan-oligosaccharide from newly isolated Aspergillus griseoaurantiacus KX010988[J]. International Journal of Biological Macromolecules, 2018, 107(Pt A): 990-999. |
| [27] |
汤伟, 李雅华, 刘露, 等. 重组毕赤酵母表达棘孢木霉几丁质酶Tachi1的酶学性质研究及表达条件优化[J]. 微生物学报, 2012, 52(3): 345-352. |
| [28] |
ZHANG H B, LIU M Y, TIAN Y J, et al. Comparative characterization of chitinases from silkworm (Bombyx mori) and bollworm (Helicoverpa armigera)[J]. Cell Biochemistry and Biophysics, 2011, 61(2): 267-275. |
| [29] |
ANDREINI C, BERTINI I, CAVALLARO G, et al. Metal ions in biological catalysis: from enzyme databases to general principles[J]. JBIC Journal of Biological Inorganic Chemistry, 2008, 13(8): 1205-1218. |
| [30] |
HE X X, YU M, WU Y H, et al. Two highly similar chitinases from marine Vibrio species have different enzymatic properties[J]. Marine Drugs, 2020, 18(3): 139. |
| [31] |
HE B, YANG L Y, YANG D F, et al. Biochemical purification and characterization of a truncated acidic, thermostable chitinase from marine fungus for N-acetylglucosamine production[J]. Frontiers in Bioengineering and Biotechnology, 2022, 10: 1013313. |
| [32] |
LEE H, LEE Y, CHOI Y. Cloning, purification, and characterization of an organic solvent-tolerant chitinase, MtCh509, from Microbulbifer thermotolerans DAU221[J]. Biotechnology for Biofuels, 2018, 11: 303. |
| [33] |
WANG Y J, JIANG W X, ZHANG Y S, et al. Structural insight into chitin degradation and thermostability of a novel endochitinase from the glycoside hydrolase family 18[J]. Frontiers in Microbiology, 2019, 10: 2457. |
| [34] |
LIU Y C, YAN Q J, YANG S Q, et al. Novel protease-resistant exochitinase (Echi47) from pig fecal environment DNA with application potentials in the food and feed industries[J]. Journal of Agricultural and Food Chemistry, 2015, 63(27): 6262-6270. |



