小花老鼠簕(Acanthus ebracteatus)生长在潮间带,该地区是海洋生态系统和陆地生态系统之间的过渡带,不仅具有调节气候、抗风消浪[1]、维持生物多样性[2]及碳汇[3]等生态功能,还具有提供旅游、渔业及土地资源的经济价值[4]。小花老鼠簕天然分布于印度、中南半岛、印度尼西亚等地。我国的广西北仑河口,海南海口、儋州、万宁、琼海、文昌及广东广州、湛江有明确分布记录[5-8]。小花老鼠簕是我国真红树植物之一,属爵床科(Acanthaceae)老鼠簕属(Acanthus),别名黄鱼簕、海簕根等。小花老鼠簕生境与老鼠簕(A.ilicifolius)类似,一般生长于潮沟两侧、红树林内缘,有时也形成小面积纯林[7]。根据世界自然保护联盟(International Union for Conservation of Nature and Natural Resources,IUCN)的标准,小花老鼠簕在我国属于濒危(Endangered, EN)等级[7]。小花老鼠簕近年来在部分自然栖息地出现灭绝现象,如海南三亚的小花老鼠簕[5]。
分子标记技术是分子生物学领域一项重要的研究技术[9]。植物的简单重复序列(Simple Sequence Repeat, SSR)可能在人工选择和环境压力下变短,所以可以通过多倍化和杂交诱导微卫星的基因发生变异,增加植物对环境的适应能力[10]。白晓倩等[11]利用SSR标记及表型性状构建板栗品种的分子身份证,研究板栗品种的遗传多样性,为板栗种质资源的品种鉴定、合理利用及保护提供理论依据。Ha等[12]通过对38份紫苏(Perilla frutescens)种质29个SSR标记和3个种子性状的关联分析,检测到6个与种子萌发率(Seed Germination Rate, SGR)相关的SSR标记、8个与种子硬度(Seed Hardness, SH)相关的标记、7个与种子大小(Seed Size, SS)相关的SSR标记。Li等[13]基于SSR标记基因型数据及形态性状的表型数据得到遗传距离的聚类分析,将127份大蒜(Allium sativum)材料分为3个聚类。虽然SSR技术在国内外有很多研究及应用,但是在爵床科(Acanthaceae)植物遗传多样性方面的相关研究却不多。公培民等[14]利用Illumina HiSeqTM2000对马蓝(Baphicacanthus cusia)转录组进行高通量测序,使用软件MIcroSAtellite(MISA)分析转录组中的SSR位点信息,发现马蓝SSR出现的频率高,重复种类丰富,为研究马蓝品质改良、基因定位及遗传多样性提供了科学依据。李俊仁等[15]通过基因功能注释发现,穿心莲(Andrographis paniculata)含SSR的Unigene(广泛通用的基因数据库)主要与基础代谢功能相关。
微卫星标记在红树植物遗传多样性研究中得到了广泛的研究和应用。Geng等[16]利用核微卫星标记和叶绿体微卫星标记对华南沿海地区占主导地位的7个红树物种进行了基因分型,估计了这7个红树物种的历史和当代基因流、遗传多样性、种群结构,结果表明这7个物种都表现出很少的单倍型、低水平的遗传多样性和高水平的近亲繁殖,这可能是由于它们的边缘地理分布、人类驱动、自然压力导致的栖息地丧失和破碎化。Cerón-souza等[17]用11个微卫星位点分析了980株亮叶白骨壤(Avicennia germinans),并用6个微卫星位点分析了940株美洲红树(Rhizophora mangle),并对两个非编码cpDNA区域进行了二次采样,以了解巴拿马加勒比海和太平洋海岸4个主要河口区域这两个物种的种群结构和基因流,发现红树林中的基因传播通常局限于河口,沿海地貌和罕见的远距离传播事件也会影响其结构水平。阮宇等[18]通过SSR标记,利用近似贝氏计算(Approximate Bayesian Computation,ABC)推测海峡两岸4个分布区域秋茄的起源及其拓殖路线,得出海峡两岸秋茄的拓殖路线是从大陆东南的南方种群随黑潮迁移至台湾北部,再从台湾北部拓殖到台湾南部的结论。Millán-aguilar等[19]在墨西哥锡那罗亚州3个潟湖系统研究了亮叶白骨壤的遗传多样性和精细遗传结构,发现受干扰和保留的地点之间的遗传多样性和总体遗传结构相似,然而却观察到了较低水平的精细尺度空间遗传结构,并将此归因于每个潟湖系统的局部动态、其保护状态和碎片化程度的差异,此外,低连通性和干扰的影响可能会限制传粉者的移动和种子的传播能力,导致遗传多样性水平低、出现近亲繁殖的现象。Hu等[20]通过对银叶树(Heritiera littoralis)的转录组分析,鉴定了1 393个单核苷酸多态性(SNP)和433个SSR标记,其中36个SNP标记中的19个通过裂解扩增多态性序列(Cleaved Amplified Polymorphic Sequences, CAPS)得到了验证,24个SSR标记中的16个通过荧光毛细管电泳得到了确认。但目前仍缺乏对小花老鼠簕遗传多样性的研究。
本研究拟选择9对多态微卫星标记,分析广东广州、广东湛江、广西防城港及海南文昌的小花老鼠簕的遗传多样性,为小花老鼠簕遗传多样性保护及创新利用提供理论依据。
1 材料与方法 1.1 材料 1.1.1 植物材料对广东广州(GDGZ: Guangzhou, Guangdong;22°54′36.38″N, 113°32′52.40″E)、广东湛江(GDZJ: Zhanjiang, Guangdong; 21°27′43.70″N, 109°58′25.26″E)、广西防城港(GXFC: Fangchenggang, Guangxi; 21°30′59.30″N,108°13′36.09″E)及海南文昌(HNWC: Wenchang, Hainan; 19°37′40.29″N,110°47′13.71″E)的小花老鼠簕进行采样。小花老鼠簕形态如图 1所示。

|
| 图 1 小花老鼠簕形态 Fig. 1 Morphology of Acanthus ebracteatus |
1.1.2 试剂
试验所用主要试剂为植物基因组DNA提取试剂盒(DP305,天根生化科技有限公司);DL2000 DNA Marker(TSJ011-100,北京擎科生物科技股份有限公司);LIZ标记分子量内标(美国ABI公司);Taq酶(加拿大Fermentas公司);DEPC水(深圳市吉真科技有限公司);脱氧核糖核苷三磷酸(dNTP,加拿大Fermentas公司)。
1.1.3 仪器试验所用主要仪器设备为高通量组织研磨机(SLGT-48,南京中科科学器材有限公司),电热恒温水浴锅(DK-S24,上海精宏实验设备有限公司),涡旋振荡器(G560E,美国Thermo Fisher Scientific公司),高速冷冻离心机(iCEM-24R,杭州奥盛仪器有限公司),超微量分光光度计(OD-1000+,美国One Drop公司),电泳仪(JY300C,北京君意东方电泳设备有限公司),凝胶成像仪(Tanon-1600,上海天能生命科学有限公司),移液器(Research plus,德国Eppendorf公司),PCR仪(黑金刚EDC-810,北京东胜创新生物科技有限公司),基因测序仪(3730XL,美国ABI公司),台式高速冷冻离心机(Allegra 25R,美国Beckman公司),微量台式离心机(Micro 17R,美国Thermo Fisher Scientific公司),电热恒温水槽(DK-8B,上海精宏实验设备有限公司),立式压力蒸汽灭菌器(YXQ-LS-50SII,上海博迅实业有限公司医疗设备厂)。
1.2 方法 1.2.1 样品采集在每个地点对40个个体进行采样,共计160个个体。采样间隔至少10 m,采集部位为幼叶。所有样品立即放入装有冰袋的泡沫箱中带回实验室,并在-80 ℃下储存。用干冰运输到南京集思慧远生物科技有限公司进行STR遗传标记检测分析。
1.2.2 核和叶绿体微卫星基因分型用植物基因组DNA提取试剂盒提取样品总DNA,并在-80 ℃下保存。
为进行微卫星分析,用6个多态核微卫星(nSSR)标记以及3个叶绿体微卫星(cpSSR)标记对小花老鼠簕的样本进行基因分型。所用的nSSR标记为Acil13、Acai02、Acai08、Acai11、Acai12、Acai13[21],cpSSR标记为Acaicp02、Acaicp03、Acaicp04[22](表 1)。PCR扩增条件如下:15 μL PCR反应体系含有1 μL DNA、1×缓冲液、2.5 mmol/L Mg2+、0.2 mmol/L dNTP、1 μmol/L引物和1.5 U Taq DNA聚合酶。PCR条件为94 ℃ 3 min,35个循环(94 ℃ 15 s,55 ℃ 15 s,72 ℃ 30 s),72 ℃延伸3 min。
| 位点 Locus |
引物序列(5′→3′) Primer sequence (5′→3′) |
Tm/℃ | 荧光染料 Fluorescent dye |
| Acil13 | ATAATTTATCATGGCCATCG AATGTGGAGAATTTGGCATCTGGTGCG | 55 | FAM |
| Acai02 | ACACACACACACTCTCTCTCTC CCATCTGAAAATAGATGGTAGGAC | 56 | HEX |
| Acai08 | TCTCTCTCTCTCACACACACAC GAGGTACTAGGGTTTCAACATGAT | 56 | TAMRA |
| Acai11 | TCTCTCTCTCTCACACACACAC CCCCCATATTTGTATATGTTTG | 52 | TAMRA |
| Acai12 | TCTCTCTCTCTCACACACACAC CCACCAATGGTGCTGAATACATTG | 58 | TAMRA |
| Acai13 | TCTCTCTCTCTCACACACACAC CCGACAAGCTCCTCCAGTATTTTG | 60 | TAMRA |
| Acaicp02 | GCATTTACCTATTATAGAGA CGTAAAACCGAGAGAAATTC | 48 | HEX |
| Acaicp03 | CAGAGAAGCCTATGTATGAA AAATGAAGCACTCCAGATAC | 52 | FAM |
| Acaicp04 | CATAGACAGCGTAATCTAAA AGATGCTGTCTTGCTAAGAC | 50 | HEX |
将含有12.5 μL超纯去离子甲酰胺、0.25 μL ROX标记的分子量标记和1.5 μL 10倍稀释的PCR产物的待测样品,置于基因测序仪样本架上检测。
1.2.3 遗传多样性指标统计 1.2.3.1 单位点遗传多样性指标统计SSR分型数据根据各软件格式要求进行转换。使用GenAlEx 6.51b2软件计算单个SSR位点在样本整体的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)和Shannon′s多样性指数(I)。使用Popgene 1.32软件计算各位点近交系数(Fis)、各位点分化系数(Fst)、各位点平均近交系数(Fit)。使用Powermarker 3.25软件中的Summary Statistics功能计算各位点的多态性信息含量(PIC)和基因多样性(Gene diversity),使用GenePOP 4.7软件计算无效等位基因频率(Fna)。多态性信息是等位基因频率和等位基因数的变化函数。多态性信息含量越高,在一个群体中,该座位上杂合子比例则越大,提供的遗传信息就越高。Botstein等[23]提出了衡量基因变异程度高低的多态性信息指标,当PIC>0.50时,该位点为高度多态位点;当0.25 < PIC < 0.50时,该位点为中度多态位点;当PIC < 0.25时,为低度多态位点。
1.2.3.2 居群遗传多样性指标统计利用GenAlEx 6.51b2软件分析每个群体的等位基因数、有效等位基因数、观测杂合度、期望杂合度、Shannon′s多样性指数、群体内近交系数(F)。
1.2.3.3 Jaccard遗传相似系数矩阵计算使用Ntsys 2.10e软件中的Qualitative data功能计算Jaccard遗传相似系数(Jaccard similarity coefficient);基于Jaccard遗传相似系数,使用软件中的Clustering功能下的SAHN进行非加权类平均聚类法(UPGMA)聚类;使用软件中的Graphics功能下的Tree plot功能进行绘图。
1.2.4 聚类分析 1.2.4.1 个体聚类分析个体聚类分析使用PowerMarker 3.2.5软件中的Compute Frequency功能计算各样本下各标记的等位基因频率,然后使用Frequency Based Distance功能基于等位基因频率计算Nei′s(1983)遗传距离,最后使用软件的UPGMA/NJ Tree功能进行UPGMA和邻位相连聚类法(Neighbor-Joining clustering)两种方法绘制聚类树,并使用MEGA 7.0软件对聚类树进行编辑美化。
1.2.4.2 群体聚类分析群体聚类分析使用PowerMarker 3.2.5软件计算基于等位基因频率的Nei′s(1983)遗传距离,使用邻位相连聚类法和UPGMA两种方法绘制聚类树。
1.2.5 分子遗传变异方差分析(AMOVA)利用GenAlEx 6.51b2软件进行分子遗传变异方差分析,计算总体群体间和群体内的分子遗传变异平方和(SS)、均方(MS)、变异方差(EST.Var.),并设置999次permutation。
1.2.6 群体结构分析及二维主坐标分析利用Structure软件分析来自广东广州、广东湛江、广西防城港及海南文昌4个群体的160个小花老鼠簕基因型的群体结构。利用GenALEX 6.51b2软件计算遗传距离矩阵,基于GD矩阵进行二维主坐标分析(Principal Co-ordinates Analysis,PCoA),并绘制主坐标1和主坐标2的PCoA散点图,并将预设群体用不同颜色在图中标注,导出前10个主成分对应的PCoA矩阵。
1.2.7 哈迪温伯格检测利用Genepop 4.7软件进行各群体和总体标记的哈迪温伯格检测和无效等位基因分析。
2 结果与分析 2.1 微卫星位点分析从表 2可以看出,9个微卫星位点一共包含61个等位基因,每个位点的平均等位基因数是6.778,等位基因数的范围从2(Acaicp02)到14(Acil13)。Shannon′s多样性指数为0.304(Acaicp04)-2.067(Acai12)。观测杂合度在0.000(Acaicp02, Acaicp03, Acaicp04)和0.744(Acil13)之间。期望杂合度的取值在0.150(Acaicp04)和0.840(Acai12)之间,平均值为0.638。多态性信息含量在0.140 7(Acaicp04)和0.823 0(Acai12)之间。6个核微卫星位点(Acil13、Acai02、Acai08、Acai11、Acai12、Acai13)的多态性信息含量均大于0.50,都呈现出高度多态性。因此,这些位点可以提供的遗传信息相对较多。小花老鼠簕的cpSSR遗传多样性处于中低水平,Acaicp02 (PIC=0.374 0)和Acaicp03 (PIC=0.467 3)为中度多态位点,Acaicp04(PIC=0.140 7)为低度多态位点。
| 位点Locus | 等位基因数Na | 有效等位基因数Ne | Shannon′s多样性指数I | 观测杂合度Ho | 期望杂合度He | 各位点近交系数Fis | 各位点平均近交系数Fit | 各位点分化系数Fst | 多态性信息含量PIC | 基因多样性Gene diversity | 无效等位基因频率Fna | 基因流Nm |
| Acil13 | 14 | 6.204 | 2.005 | 0.744 | 0.839 | -0.484 | 0.113 | 0.403 | 0.819 4 | 0.838 8 | 0.051 8 | 0.371 |
| Acai02 | 9 | 3.378 | 1.543 | 0.644 | 0.704 | -0.281 | 0.085 | 0.286 | 0.672 8 | 0.703 9 | 0.056 4 | 0.624 |
| Acai08 | 6 | 3.556 | 1.421 | 0.688 | 0.719 | -0.060 | 0.044 | 0.098 | 0.670 4 | 0.718 8 | 0.059 8 | 2.313 |
| Acai11 | 6 | 3.442 | 1.399 | 0.400 | 0.710 | -0.239 | 0.436 | 0.545 | 0.660 3 | 0.709 5 | 0.473 6 | 0.209 |
| Acai12 | 12 | 6.248 | 2.067 | 0.681 | 0.840 | -0.120 | 0.189 | 0.276 | 0.823 0 | 0.839 9 | 0.176 2 | 0.656 |
| Acai13 | 6 | 3.571 | 1.386 | 0.406 | 0.720 | -0.348 | 0.436 | 0.581 | 0.671 5 | 0.720 0 | 0.201 5 | 0.180 |
| Acaicp02 | 2 | 1.992 | 0.691 | 0.000 | 0.498 | 1.000 | 1.000 | 0.117 | 0.374 0 | 0.498 0 | 0.684 7 | 1.884 |
| Acaicp03 | 3 | 2.281 | 0.902 | 0.000 | 0.562 | 1.000 | 1.000 | 0.077 | 0.467 3 | 0.561 6 | 0.726 6 | 2.982 |
| Acaicp04 | 3 | 1.177 | 0.304 | 0.000 | 0.150 | 1.000 | 1.000 | 0.089 | 0.140 7 | 0.150 2 | 0.958 6 | 2.561 |
| Mean | 6.778 | 3.539 | 1.302 | 0.396 | 0.638 | 0.163 | 0.478 | 0.275 | 0.588 8 | 0.637 9 | 0.376 6 | 1.309 |
2.2 遗传多样性分析
遗传多样性评估结果(表 3)显示,各群体等位基因数范围为2.444(GDGZ)到4.111(HNWC), 平均值为3.167。有效等位基因数为1.881-2.262。Shannon′s多样性指数最低为0.632(GDGZ),最高为0.851(GDZJ),观测杂合度和期望杂合度的平均值分别为0.396和0.442。
| 群体Popu- lation | 等位基因数Na | 有效等位基因数Ne | Shan- non′s多样性指数I | 观测杂合度Ho | 期望杂合度He |
| GDGZ | 2.444 | 1.881 | 0.632 | 0.453 | 0.399 |
| GDZJ | 2.889 | 2.262 | 0.851 | 0.667 | 0.522 |
| GXFC | 3.222 | 2.003 | 0.763 | 0.186 | 0.443 |
| HNWC | 4.111 | 2.062 | 0.754 | 0.278 | 0.405 |
| Mean | 3.167 | 2.052 | 0.750 | 0.396 | 0.442 |
| Note: GDGZ indicates Guangzhou, Guangdong; GDZJ indicates Zhanjiang, Guangdong; GXFC indicates Fangchenggang, Guangxi; HNWC indicates Wenchang, Hainan. | |||||
2.3 聚类及遗传结构分析
利用PowerMarker 3.2.5软件,基于Nei′s遗传距离对4个群体构建UPGMA树(图 2),聚类结果显示4个群体被分为两大类,广东广州单独聚为一类,广东湛江、广西防城港和海南文昌聚为一类。第二大类中,广西防城港和海南文昌先聚为一类,再与广东湛江聚为一类。
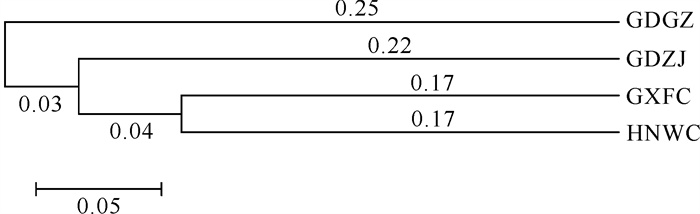
|
| GDGZ: Guangzhou, Guangdong; GDZJ: Zhanjiang, Guangdong; GXFC: Fangchenggang, Guangxi; HNWC: Wenchang, Hainan. 图 2 基于UPGMA的小花老鼠簕4个群体的遗传聚类图 Fig. 2 Dendrogram of 4 A.ebracteatus populations based on UPGMA |
2.4 群体间的遗传分化与群体结构估计
4个群体的SSR数据的分级AMOVA显示,4个小花老鼠簕群体间变异为36%,群体内变异为64%,遗传变异主要来自群体内不同的个体间(表 4)。
| 变异来源 Source of variation |
自由度 Degree of freedom |
离差平方和 Sum of squares |
均方 MS |
估计方差 Estimated variation |
变异百分比/% Percentage of variation/% |
| Among populations | 3 | 281.863 | 93.954 | 1.149 | 36 |
| Within populations | 316 | 636.663 | 2.015 | 2.015 | 64 |
| Total | 319 | 918.526 | 3.164 | 100 |
2.5 群体结构分析
由于Structure软件在K=4时能较好地分为4个主要色块,表明最优K值为4。海南文昌的色块相较于其他群体的色块底部更复杂一些(图 3),表明海南文昌的小花老鼠簕遗传多样性最高。由于海南文昌小花老鼠簕的等位基因数(4.111)高于其他群体(表 3),结合等位基因数及Structure软件分析,证明海南文昌的小花老鼠簕遗传多样性最高。
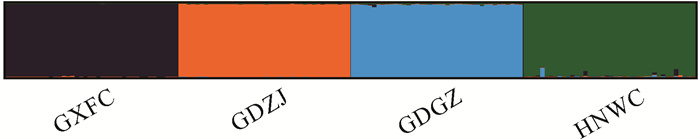
|
| GDGZ: Guangzhou, Guangdong; GDZJ: Zhanjiang, Guangdong; GXFC: Fangchenggang, Guangxi; HNWC: Wenchang, Hainan 图 3 160个小花老鼠簕基因型的群体结构 Fig. 3 Population structure of 160 genotypes of A.ebracteatus |
2.6 二维主坐标分析
利用GenALEX 6.51b2软件对160份样本进行主成分分析(图 4),进一步验证Structure的群体分析结果,主坐标1和主坐标2分别揭示总变异的21.44%和16.95%。其结果与UPGMA树基本一致(图 4),GDGZ与其他群体之间有明显的遗传分化,其余3个群体之间较接近。
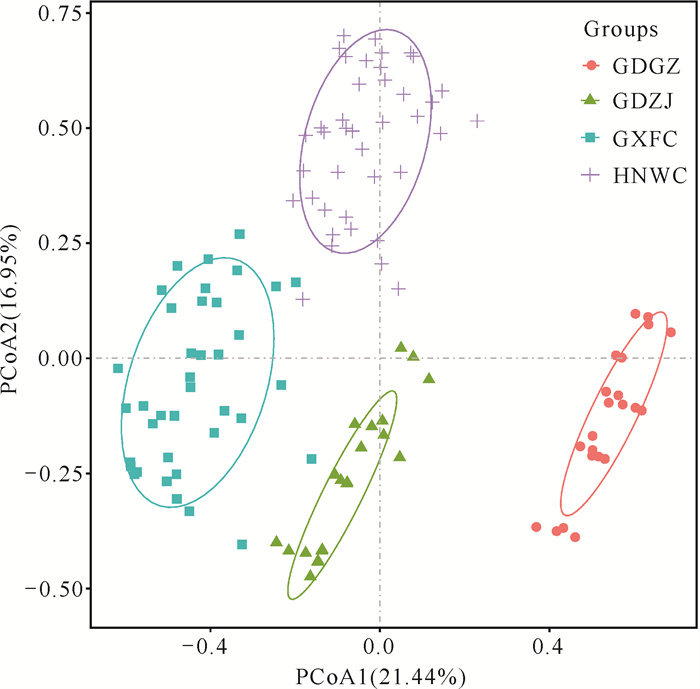
|
| GDGZ: Guangzhou, Guangdong; GDZJ: Zhanjiang, Guangdong; GXFC: Fangchenggang, Guangxi; HNWC: Wenchang, Hainan 图 4 二维主坐标分析结果 Fig. 4 Plot of principal co-ordinates analysis |
3 讨论
小花老鼠簕在我国海南、广东和广西均有分布。海南文昌清澜港是其集中分布区,三亚榆林河的小花老鼠簕已经灭绝,儋州湾和东寨港有少量分布[24]。王日明等[25]报道钦江河口潮滩分布有部分小花老鼠簕,但是笔者采样时没有找到小花老鼠簕群落。因此,本研究仅探讨广东广州、广东湛江、广西防城港及海南文昌4个小花老鼠簕群体。
本研究选用9对SSR标记对广东广州、广东湛江、广西防城港及海南文昌的小花老鼠簕进行遗传多样性分析。9个微卫星标记均来自小花老鼠簕的近缘种老鼠簕,其中6个核微卫星标记的引物与Geng等[21]开发的老鼠簕的核微卫星引物一致,3个叶绿体微卫星标记的引物与Geng等[22]开发的老鼠簕叶绿体微卫星引物一致。Geng等[21]从老鼠簕和榄李(Lumnitzera racemosa)中分别分离到8和9个核微卫星基因座,每个基因座的等位基因数在老鼠簕中有2到8个不等,在榄李中从2到9个不等,老鼠簕的观测杂合度为0.200-0.875,榄李为0.025-0.350,这些核微卫星标记能有效分析这两种红树植物的遗传多样性和种群遗传结构。Geng等[22]从老鼠簕、桐花树(Aegiceras corniculatum)、海榄雌(Avicennia marina)和榄李中开发多态性叶绿体微卫星标记,对229株老鼠簕、509株桐花树、369株海榄雌和216株榄李进行研究,发现每个标记有2-4个等位基因,基因多样性在0.005到0.675之间,这些cpSSR标记将有助于分析这4个物种的母系分布和种群遗传结构。本研究所用标记检测到的等位基因数为2-14个。本研究使用的6个核微卫星位点的多态性信息含量均大于0.50,根据Botstein等[23]的标准,均呈现出高度多态性。因此,这些位点可以提供的遗传信息相对较多。小花老鼠簕的cpSSR遗传多样性处于中低水平,Acaicp02 (PIC=0.374 0)和Acaicp03 (PIC=0.467 3)为中度多态位点,Acaicp04(PIC=0.140 7)为低度多态位点。综合考虑等位基因数、多态性信息含量及Shannon′s多样性指数3个指标,Acil13、Acai02、Acai08、Acai11、Acai12、Acai13等核SSR位点表现出较高的遗传多样性,表明这些位点较好地反映了广东广州、广东湛江、广西防城港及海南文昌的小花老鼠簕的遗传多样性,适合应用于广东广州、广东湛江、广西防城港及海南文昌的小花老鼠簕的遗传多样性检测。
群体遗传多样性表明,4个小花老鼠簕群体的遗传多样性较丰富,等位基因数为2.444-4.111,平均值为3.167。观测杂合度和期望杂合度的平均值分别为0.396和0.442。广东广州、广东湛江、广西防城港及海南文昌4个群体的小花老鼠簕的遗传多样性相比较,海南文昌的小花老鼠簕的等位基因数(4.111)高于其余3个群体。在Structure分析中,海南文昌的色块相较于其他群体的色块底部更复杂一些(图 3),表明海南文昌的小花老鼠簕的遗传潜力较大,物种的杂合程度较高。
聚类和主成分分析结果与各群体的地理位置分布较为一致,4个群体被分为两大类,除广东广州的小花老鼠簕外,广东湛江、广西防城港及海南文昌的小花老鼠簕聚为一类,符合各群体地理位置分布。本结果显示广东广州的小花老鼠簕与其他各群体遗传分化最大。
本研究采用6对nSSR标记及3对cpSSR标记,利用全自动DNA测序仪取代传统的垂直聚丙烯酰胺凝胶电泳进行SSR基因分型。SSR荧光标记毛细管电泳检测方法能够直接读出目标DNA片段的准确大小。显然,相比于常规的聚丙烯酰胺凝胶电泳检测,SSR荧光标记毛细管电泳检测数据更为精确,同时解决了大规模不同批次DNA指纹鉴定数据准确比较及有效整合的难题[26]。然而,若要更全面反映小花老鼠簕的遗传多样性,应将更多的小花老鼠簕群体纳入进来。
在过去的几十年中,小花老鼠簕种群的数量和规模急剧减少。小花老鼠簕现在是一种濒危植物,但是群体遗传多样性表明,4个小花老鼠簕群体的遗传多样性较丰富。尽管本研究中使用了有限的基因座,但为小花老鼠簕的遗传多样性提供了基础数据。在这项研究中,基因座之间的基因流差异较大(表 2),这可能与这些基因座上的选择压力差异有关。
小花老鼠簕面临濒危可能是由于人类活动对其栖息地的破坏、繁育能力差以及病虫害等导致的,而非缺乏遗传多样性。由于笔者曾在野外发现虾塘污水排放导致小花老鼠簕烂根,所以推测人为干扰可能是小花老鼠簕面临的最大威胁。建议采用以下保护措施:(1)优先采用就地保护策略,如防止药用用途的非法采挖,同时加强对小花老鼠簕生境的保护。(2)通过人工繁殖来扩大种群规模。可以通过人工育种培育出大量的小花老鼠簕,然后将它们移植到野外,以达到扩大种群规模的目的。(3)采取迁地保护战略,在其他地区建立新的种群。(4)加强对小花老鼠簕繁殖生物学的研究。运用繁殖生物学的原理与方法,对小花老鼠簕授粉受精、胚珠发育、种子萌发、花部特征及交配系统等方面进行分析研究,如采用荧光染色观察花粉粒萌发及花粉管生长,采用石蜡切片法对胚珠结构进行观察,研究小花老鼠簕是否为需光种子,对温度的适应性,抗盐能力,在自然生境下的萌发率及幼苗成活率,花果期、群体花期及单花花期,花粉活力及柱头的可授性,杂交指数、花粉胚珠比,结合人工控制授粉试验,判断小花老鼠簕群体内是否存在以异交为主的混合交配系统,是否存在部分自交亲和现象,是否需要传粉媒介(如昆虫和风)等。(5)加强对小花老鼠簕病虫害防治方面的研究。
4 结论本研究通过9对SSR引物对广东广州、广东湛江、广西防城港及海南文昌4个群体的小花老鼠簕进行遗传多样性分析。9对SSR引物共检测到61个等位基因。本研究使用的6个核微卫星位点的多态性信息含量均大于0.50,都呈现出高度多态性。4个群体被分为两大类,聚类结果较好地反映了各群体的地理位置分布。9个多态性微卫星位点揭示了4个小花老鼠簕群体的遗传多样性较丰富。小花老鼠簕的濒危可能是由于人类活动、繁育能力差以及病虫害等导致的。建议优先采用就地保护策略,并采用人工繁殖来扩大种群规模,加强对小花老鼠簕繁殖生物学以及病虫害防治方面的研究。本研究结果为小花老鼠簕的保护、评价以及开发利用提供了科学依据。
| [1] |
吴威, 李彩霞, 陈雪初. 基于生态系统服务的海岸带生态修复工程成效评估: 以鹦鹉洲湿地为例[J]. 华东师范大学学报(自然科学版), 2020(3): 98-108. |
| [2] |
李晶, 雷茵茹, 崔丽娟, 等. 我国滨海滩涂湿地现状及研究进展[J]. 林业资源管理, 2018(2): 24-28, 137. |
| [3] |
CHEN B Q, XIAO X M, LI X P, et al. A mangrove forest map of China in 2015:analysis of time series Landsat 7/8 and Sentinel-1A imagery in Google Earth Engine cloud computing platform[J]. ISPRS Journal of Photogrammetry and Remote Sensing, 2017, 131: 104-120. DOI:10.1016/j.isprsjprs.2017.07.011 |
| [4] |
COSTANZA R, D'ARGE R, GROOT R D, et al. The value of the world's ecosystem services and natural capital[J]. Nature, 1997, 25(1): 3-15. |
| [5] |
范航清, 王文卿. 中国红树林保育的若干重要问题[J]. 厦门大学学报(自然科学版), 2017, 56(3): 323-330. |
| [6] |
TOMLINSON P B. The botany of mangroves[M]. Ca-mbridge: Cambridge University Press, 1986.
|
| [7] |
王文卿, 王瑁. 中国红树林[M]. 北京: 科学出版社, 2007.
|
| [8] |
廖宝文, 张乔民. 中国红树林的分布、面积和树种组成[J]. 湿地科学, 2014, 12(4): 435-440. |
| [9] |
周国强, 严成其, 杨勇, 等. 分子标记技术的发展及其在水稻抗白叶枯病研究中的应用[J]. 浙江农业学报, 2010, 22(4): 533-538. |
| [10] |
GAO C H, TANG Z L, YIN J M, et al. Characterization and comparison of gene-based simple sequence repeats across Brassica species[J]. Molecular Genetics and Genomics, 2011, 286: 161-170. |
| [11] |
白晓倩, 陈于, 张仕杰, 等. 基于表型性状和SSR标记的板栗品种遗传多样性分析及分子身份证构建[J]. 植物遗传资源学报, 2022, 23(4): 972-984. |
| [12] |
HA Y J, SA K J, LEE J K. Identifying SSR markers associated with seed characteristics in Perilla (Perilla frutescens L.)[J]. Physiology and Molecular Biology of Plants, 2021, 27(1): 93-105. |
| [13] |
LI X X, QIAO L J, CHEN B R, et al. SSR markers development and their application in genetic diversity evaluation of garlic (Allium sativum) germplasm[J]. Plant Diversity, 2022, 44(5): 481-491. |
| [14] |
公培民, 宁书菊, 叶齐, 等. 马蓝转录组中SSR位点信息特征分析[J]. 分子植物育种, 2020, 18(12): 4000-4008. |
| [15] |
李俊仁, 陈秀珍, 汤小婷, 等. 穿心莲转录组SSR位点信息分析[J]. 中国中药杂志, 2018, 43(12): 2503-2508. |
| [16] |
GENG Q F, WANG Z S, TAO J M, et al. Ocean currents drove genetic structure of seven dominant mangrove species along the goastlines of Southern China[J]. Frontiers Genetics, 2021, 12615911. |
| [17] |
CERÓN-SOUZA I, BERMINGHAM E, MCMILLAN W O, et al. Comparative genetic structure of two mangrove species in Caribbean and Pacific Estuaries of Panama[J]. BMC Evolutionary Biology, 2012, 12: 205. |
| [18] |
阮宇, 吕佳, 李俊清, 等. 利用近似贝氏计算推论台湾海峡沿岸秋茄种群的拓殖路线[J]. 生态学报, 2015, 35(13): 4304-4313. |
| [19] |
MILLÁN-AGUILAR O, MANZANO-SARABIA M, NETTEL-HERNANZ A, et al. Genetic diversity of the black mangrove Avicennia germinans (L.) stearn in northwestern Mexico[J]. Forests, 2016, 7(9): 197. |
| [20] |
HU W M, WANG S W, JIAN S G, et al. Discovery and comparative assessment of SNPs and SSRs for genetic diversity analysis of wild Heritiera littoralis Dryand (Sterculiaceae), an endangered mangrove tree species[J]. Conservation Genetics Resources, 2015, 7(3): 663-668. |
| [21] |
GENG Q F, LIAN C L, TAO J M, et al. Permanent Genetic Resources: development of microsatellite markers for two nonviviparous mangrove species, Acanthus ilicifolius and Lumnitzera racemosa[J]. Molecular Ecology Resources, 2008, 8(2): 377-380. |
| [22] |
GENG Q F, KIMURA M K, LIAN C L, et al. Isolation and characterization of chloroplast microsatellite markers in four mangrove species, Aegiceras corniculatum, Avicennia marina, Acanthus ilicifolius and Lumnitzera racemosa[J]. Conservation Genetics, 2009, 10(4): 1133-1140. |
| [23] |
BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980, 32(3): 314-331. |
| [24] |
张颖, 陈光程, 钟才荣. 中国濒危红树植物研究与恢复现状[J]. 应用海洋学学报, 2021, 40(1): 142-153. |
| [25] |
王日明, 梁喜幸, 周晓妍, 等. 钦江河口潮滩红树林群落空间分布[J]. 遥感学报, 2022, 26(6): 1143-1154. |
| [26] |
程本义, 夏俊辉, 龚俊义, 等. SSR荧光标记毛细管电泳检测法在水稻DNA指纹鉴定中的应用[J]. 中国水稻科学, 2011, 25(6): 672-676. |



