2. 中国水产科学研究院东海水产研究所, 上海 200090
2. East China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Shanghai, 200090, China
锈斑蟳(Charybdis feriata)俗称红花蟹,隶属节肢动物门(Arthropoda)甲壳纲(Crustacea)梭子蟹科(Portunidae)蟳属(Charybdis),是我国大型水产经济蟹类之一,主要分布于广西、广东、台湾、海南等东南沿海地区的近岸海域[1]。锈斑蟳多栖息于近岸浅海底或岛礁,属底栖生物食性类型,主要以甲壳类、鱼类为食,兼食珊瑚虫、底栖藻类和腹足类,其肉质鲜美、营养价值高,具有较高的经济价值[2, 3]。20世纪90年代以前,锈斑蟳主要为底拖网、拖虾网等兼捕对象,自东海中、南部海域三疣梭子蟹(Portunus trituberculatus)资源衰落和蟹笼作业兴起后,锈斑蟳成为蟹笼、定制刺网作业的主捕对象,加上锈斑蟳资源群体数量不大,缺少如细点圆趾蟹(Ovalipes punctatus)的明显高生物量区域[4, 5],导致近年来锈斑蟳生物资源量逐渐减少,分布范围不断缩小[6],可能造成群体间的基因交流频率降低,遗传多样性发生改变。目前对锈斑蟳的研究主要集中在渔业资源调查[1]、人工育苗[7]、胚胎发育[8]、营养成分评价[9]、食性与营养级研究[10]等方面,尚未对其种群遗传多样性和遗传结构开展系统评价和研究。
简单重复序列(Simple Sequence Repeats,SSR)长度多态性又称微卫星DNA(Microsatellite DNA)标记,是一类由1-6个核苷酸为基本单位组成的重复串联序列[11]。由于SSR具有进化所受选择压力小、共显性遗传、易于分析、重复性较好等特点,现已成为第二代分子辅助标记方法[12],被广泛应用于遗传多样性检测及遗传差异性评估中。利用高通量测序数据是目前开发SSR引物的主要方法,常用的高通量测序技术主要为全基因组测序(全基因组中测序、全基因组低深度重测序)、靶向捕获测序技术和简化基因组测序[13]。全基因组测序技术是通过对全基因组文库扫描测序,获得整个基因组范围的遗传变异信息;靶向捕获测序技术是对靶向位点进行测序,获取目标位点的变异信息;而简化基因组测序技术则是对特定的酶切位点进行高通量测序,从而获得目标区域的遗传标记信息。其中,由于简化基因组测序技术相比其他测序技术具有测序数据量少、生物信息学分析周期短、测序成本相对较低等优点[14-16],现已在大鳞大麻哈鱼(Oncorhynchus tshawytscha)[17]、三棘刺鱼(Gasterosteus aculeatus)[18]、印尼虎鱼(Datnioides pulcher)[19]、长体圆鲹(Decapterus macrosoma)[20]等水生生物的种群遗传学研究领域得到广泛应用。
然而,目前对锈斑蟳的微卫星DNA标记开发相对较少,除了Ma等[21]在2016年首次利用锚定PCR分离并验证了8对锈斑蟳多态性SSR引物,以及Fang等[22]通过全基因组测序技术获得并验证了14对多态性SSR引物之外,未见其他相关报道。因此,本研究拟采用简化基因组测序技术开发适用于锈斑蟳的SSR引物,并对筛选出的多态性SSR分子标记进行遗传多样性验证,以期为基于SSR微卫星DNA标记开展的锈斑蟳遗传多样性、种群遗传分析等研究提供基础资料,这对促进锈斑蟳种质资源的保护与可持续利用具有重要意义。
1 材料与方法 1.1 材料根据锈斑蟳野生资源调查文献记录[4, 23, 24],实验用锈斑蟳样本分别采集自广东省阳江市、福建省宁德市、广西壮族自治区北海市、海南省三亚市、浙江省宁波市、福建省漳州市等6个沿海城市,具体采样信息见表 1。取锈斑蟳螯足长节肌肉经液氮速冻后,-80℃保存。
| 样本Sample | 采集地点Sample collection site | 纬度Latitude | 经度Longitude | 采样数量/个Number of samples | 体重/g Body weight/g | 采样日期Sampling date |
| GD | Yangjiang City, Guangdong Province |
21°34′21.92″N | 111°49′58.13″E | 3 | 143.87±7.99 | 2023-10-20 |
| ND | Ningde City, Fujian Province |
27°03′49.86″N | 120°13′34.38″E | 3 | 137.77±9.55 | 2023-11-07 |
| GX | Beihai City, Guangxi Zhuang Autonomous Region |
21°26′34.11″N | 109°03′49.86″E | 3 | 145.70±8.15 | 2023-12-05 |
| HN | Sanya City, Hainan Province |
18°13′57.41″N | 109°30′15.36″E | 3 | 168.67±12.65 | 2023-10-31 |
| ZJ | Ningbo City, Zhejiang Province |
29°13′16.43″N | 121°57′4.75″E | 3 | 147.87±7.01 | 2023-11-27 |
| FJ | Zhangzhou City, Fujian Province |
23°54′33.43″N | 117°44′25.34″E | 3 | 164.20±10.72 | 2023-10-23 |
1.2 方法 1.2.1 DNA提取和质控
使用全自动核酸提取仪(NanoMagBio S-96,武汉纳磁生物科技有限公司)提取锈斑蟳基因组DNA。取30-50 mg样品适当剪碎放置提取板对应孔位;向样品板中每孔加入400 μL组织裂解液和5 μL蛋白酶K,振荡混匀,置于65 ℃恒温箱消化16 h;在新的提取板中每孔分装100 μL磁珠放置在提取仪工位2,分装3板500 μL/孔的75%乙醇分别放在工位3、工位4、工位5,洗脱板每孔分装100/80/60 μL(根据样品状态分装对应体积)洗脱液放置在工位6;消化结束后,将提取板取出放在提取仪工位1,启动程序裂解15 min,裂解完成后,向样品板每个孔中加入280 μL异丙醇,将样品板放在提取仪工位1;选择自定义的核酸提取程序,检查仪器状态和提取板信息后运行;运行完成之后,将洗脱板取出进行DNA浓度和电泳胶检测,4 ℃条件下保存。取2 μL DNA原液进行NanoDrop 8000检测,取2 μL DNA原液添加2 μL溴酚蓝进行1%琼脂糖凝胶电泳检测。
1.2.2 简化基因组测序取6个质控合格的样本DNA进行建库,根据预实验选择内切酶DpnII_MspI将DNA分解成小片段,富集220-450 bp的片段,构建Illumina测序文库,利用二代测序技术,基于Illumina Novaseq测序平台,开展双端测序。对原始测序数据进行过滤,生成高质量序列。将样品的数据合并成pop A,再采用FLASH(version 1.2.11, Magoc and Salzberg 2011)对序列按照overlap搭载为一条高质量reads,参数设置如下:(1)Min overlap: 10;(2)Max mismatch density: 0.2;(3)Allow “outie” pairs: false。
1.2.3 SSR位点信息分析与引物设计采用微卫星识别工具(MIcroSAtellite Identification Tool,MISA,http://pgrc.ipk-gatersleben.de/misa/) 在所有序列中搜索SSR位点,搜索所采用的参数如下:mono-10、di-6、tri-5、Tetra-5、penta-5、hexa-5。复合序列中两个不同SSR之间允许的最大间隔设置为100 bp。采用Perl程序屏蔽序列上的重复序列(用字母R代替),过滤掉侧翼序列短于20 bp的SSR(过短的侧翼序列无法进行相似性比对)。利用cdhit 4.8.1软件对过滤后的序列进行聚类,聚类所采用的核苷酸序列的相似度设置为90%,覆盖度为70%,空位罚分设置为-gap1-gap-ext 0。其中,一条序列上有两个及以上SSR的序列分开统计聚类群组。采用Perl程序对聚类结果进行解析,根据SSR的长度分别对每一个类进行统计,同一个类中所有SSR的长度一致,则该类的多态性为1;如果同一个类中SSR具有2种长度,则该类的多态性为2;依次类推,获得每一个类的SSR多态性。利用Primer3 v2.3.6软件分别对搜索到的多态性>2的Clusters内的SSR序列针对位点两端设计引物;扩增目标片段长度控制在100-400 bp,扩增目标片段位置为重复序列前1个碱基到重复序列后5个碱基,其他参数默认。对所有设计得到的引物进行筛选,筛选标准如下:(1)设计引物的SSRs类型为非单核苷酸重复模体、非复合重复模体;(2)仅选取每条read上有一个SSR的SSRs类型设计引物,以避免扩增过程中扩增出第二条SSR影响多态性;(3)设计的引物必须在某一个Clusters内,且Clusters内SSRs的长度多态性≥2;(4)每一条设计的引物在Clusters中必须有两条引物支持(避免测序错误);(5)去除完全相同的引物结果。
1.2.4 引物验证从筛选后的设计引物中选择多态性较高的引物用于实验验证,引物采用接头法合成,即合成时在上游引物加上21 bp的接头序列。采用接头法进行PCR扩增时,第一步带接头的上游引物与下游引物、模板结合,得到带有接头序列的PCR产物,第二步带荧光基团的接头引物与下游引物、第一步的PCR产物结合,得到带有荧光基团和21 bp接头序列的PCR产物。PCR扩增体系为10.0 μL,包含5.0 μL 2×Taq PCR Master Mix,10 pmol/μL上、下游引物各0.5 μL,基因组DNA 1.0 μL,用灭菌超纯水补至10.0 μL。每个群体选择2个样本,共计12个样本筛选验证所设计引物,反应在Veriti384 PCR仪(美国Thermo Fisher Scientific公司)上进行。PCR扩增程序设置为95 ℃预变性5 min;95 ℃变性30 s,62-52 ℃梯度退火30 s,72 ℃延伸30 s,运行10个循环;95 ℃变性30 s,52 ℃退火30 s,72 ℃延伸30 s,运行25个循环;72 ℃延伸20 min,最后4 ℃保存。PCR反应结束后,扩增产物经荧光毛细管电泳检测。使用GeneMarker 3.0.0软件对结果进行分析,获得每个样品的等位基因数、峰图和基因型。
2 结果与分析 2.1 简化基因组测序数据质量锈斑蟳6个样本的简化基因组测序共获得原始数据6.78 GB,每个样本的数据量为(1 129 203 648±145 715 946.0) bp,序列数为7 841 692±1 011 916.3,GC比例为43.37±0.73,Q30%为95.04±0.23。对数据进行质控过滤后得到高质量数据6.44 GB,每个样本过滤后的高质量序列数据量为(1 073 912 793±139 382 244.1) bp,序列数为7 563 637±979 862.3,GC比例为43.18±0.73,Q30%为97.36±0.05(表 2)。结果表明,送测样本的数据质量满足分析要求,测序数据准确度较高,GC比例在正常范围。
| 样本Sample | 过滤前 Before filter |
过滤后 After filter |
|||||||
| 序列数 Sequence number |
数据量/bp Data/bp |
GC比例 GC content |
Q30% | 序列数 Sequence number |
数据量/bp Data/bp |
GC比例 GC content |
Q30% | ||
| GD01 | 8 142 426 | 1 172 509 344 | 43.79 | 95.16 | 7 857 916 | 1 116 765 135 | 43.63 | 97.34 | |
| ND01 | 7 973 226 | 1 148 144 544 | 43.68 | 94.57 | 7 672 878 | 1 085 459 924 | 43.44 | 97.26 | |
| GX01 | 6 638 540 | 955 949 760 | 43.33 | 95.19 | 6 418 858 | 912 078 772 | 43.16 | 97.39 | |
| HN01 | 8 518 824 | 1 226 710 656 | 41.82 | 95.18 | 8 234 968 | 1 169 521 701 | 41.64 | 97.44 | |
| ZJ01 | 6 452 318 | 929 133 792 | 44.08 | 94.93 | 6 202 428 | 880 836 211 | 43.90 | 97.36 | |
| FJ01 | 9 324 818 | 1 342 773 792 | 43.49 | 95.19 | 8 994 774 | 1 278 815 014 | 43.33 | 97.36 | |
| Mean value | 7 841 692± 1 011 916.3 | 1 129 203 648± 145 715 946.0 | 43.37± 0.73 | 95.04± 0.23 | 7 563 637± 979 862.3 | 1 073 912 793± 139 382 244.1 | 43.18± 0.73 | 97.36± 0.05 | |
2.2 简化基因组中的SSR位点情况
将6个样本的数据进行序列合并后得到popA样本集,样本popA中可检测数据量为2.24 GB,可检测序列数为9 568 560。通过MISA软件开展SSR搜索后识别出SSR位点2 419 242个,含有SSR的序列数为1 786 277,其中含有超过一个SSR位点的序列数为480 143,存在复合性SSR位点的序列数为604 684。
对锈斑蟳不同SSR的类型和基序等进行统计(表 3)。其中,二核苷酸重复基序最多,数量为1 063 280个,占总位点的43.95%;其次为单核苷酸的重复基序,数量为694 638个,占总位点的28.71%;三核苷酸重复基序的数量为559 377个,占总位点的23.12%,四核苷酸和五核苷酸重复基序数量分别为85 909和10 148个,分别占总位点的3.55%和0.41%;六核苷酸重复基序的数量最少,仅有5 890个,占总位点的0.24%。在这些SSR位点中,单核苷酸重复位点中A/T为主要基序,占该类型基序的97.72%;二核苷酸重复基序类型有12种,其中主要基序为AG/CT和AC/GT,占比分别为57.72%和40.33%;三核苷酸重复基序有60种,ACT/AGT、AGG/CCT和AAT/ATT为主要基序,共占该类型基序的59.12%;四核苷酸重复基序的种类数为207种,其中AAAT/ATTT和ACAG/CTGT为主要基序,占该类型基序的38.53%;五核苷酸的重复基序类型数量为275,主要基序分别为AACCT/AGGTT。六核苷酸重复基序类型种类数最多,达287种,其中主要基序为ACAGAG/CTCTGT和AGAGGG/CCCTCT,数量分别为1 187和1 170个。
| SSR类型SSR type | 重复基序数量Number of repeated motifs | 占总基序数量的比例/% Percentage in total number of motifs/% | 重复基序类型数量Number of motif type | 主要基序Main motif |
| Mono-nucleotide | 694 638 | 28.71 | 4 | A/T |
| Di-nucleotide | 1 063 280 | 43.95 | 12 | AG/CT,AC/GT |
| Tri-nucleotide | 559 377 | 23.12 | 60 | ACT/AGT,AGG/CCT,AAT/ATT |
| Tetra-nucleotide | 85 909 | 3.55 | 207 | AAAT/ATTT,ACAG/CTGT |
| Penta-nucleotide | 10 148 | 0.41 | 275 | AACCT/AGGTT |
| Hexa-nucleotide | 5 890 | 0.24 | 287 | ACAGAG/CTCTGT,AGAGGG/CCCTCT |
2.3 锈斑蟳SSR位点基序的分布
锈斑蟳基因组不同类型SSR位点基序的重复次数统计见表 4,单核苷酸的基序重复次数主要集中在10-13次,二核苷酸的基序重复次数主要集中在6-7次,三核苷酸、四核苷酸和五核苷酸的基序重复次数主要集中在5-7次,六核苷酸的基序重复次主要集中在5-6次。
| 重复次数 Repeat number |
重复基序的分布数量 Amounts of motifs |
|||||
| 单核苷酸 Mono-nucleotide |
二核苷酸 Di-nucleotide |
三核苷酸 Tri-nucleotide |
四核苷酸 Tetra-nucleotide |
五核苷酸 Penta-nucleotide |
六核苷酸 Hexa-nucleotide |
|
| 5 | 0 | 0 | 131 289 | 34 747 | 5 591 | 3 067 |
| 6 | 0 | 110 740 | 110 974 | 18 062 | 2 078 | 1 427 |
| 7 | 0 | 62 487 | 62 892 | 11 630 | 1 144 | 558 |
| 8 | 0 | 42 716 | 45 118 | 7 004 | 487 | 363 |
| 9 | 0 | 37 234 | 32 888 | 4 243 | 277 | 145 |
| 10 | 219 563 | 30 358 | 27 309 | 2 872 | 260 | 88 |
| 11 | 103 880 | 28 627 | 37 296 | 2 384 | 173 | 71 |
| 12 | 67 665 | 27 380 | 16 497 | 1 407 | 47 | 40 |
| 13 | 53 936 | 24 662 | 13 419 | 935 | 21 | 40 |
| 14 | 41 639 | 22 879 | 12 417 | 744 | 24 | 24 |
| 15 | 32 064 | 22 071 | 10 272 | 538 | 12 | 39 |
| 16 | 23 119 | 21 897 | 8 493 | 471 | 12 | 7 |
| 17 | 16 602 | 19 592 | 8 509 | 201 | 6 | 7 |
| 18 | 13 458 | 19 909 | 6 547 | 179 | 7 | 0 |
| 19 | 12 281 | 18 356 | 9 125 | 121 | 3 | 3 |
| 20 | 12 678 | 19 816 | 4 933 | 86 | 2 | 9 |
| Above 20 | 97 753 | 555 456 | 21 399 | 285 | 4 | 2 |
2.4 多态性引物筛选与验证
利用CD-HIT 4.8.1软件进行SSR聚类后共获得94 793个聚群。对这些聚群进行多态性评估,多态性为1的聚群数量为60 205个,占总聚群的63.51%,多态性>1的数量为34 588个,占总聚群的36.49%。
利用Primer3 v2.3.6软件对搜索到的多态性>1的聚群内的SSR序列针对位点两端设计引物,筛选过滤后共获得21 330对锈斑蟳微卫星引物,优先选取多态性高的192对设计引物,利用12个锈斑蟳样本DNA进行PCR扩增,对PCR产物进行琼脂糖凝胶电泳检测。以SPT689F/SPT690R引物为例,PCR产物表现出明显的单一条带以及较好的多态性(图 1)。
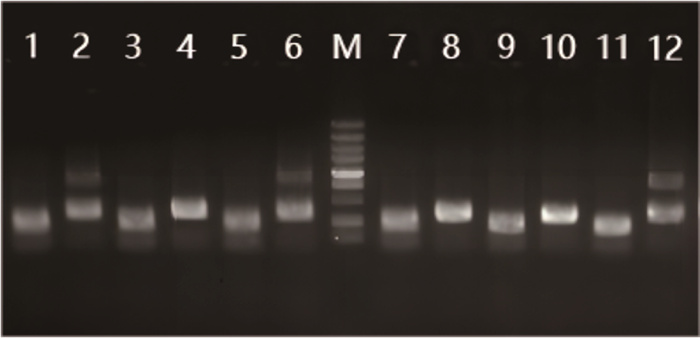
|
| Marker (M) bands are 100, 250, 500, 750, 1 000, 1 500, 2 000, 3 000, and 5 000 bp from bottom to top, and 1-12 represent individual samples. 图 1 微卫星引物SPT689F/SPT690R的12个样本PCR产物结果 Fig. 1 PCR product results of 12 samples with microsatellite primers SPT689F/SPT690R |
对检测合格的PCR产物进行毛细管电泳分型,根据荧光信号,采用GeneMarker 3.0.0软件对分型结果进行读取,以XBX007位点为例(图 2),毛细管电泳分型谱带清晰,杂峰少,最后筛选出14对具有较高多态性的SSR引物(表 5)。这14对引物的等位基因数共136个,每个引物的等位基因数在4-14,引物的期望杂合度(He)为0.503-0.892,观测杂合度(Ho)为0.167-1.000,多态信息含量(PIC)为0.456-0.882(表 6)。
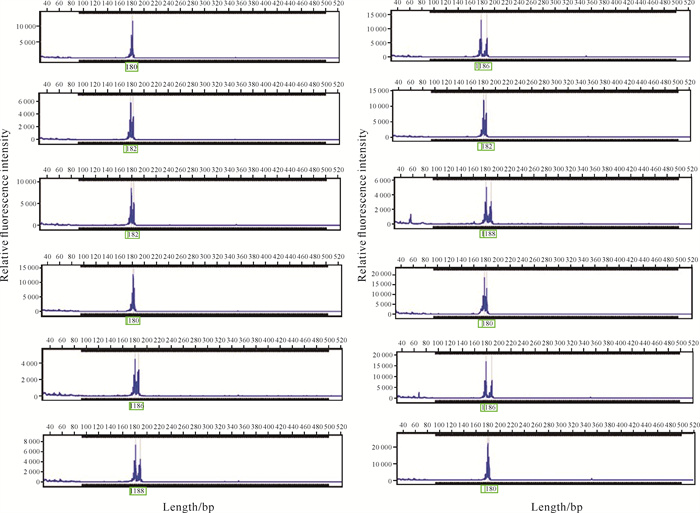
|
| 图 2 12个个体的微卫星引物XBX007基因分型结果 Fig. 2 Gene typing results of 12 individuals based on SSR primer XBX007 |
| SSR位点 SSR loci |
正向引物序列(5′→3′) Forward primer sequence (5′→3′) |
反向引物序列(5′→3′) Reverse primer sequence (5′→3′) |
重复单元 Repeat motif |
预期长度/bp Expected size/bp |
| XBX005 | CACTTTTGGCATCAGGTGAG | GTGCAGTTAAGGTGGGGATG | (AG)19 | 164 |
| XBX007 | GAGGAAAGCAAAACCGATGA | CCGTGCAGTGACTGATGACT | (CA)18 | 171 |
| XBX031 | ACTGCGCGAGGAGAATAAAG | TGGCCAAGGTTGAGCTACTT | (CA)23 | 226 |
| XBX046 | GTATTCCCCTTGGACTGGGT | GAAAGTGGCGAGGATTGGTA | (TC)22 | 136 |
| XBX088 | GGAGTGAGTGAATGCCAAAAA | TTTGTGTTGCTTCATCTTTTGAA | (TC)27 | 176 |
| XBX102 | TGCTTATGCGTATGGAGTGC | CGAAAAACAAAGCGTCACTG | (CA)44 | 193 |
| XBX110 | GGCGACATTTCAACAGACAA | TCATCGTCTGAGCAAGTTGG | (CA)29 | 249 |
| XBX112 | ATGAGAGAAGGGGCGTATGT | GCGGAAATTCTTGGACGATA | (CA)25 | 198 |
| XBX118 | GACTAATTAACCGTGTTGGGC | TGTTTGGCGGGAGAAGTTAC | (AG)11 | 232 |
| XBX122 | TCTCCCTCTGCTTGTCATTG | ATATAGCAGGAAATGCCCCC | (AG)25 | 141 |
| XBX133 | AAATACGGTGTTGGAGAGGG | CCAACAACAAAGCAACGAAA | (TC)20 | 99 |
| XBX135 | AGGTGGAAGGTTACGGTGGT | TAGGGAGAAGGATGTGCTCG | (TG)12 | 195 |
| XBX152 | TTAAATGGGATGAGTTGAGCG | AATACGCCCGCAGTAAATTG | (GT)19 | 210 |
| XBX153 | TTTCACAACAGCGGAGTCAG | GAGGGAAGGACAAGGGAGAG | (CT)15 | 153 |
| 微卫星位点 SSR loci |
样本数 Number |
等位基因数 Number of alleles |
有效等位基因数 Effective number of alleles |
观测杂合度(Ho) Observed heterozygosity (Ho) |
期望杂合度(He) Expected heterozygosity (He) |
多态信息含量(PIC) Polymorphism Information Content (PIC) |
| XBX005 | 12 | 14 | 8.443 | 0.917 | 0.882 | 0.871 |
| XBX007 | 12 | 6 | 4.810 | 0.917 | 0.792 | 0.764 |
| XBX031 | 12 | 12 | 9.271 | 0.750 | 0.892 | 0.882 |
| XBX046 | 12 | 6 | 2.132 | 0.667 | 0.531 | 0.509 |
| XBX088 | 12 | 12 | 6.393 | 0.583 | 0.844 | 0.828 |
| XBX102 | 12 | 4 | 2.013 | 0.167 | 0.503 | 0.456 |
| XBX110 | 12 | 10 | 6.111 | 0.833 | 0.836 | 0.819 |
| XBX112 | 12 | 12 | 8.472 | 0.667 | 0.882 | 0.871 |
| XBX118 | 12 | 11 | 7.790 | 1.000 | 0.872 | 0.860 |
| XBX122 | 12 | 13 | 9.257 | 1.000 | 0.892 | 0.882 |
| XBX133 | 12 | 10 | 8.016 | 1.000 | 0.875 | 0.863 |
| XBX135 | 12 | 11 | 6.400 | 0.917 | 0.844 | 0.827 |
| XBX152 | 12 | 8 | 2.964 | 0.667 | 0.663 | 0.636 |
| XBX153 | 12 | 7 | 4.365 | 0.750 | 0.771 | 0.738 |
3 讨论
设计出足够数量的SSR标记是群体遗传结构分析及种质资源保护研究等方面的基础。传统的微卫星标记开发方法是利用基因组文库构建微卫星富集文库后进行杂交测序,步骤较为繁琐且耗时长、效率低,无法满足遗传学研究中需要快速开发大量分子标记的要求[14]。近年来随着高通量测序技术的发展,更为高效廉价的简化基因组测序已被较多地应用于SSR引物的开发。本研究基于简化基因组测序方法共获得2 419 242个锈斑蟳微卫星位点,包含SSR的序列数量为1 786 277条,这些微卫星位点可以设计较多数量的引物,为下一步研究提供条件。本研究所鉴定到的微卫星位点以二核苷酸重复基序为主,其次是单核苷酸重复基序,这与在远海梭子蟹(Portunus pelagicus)[25]、蛛雪蟹(Chionoecetes opilio)[26]、中华绒螯蟹(Eriocheir sinensis)[27]、凡纳滨对虾(Litopenaeus vannamei)[28]、脊尾白虾(Exopalaemon carinicauda)[29]等其他甲壳类动物SSR标记中发现的二碱基重复基序数量最多的研究结果一致。同时,锈斑蟳重复基序的类型也表现出一定的偏倚性,如单核苷酸、五核苷酸重复基序中主要基序分别为A/T、AACCT/AGGTT,其中A/T为大多数无脊椎动物的主要基序[30],AACCT/AGGTT与其他甲壳类动物基序存在差异,这可能与物种特异性有关。
本研究经简化基因组技术测序组装、筛选过滤与扩增验证,开发确认了14对锈斑蟳SSR多态性引物,获得率总体为7.3%。从遗传多样性来看,微卫星标记中的杂合度能较好地反映群体中等位基因的丰富程度和均匀程度[31],本研究所得14对锈斑蟳SSR引物的Ho为0.167-1.000,平均值为0.770,处于较高水平,表明锈斑蟳样本种群有较高的遗传多样性。从片段多态性来看,SSR等位基因数目和PIC能从一定程度上反映遗传标记包含或提供的遗传信息含量[31],本研究获得的14对多态性微卫星引物中,等位基因数为4-14,高于Cui等[32]获得的拟穴青蟹(Scylla paramamosain)微卫星等位基因数(5-9),和Yap等[25]获得的远海梭子蟹微卫星等位基因数(2-17)相近,但远低于Puebla等[26]获得的蛛雪蟹微卫星标记等位基因数(14-79),这种差异可能是群体物种的选择以及不同位点本身的变异情况不同所致。同时,这些引物的PIC为0.456-0.882,其中仅XBX102为中度多态性位点(0.25<PIC<0.50),其他均为高度多态性位点(PIC≥0.50),表明本研究开发的微卫星DNA标记具有较高的多态性,将其用于锈斑蟳群体遗传多样性研究的可靠性较高。
对比基于锈斑蟳全基因组数据的微卫星标记开发结果[22]可以发现,锈斑蟳全基因组测序共获得原始数据126.6 GB,鉴定出3 779 209个微卫星位点,而本研究仅基于6.78 GB简化基因组原始测序数据,就可以分离鉴定出2 419 242个微卫星位点,表明基于简化基因组数据的微卫星位点的开发效率高于全基因组。此外,本研究基于简化基因组获得的SSR引物的Ho、等位基因数目和PIC平均值分别为0.770、9.710、0.770,均高于全基因组测序获取的SSR引物平均值(0.691、6.800、0.652),且简化基因组获取的14个引物中高度多态性位点比例为92.86%,高于全基因组测序获取的SSR引物高度多态性位点比例(78.57%),这表明高通量测序技术中简化基因组测序技术能相对高效地获得目标物种的序列遗传信息,有利于筛选多态性高、特异性强的引物,是一种较为理想可行的技术[33]。综上,本研究基于简化基因组开发的锈斑蟳微卫星DNA标记和多态性引物,可为后续锈斑蟳群体遗传多样性评价、种质资源保护和管理、遗传连锁图谱构建、分子标记辅助育种等工作提供数据基础,为锈斑蟳品种选育和品系遗传种质评估提供科学依据。
| [1] |
黄培民. 东海南部锈斑蟳数量分布及生物学特点[J]. 福建水产, 2006, 28(1): 23-25. |
| [2] |
王红勇, 姚雪梅. 虾蟹生物学[M]. 北京: 中国农业出版社, 2007.
|
| [3] |
张珏. 溶解氧、温度、盐度、氨氮和亚硝酸盐氮对锈斑蟳(Charybdis Feriatus)存活和摄饵的影响[D]. 湛江: 广东海洋大学, 2010.
|
| [4] |
俞存根, 宋海棠, 姚光展. 东海中南部海域锈斑蟳渔业生物学和数量分布[J]. 湛江海洋大学学报, 2005, 25(3): 24-28. |
| [5] |
俞存根, 宋海棠, 姚光展. 东海细点圆趾蟹数量分布的研究[J]. 水产学报, 2005(2): 198-204. |
| [6] |
王语同. 闽南渔场蟹笼作业渔获组成和主要种类[J]. 河北渔业, 2011(9): 16-21. |
| [7] |
邹雄, 杨明秋, 蒲利云, 等. 锈斑蟳人工育苗技术[J]. 湖北农业科学, 2023, 62(9): 97-100. |
| [8] |
廖永岩, 黄惠良, 王能钦. 室内锈斑蟳幼体发育的形态观察[J]. 热带海洋学报, 2010, 29(4): 138-147. |
| [9] |
陈瑜, 许丹, 何鹏飞, 等. 锈斑蟳蒸煮液和酶解液氨基酸组成及滋味分析[J]. 食品研究与开发, 2022, 43(18): 157-163. |
| [10] |
黄美珍. 福建海区拥剑梭子蟹、红星梭子蟹和锈斑蟳的食性与营养级研究[J]. 台湾海峡, 2004(2): 159-166. |
| [11] |
BECKMAN J S, WEBER J L. Survey of human and rat microsatellites[J]. Genomics, 1992, 12(4): 627-631. DOI:10.1016/0888-7543(92)90285-Z |
| [12] |
LIU Z J, CORDES J F. DNA marker technologies and their applications in aquaculture genetics[J]. Aquaculture, 2004, 238(1/2/3/4): 1-37. |
| [13] |
石少磊, 武丽娜, 冯羿方, 等. 基因组检测技术在肉鸡遗传育种中的应用[J]. 中国畜禽种业, 2023, 19(12): 122-128. |
| [14] |
贺玉姣, 胡万军, 都玉蓉, 等. 基于高原鼠兔简化基因组数据的微卫星引物开发[J]. 野生动物学报, 2021, 42(3): 730-736. |
| [15] |
宁馨, 姜小龙, 邓敏, 等. 基于简化基因组数据开发岭南青冈微卫星引物[J]. 植物研究, 2020, 40(4): 629-634. |
| [16] |
ANDREWS K R, GOOD J M, MILLER M R, et al. Harnessing the power of RADseq for ecological and evolutionary genomics[J]. Nature Reviews Genetics, 2016, 17(2): 81-92. DOI:10.1038/nrg.2015.28 |
| [17] |
LARSON W A, SEEB L W, EVERETT M V, et al. Genotyping by sequencing resolves shallow population structure to inform conservation of Chinook salmon (Oncorhynchus tshawytscha)[J]. Evolutionary Applications, 2014, 7(3): 355-369. DOI:10.1111/eva.12128 |
| [18] |
HOHENLOHE P A, AMISH S J, CATCHEN J M, et al. Next-generation RAD sequencing identifies thousands of SNPs for assessing hybridization between rainbow and westslope cutthroat trout[J]. Molecular Ecology Resources, 2011, 11(Suppl 1): 117-122. |
| [19] |
屈政委, 宋红梅, 汪学杰, 等. 印尼虎鱼RAD-seq数据中微卫星标记的初步筛选及特征分析[J]. 淡水渔业, 2019, 49(4): 9-15. |
| [20] |
孔啸兰, 李敏, 陈作志, 等. 基于RAD-seq技术的长体圆鲹二、三核苷酸重复微卫星标记开发与评价[J]. 南方水产科学, 2019, 15(3): 97-103. |
| [21] |
MA H Y, ZOU X, JI X S, et al. Discovery and characterization of a first set of polymorphic microsatellite markers in red crab (Charybdis feriatus)[J]. Journal of Genetics, 2013, 92(3): e113-e115. |
| [22] |
FANG S B, WU R X, SHI X, et al. Genome survey and identification of polymorphic microsatellites provide genomic information and molecular markers for the red crab Charybdis feriatus (Linnaeus, 1758) (Decapoda: Brachyura: Portunidae)[J]. Journal of Crustacean Biology, 2020, 40(1): 76-81. DOI:10.1093/jcbiol/ruz074 |
| [23] |
杨思谅, 陈惠莲, 戴爱云. 中国动物志: 无脊椎动物第四十九卷甲壳动物亚门十足目梭子蟹科[M]. 北京: 科学出版社, 2011.
|
| [24] |
钟爱华. 舟山沿海锈斑蟳肌肉氨基酸组成及营养分析[J]. 中国农学通报, 2015, 31(2): 97-100. |
| [25] |
YAP E S, SEZMIS E, CHAPLIN J A, et al. Isolation and characterization of microsatellite loci in Portunus pelagicus (Crustacea: Portunidae)[J]. Molecular Ecology Notes, 2002, 2(1): 30-32. |
| [26] |
PUEBLA O, PARENT É, SÉVIGNY J M. New microsatellite markers for the snow crab Chionoecetes opilio (Brachyura: Majidae)[J]. Molecular Ecology Notes, 2003, 3(4): 644-646. |
| [27] |
ZHU Z Y, SHI Y H, LE G W. Isolation and characterization of polymorphic microsatellites from Chinese mitten crab, Eriocheir sinensis[J]. Molecular Ecology Notes, 2006, 6(3): 838-839. |
| [28] |
DENISE K G, ACACIA A W. Characterization of 35 new microsatellite genetic markers for the pacific whiteleg shrimp, Litopenaeus vannamei: their usefulness for studying genetic diversity of wild and cultured stocks, tracing pedigree in breeding programs, and linkage mapping[J]. Journal of Shellfish Research, 2007, 26(4): 1203-1216. |
| [29] |
段亚飞, 张喆, 李吉涛, 等. 脊尾白虾血细胞ESTs的生物信息学与微卫星序列特征分析[J]. 水产科学, 2016, 35(5): 562-567. |
| [30] |
JIA S W, LI Y C, CHEN S Q, et al. Microsatellite markers for Monitipora digitata designed using restriction-site associated DNA sequencing[J]. Frontiers in Marine Science, 2023, 10: 1019419. |
| [31] |
国伟, 沈佐锐. 微卫星DNA的多态性及其应用[J]. 生物技术通讯, 2004, 15(2): 158-159. |
| [32] |
CUI H, MA H, MA L, et al. Development of eighteen polymorphic microsatellite markers in Scylla paramamosain by 5' anchored PCR technique[J]. Molecular Biology Reports, 2011, 38(8): 4999-5002. |
| [33] |
欧阳泽怡, 李志辉, 牟虹霖, 等. 基于简化基因组开发青冈和滇青冈微卫星引物[J/OL]. 南京林业大学学报(自然科学版), 2023, 1-13(2023-05-25)[2024-04-17]. http://kns.cnki.net/kcms/detail/32.1161.s.20230524.1029.002.html.
|



