2. 广西科学院人工智能研究院, 广西南宁 530007
2. Artificial Intelligence Research Institute, Guangxi Academy of Sciences, Nanning, Guangxi, 530007, China
现代新药的研发往往周期长、耗资大且具有一定的随机性和盲目性[1]。通常,研发一种新药大约需要26亿美元[2],且新药从研发开始到成功上市通常耗时10-17年[3]。药物的重定位,即对现有的、已获批准的药物确定新的用途,这将大大减少药物研发所需的时间和成本[4]。药物-靶标相互作用(Drug-Target Interaction, DTI) 的研究,在新药研发和药物重定位领域都是一项非常复杂的关键任务。有效、准确的DTI数据能够为新药设计、药物毒副作用研究、药物重定位研究和精准医疗提供强有力的支持。随着计算机技术的发展,利用计算机方法预测DTI成为可能且成效显著。尤其是,近年来化学、生物与药物领域的数据库不断涌现,基于数据驱动的DTI预测方法应运而生,迅速引起了人们的广泛关注并成为人工智能辅助药物研发领域的热门研究主题[5]。基于数据驱动的DTI预测可望替代较多的生物学实验,从而加快药物研发效率、降低成本,减少实验动物的使用。
DTI预测方法可以分为基于配体的方法[6]、基于对接的方法[7]和化学基因组学方法[8]。基于配体的方法是指基于蛋白质配体的相似性来发现DTI。基于对接的方法则是利用蛋白质三维空间结构数据,执行模型,评估其与特定药物结合的关系。化学基因组学方法一般使用机器学习技术对靶标和药物的基因组以及化学信息等数据进行DTI建模和预测[9, 10]。然而,由于不少配体和蛋白质的三维结构尚不明确,致使基于配体的方法和基于对接的方法存在较大的局限性。因此,化学基因组学方法是比较有发展前途的DTI预测方法,并成为目前的研究热点。
近年来,支持向量机[11]、模糊逻辑[12]、集成学习[13-16]等许多传统机器学习方法和深度学习方法[17, 18]已被有效地用于构建化学基因组学的DTI预测模型中[19-21],这些模型需要学习药物相关信息(如药物的化学信息)、靶点相关信息(如蛋白质序列信息)和已知的DTI信息。有效获取药物和蛋白质的高级特征信息,对提高DTI预测的准确率有着重要的意义。传统机器学习方法均需要大量的专业和经验知识对药物和靶标进行特征表征和选择,即特征工程,特征工程的质量直接决定了预测模型的性能。而基于深度学习的DTI预测方法无须特征工程,即可自动学习输入的化合物与蛋白质数据中的抽象特征,发现复杂分子模式,并将学习到的抽象特征与复杂分子模式融合进行预测,因而比较有发展潜力。虽然如此,但是现有的基于深度学习的DTI预测方法普遍未能较好地处理蛋白质氨基酸数据中存在的大量冗余信息,获得的特征维度较高,从而在一定程度上限制了分类预测的准确率与效率。对此,Cheng等[22]基于图形注意力网络和多头自我注意力机制来预测DTI,使用注意力得分来考虑蛋白质中哪种氨基酸子序列对DTI预测更重要,获得了较好的DTI预测性能。该方法用图神经网络学习蛋白质结构,这需要假设蛋白质的高级结构稳定,然而蛋白质的高级结构实际上并不符合此假设。因此,进一步研究设计用于预测DTI的有效深度学习模型仍然很有吸引力。
事实上,由于用生化实验验证DTI非常费力和耗时,所以当前很多靶蛋白的已知配体很少,这意味着可利用的已知DTI信息较少,这种情况同时也限制了基于传统机器学习和深度学习方法的DTI预测性能[23]。对此,一些学者探索了在DTI信息的基础上融入药物相似性信息、异构DTIs网络的统计特征与网络特征信息,在一定程度上提高了DTI预测准确性[23-25]。Thafar等[23]通过使用带重启的随机行走算法来生成全局网络拓扑信息,并将全局网络拓扑信息应用于预测模型中;Xuan等[26]通过学习药物、蛋白质相关的多尺度属性和异质连接形成的全局拓扑结构而获得DTI特征,并进行预测建模。然而,关于药物和靶点的异构多源数据之间的相互联系、内部相似性和DTI的深层特征的有效整合仍是一个具有挑战性的问题[26, 27]。
自编码器是一种无监督的神经网络模型,可以学习到输入数据的隐含特征,并以此特征重构出原始输入数据。自编码器常被应用于数据压缩和特征提取中。近年来,有些学者将自编码器应用于解决DTI建模的相关问题。Sun等[28]建立了一个异构网络来整合药物、蛋白质和疾病的信息并进行DTI预测,该方法的原始药物特征通过多层编码器投影到嵌入(蛋白质)空间,并通过解码器进一步投影到标签(疾病)空间,以保持药物化学性质和功能的一致性。Ye等[29]通过图形自动编码器保留药物和靶标数据的重建信息,利用子空间层获得不同的强功能子集,并在集合层中全面利用这些强功能子集,从而可以更好地训练深度神经网络(DNN)进行DTI预测。Sun等[30]构建了一个药物目标异质网络,以整合与药物、靶标相关的各种连接,即药物之间或靶标之间的相似性和相互作用,以及药物与靶标之间的相互作用。该方法首先利用图形卷积自编码器在网络中深入集成不同类型的连接,在低维特征空间中靶向节点,然后利用对抗生成网络将节点的特征向量正规化为高斯分布,最后基于集合学习模型LightGBM构建了一个分类器来预测药物和靶标的相互作用,以抵消类不平衡的负面影响。Li等[31]将药物与靶点的关联预测视为链接预测,采用图卷积自编码器框架构建药物和靶点嵌入,然后应用双线性解码器重建DTI矩阵。Sajadi等[32]通过结合矩阵分解和去噪自编码器,同时从药物、靶点的结构信息和相互作用矩阵中学习其隐藏因子,并通过药物和靶点的相似性矩阵来解决相互作用矩阵的稀疏性问题。Li等[33]提出一种基于图形个性化传播的自动编码器模型来有效预测DTI,该方法将药物/蛋白质的相似性作为二部图的节点特征,通过构造一个图神经网络的编码器来聚集图中每个节点的邻域特征,以学习其图嵌入,药物和蛋白质节点被嵌入双线性解码器中,以已知DTI的预测得分与其标签之间的重建误差为训练目标进行图中每个药物-蛋白质对得分矩阵的计算。总的来说,以上研究在DTI预测相关问题上取得了较好的效果,但是现有的相关算法仍然无法满足产业应用对预测准确性和性能效率的需求。
在此背景下,本研究旨在围绕上述深度学习模型设计问题、药物-靶标对相关的深层特征有效学习和整合问题,研究并创建更有效的深度学习模型,以提高DTI预测能力。为此,本研究提出一种基于去噪自编码器融合相似度的DTI预测方法(Denoising Auto-Encoder and Similarity for Drug-Target Interactions,DAES-DTI),拟有效提取并融合药物、靶点数据之间的相互联系、内部相似性和DTI的深层特征来预测DTI,提高DTI预测性能,为新药研发和药物重新定位提供更优的DTI预测支持。
1 问题描述给定一组药物D={d1, d2, …, dn},一组靶标T={t1, t2, …, tm},以及一个已知药物和靶标之间相互作用矩阵Y∈Rn×m,其中n为药物数量,m为靶标数量,每项Yi, j∈{0, 1},i=1, 2, …, n; j=1, 2, …, m。如果药物di与靶标tj之间有已知的相互作用,则Yi, j的值为1,否则为0。DTI预测常被看成是一个二元分类问题,其目标是学习和使用上述D、T、Y和其他信息源的潜在特征来预测新的药物-靶标相互作用对。
2 材料与方法 2.1 材料 2.1.1 数据集本研究使用药物-靶标相互作用预测研究领域的Enzymes、Ion channels、GPCRs和Nuclear receptors等4个基准数据集[34]进行建模预测实验,这4个基准数据集的统计信息如表 1所示。
| 数据集 Dataset |
药物种数 Numbers of drugs |
靶标种数 Numbers of targets |
交互对数 Numbers of drug-target interaction |
| Enzymes | 445 | 664 | 2 926 |
| Ion channels | 210 | 204 | 1 476 |
| GPCRs | 223 | 95 | 635 |
| Nuclear receptors | 54 | 26 | 90 |
2.1.2 评价指标
本研究用DTI预测研究中最常用的评价指标AUC (Area Under the receiver operator characteristic Curve)、AUPR (Area Under the Precision-Recall curve)来评价模型的表现。具体地,AUC是分别由真阳性率(TPR)和假阳性率(FPR)为纵横坐标轴绘制的曲线下(与横坐标轴之间包含的区域)面积,AUPR是分别由精确率(Precision)和召回率(Recall)为纵横坐标轴绘制的曲线下(与横坐标轴之间包含的区域)面积。曲线下面积可通过积分的方法计算,有限样本情况下可通过有限梯形分解求和的方法计算,其中:
| $ \mathrm{TPR}=\frac{T P}{T P+F P}, $ | (1) |
| $ \mathrm{FPR}=\frac{F P}{F P+T N}, $ | (2) |
| $ \text { Precision }=\frac{T P}{T P+F P}, $ | (3) |
| $ \text { Recall }=\frac{T P}{T P+F N} 。$ | (4) |
为了评估本研究所提出的DTI预测模型DAES-DTI的有效性,本研究选择当前DTI预测领域9个较先进的模型,即aSDAE[32]、PPAEDTI[33]、AutoDTI ++[35]、CMF[36]、Bi-PSSM[37]、iDTI-ESBoost[38]、CNNDTI[39]、NFSPDTI[40]和EFMSDTI[41]等作为实验比较的基线模型,使用十折交叉验证方法做实验验证,并取模型5次重复运行结果的平均值作为最终结果。此外,与9个基线模型相同,本研究实验学习率设定为0.001,正则化系数λ设定为10-6,用Adam作为优化算法来训练DAES-DTI模型,其他主要超参数设置如表 2所示。特别地,本研究与9个较先进模型比较的数据全部引自相应的参考文献,并非复现这些模型重做实验获得。这9个较先进模型的实验参数设置详见参考文献[32, 33, 35-41]。本研究实验在配置了Intel 2.4 GHz*20的CPU、GeForce RTX2080Ti的GPU、64 GB的内存、64位的Ubuntu 18的计算机环境下进行。
| 超参数 Hyperparameter |
设置值 Setting value |
| Learning rate | 0.001 |
| Batch size | 256 |
| Hidden size | 512 |
| Number of hidden layers | 3 |
| Optimizer | Adam |
| Epoch | 300 |
| Gaussian noise | 0.000 01 |
2.2 方法 2.2.1 自编码器与去噪自编码器
自编码器(AutoEncoder)是一种使用反向传播算法将输出值尽可能逼近输入值的神经网络,它主要应用于无监督学习。自编码器的传播过程可以简单分为两部分:输入到隐层的编码(coding)过程和隐层到输出的解码(decoding)过程,且拥有对称的网络结构。编码是指自编码器先将输入压缩成隐层空间特征,然后通过这些特征重构输出,从而学习输入数据中的隐含表示;解码是指自编码器利用学习到的特征重构原始输入。自编码器可以对模型进行反向传播训练直至损失最小化,同时对数据尽可能精确地复制。通过这个过程,自编码器能学习到数据中的重要特征。
去噪自编码器(Denoising AutoEncoder,DAE)是在自编码器网络的基础上,为解决过拟合问题而在原始输入数据中加入噪声,从而使自编码器学习得到一个更加具有鲁棒性的特征表达[32]。如图 1所示,DAE将噪声引入原始数据后,编码器编码的是原始输入数据的受损坏版本(即包含了噪声)。DAE使用和其他传统神经网络相同的方式(本研究使用梯度下降的反向传播算法)进行训练。使用上述含噪声的数据集进行模型训练得到隐含层表示向量h,但是仍然是通过最小化原始输入数据与重构信号之间的误差来对网络参数进行调整。例如,图 1中的X1是由数据集样本X通过函数fnoise(·)产生的受损坏样本,DAE首先用编码器fen(·)对损坏的样本X1进行编码,然后用解码器fde(·)再对其特征进行解码,得到重构输出X′,最后再最小化网络输出X′和未损坏的原输入数据集样本X之间的损失Loss(X, X′),这样可使DAE网络提取到的特征更能反映原始输入的特点。本研究将使用DAE从破坏的药物与靶标数据输入中学习其隐含的潜在药物-靶标对特征。
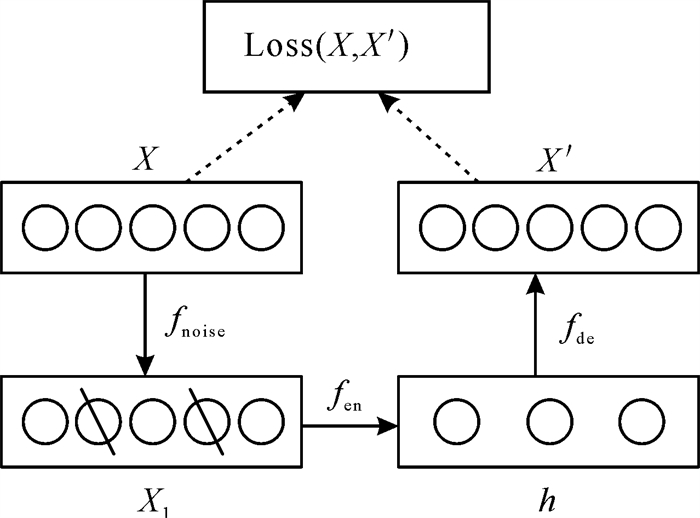
|
| 图 1 DAE模型示意图 Fig. 1 Schematic diagram of DAE model |
2.2.2 DAES-DTI的基本思想
本研究一方面利用DAE构建了一个药物-靶标对特征的无监督学习网络学习药物-靶标相互作用的深层特征,另一方面设计了一种新的非线性方法来提取药物之间和靶标之间的相似信息,然后将学习到的药物-靶标对特征信息与药物-药物、靶标-靶标的相似信息融合,以有效提取并融合药物、靶点数据之间的相互联系、内部相似性和药物-靶标相互作用的深层特征来预测药物-靶标相互作用。
基于以上思路,研究设计了DAES-DTI模型。该模型的数据处理过程如图 2所示。①在(Drug-)DAE模块中,首先,输入与目标药物(d)相互作用的所有靶标数据(xd);其次,向这些靶标数据注入高斯噪声后形成相应的受损靶标数据(x1d);再次,将受损靶标数据与目标药物的嵌入向量(Vd) 一起输入DAE的隐藏层中融合,通过编码器学习和提取药物-靶标交互特征;最后,通过解码器重构原始输入向量并评价重构损失。②在相似度计算模块中,首先设计相似度计算的指数函数,分别计算样本中药物之间的相似度和其对应靶标间的相似度;然后,使用平方损失评价预测相似度与真实相似度之间的差别,并将药物之间的相似度和其对应靶标间的相似度组合后输出。③首先将相似度损失与(Drug-)DAE模块的重构损失组合构成DAES-DTI模型的最终损失函数,然后利用误差反向传播学习算法迭代地训练模型,最后提取(Drug-)DAE模块的最中间层数据作为深层特征,并将其与相似度计算模块输出的相似度信息进行融合以构成最终的药物-靶标相互作用特征用于DTI建模预测。
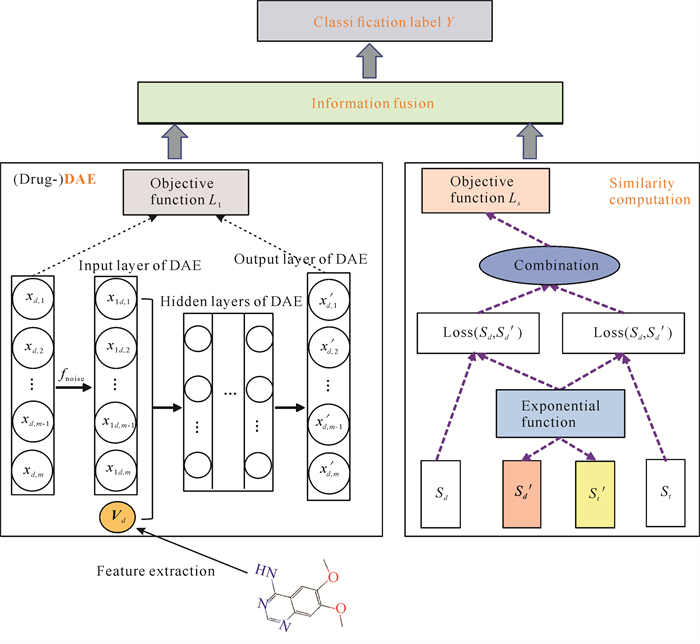
|
| 图 2 DAES-DTI模型的信处理过程示意图 Fig. 2 Schematic diagram of the information processing process of DAES-DTI model |
2.2.3 (Drug-)DAE模块的构建
DAES-DTI利用DAE同时从药物和靶标中学习潜在特征,而不是将它们分开学习,下面以(Drug-)DAE为例简述其构建过程。与一般DAE直接将受损数据输入其自编码器部件并重构原始输入的靶标数据不同,(Drug-)DAE将受损靶标数据和目标药物的嵌入向量(Vd) 在自编码器中的各隐藏层中融合并映射到一个特征空间进行药物-靶标交互特征的学习和提取,然后再通过解码器重构原始输入的靶标数据。具体地,(Drug-)DAE的网络结构(主要由一个输入层、若干隐藏层和一个输出层组成,图 3)阐述如下。
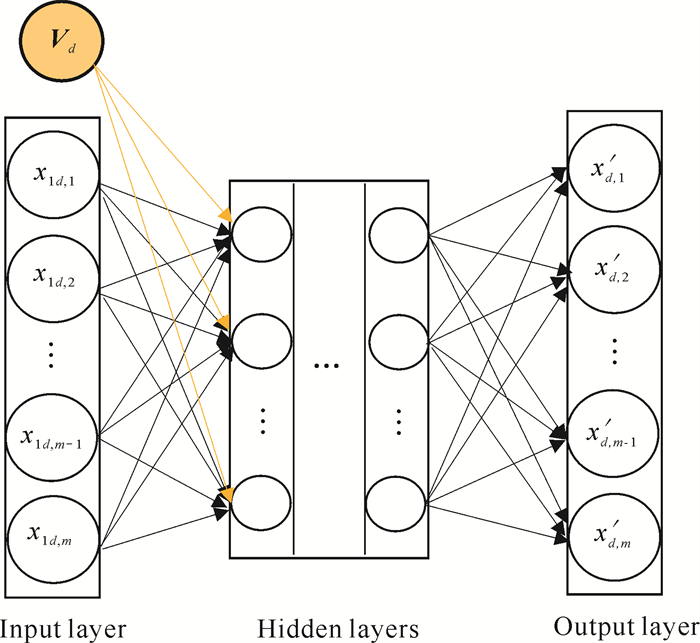
|
| 图 3 (Drug-)DAE网络结构示意图 Fig. 3 Schematic structure diagram of (Drug-)DAE network |
输入层:在输入层,将药物和靶标信息一起输送到DAE隐藏层,以便联合建模而不是传统的单独建模。首先,输入药物d的嵌入向量Vd及与其相互作用的所有靶标数据xd∈Rm。其中,药物d的嵌入向量Vd是该药物的SMILES串转换为其对应的ECFP4指纹的嵌入向量。每种药物都可以用一个ECFP4指纹描述,它是一个长度固定的二进制向量,以表示特定子结构是否存在。然后,在原始靶标数据xd中加入高斯噪声来生成受损坏的输入x1d,如式(5)所示:
| $ x_{1 d}=x_d+f_{\text {noise }}\left(x_d\right)=x_d+\varepsilon_d, $ | (5) |
其中,εd~N(0,σd),表示由fnoise(xd)生成的高斯噪声。
隐藏层:利用式(6)所示方法将药物和靶标信息转化为相应的潜在特征向量:
| $ z=a\left(W_x x_{1 d}+W_v V_d+b\right), $ | (6) |
其中,Wx∈Rk×m和Wv∈Rk×n表示隐藏层的权重,Vd∈Rn表示药物d的嵌入向量,b∈Rk、z∈Rk和a(·)分别表示隐藏层的偏置、输出值及激活函数,k为潜在特征维度。在本研究中,选择Sigmoid函数作为激活函数。
输出层:输出层的目标是利用潜在特征向量重构输入的原始靶标数据,如式(7)所示:
| $ x_d{ }^{\prime}=f\left(W^{\prime} z+b^{\prime}\right), $ | (7) |
其中,W′、b′和f(·)分别表示输出层的权重、偏置和映射函数(本研究选择恒等函数作为映射函数)。然后,定义(Drug-)DAE的目标函数如式(8)所示:
| $ \begin{aligned} & \quad L_1=\frac{1}{n} \sum\limits_{d=1}^n l\left(x_d-x_d^{\prime}\right)+\lambda\left(\left\|W_x\right\|^2+\right. \\ & \left.\left\|W^{\prime}\right\|^2+\left\|W_v\right\|^2\right), \end{aligned} $ | (8) |
其中,l(·)为误差函数,λ是正则化系数。
类似地,也可将靶标的潜在特征与该靶标相互作用的所有药物联系起来构建(Target-)DAE。(Target-)DAE的结构和计算过程与(Drug-)DAE的类似,只是原始输入数据为输入与给定靶标相互作用的所有药物的数据,本研究不再详细阐述。
2.2.4 相似信息的融入为了更有效地学习药物-靶标对的特征信息,本研究提出了一种新的非线性方法提取药物-药物、靶标-靶标的相似信息,以提高DTI预测模型性能。
现有线性计算方法主要通过对药物d与i,以及靶标t与j的潜在特征使用点积运算来估算它们之间的相似度[24]。令P ∈Rn×k、Q ∈Rm×k分别表示药物和靶标的潜在特征矩阵,则相似度s的计算如式(9)所示:
| $ s_{d, i}=\boldsymbol{p}_d \boldsymbol{p}_i, s_{t, j}=\boldsymbol{q}_t \boldsymbol{q}_j。$ | (9) |
为了更好地捕捉药物-药物、靶标-靶标之间的关系,本研究定义了如公式(10)、(11)所示的指数函数进行相似度s的计算:
| $ s_{d, i}=\exp \left(-\left\|\boldsymbol{p}_d-\boldsymbol{p}_i\right\|^2\right), $ | (10) |
| $ s_{t, j}=\exp \left(-\left\|\boldsymbol{q}_t-\boldsymbol{q}_j\right\|^2\right), $ | (11) |
同时使用平方损失评价预测相似度与真实相似度之间的差别,如式(12)所示:
| $ \left.L_s=\lambda_d\left\|\boldsymbol{S}_d-\boldsymbol{S}_d^{\prime}\right\|^2+\lambda_t\left\|\boldsymbol{S}_t-\boldsymbol{S}_t^{\prime}\right\|^2\right), $ | (12) |
其中,Sd和St分别表示药物-药物和靶标-靶标的真实相似度矩阵,Sd′和St′分别表示药物-药物和靶标-靶标的预测相似度矩阵。最后,将相似信息损失Ls与DAE的损失L1组合构成DAES-DTI模型的最终损失函数L,如式(13)所示:
| $ L=L_1+L_s 。$ | (13) |
DAE隐藏层的层数是DAES-DTI中的一个重要超参数。为了探究DAE隐藏层的层数对DAES-DTI性能的影响,在Enzymes、Ion channels、GPCRs和Nuclear receptors 4个基准数据集上分别设置了(Drug-)DAE网络隐藏层的层数为1、3、5、7来进行实验比较,结果如图 4所示。可见,隐藏层的层数从1变化到7的过程,所有数据集上的AUC都先迅速增加,然后再逐渐下降。当隐藏层的层数为3时,模型在各数据集的性能几乎达到最佳状态;但在数据集Enzymes和Ion channels上隐藏层的层数为3和5时,DAES-DTI的性能几乎相当。这表明在(Drug-)DAE网络的隐藏层层数为1时DAES-DTI处于欠拟合状态,层数为7时DAES-DTI处于过拟合状态,层数为5时在样本较少的数据集GPCRs和Nuclear receptors上DAES-DTI处于过拟合状态,因而这些情况下DAES-DTI的AUC值比层数为3时更低。因此,DAES-DTI在与基线模型的比较实验中设定隐藏层的层数为3。
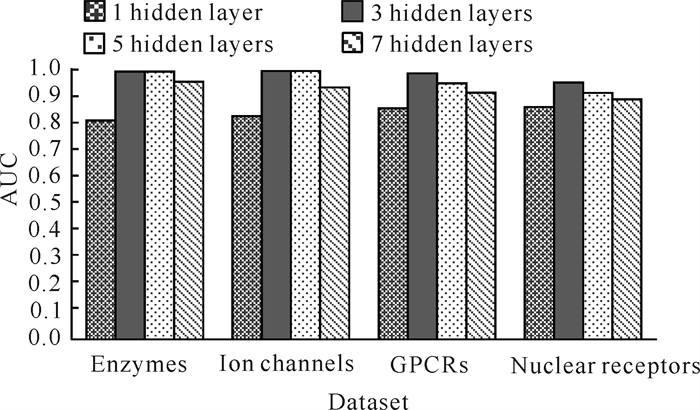
|
| 图 4 不同隐藏层层数的(Drug-)DAE对DAES-DTI性能的影响 Fig. 4 Effect of (Drug-)DAE with different number of hidden layers on DAES-DTI performance |
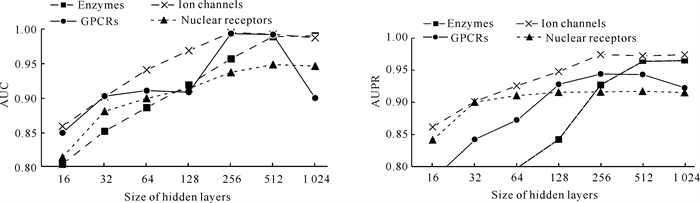
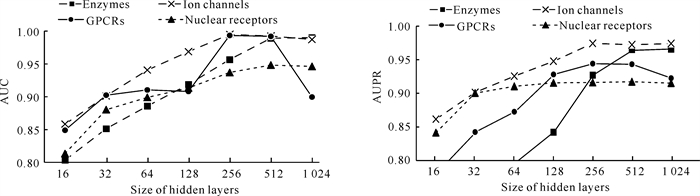
DAE隐藏层的大小(即隐藏层的神经元数量)是DAES-DTI能从输入中学习有用的潜在特征的一个关键因素。为了探究DAE隐藏层的大小对DAES-DTI性能的影响,在4个基准数据集上分别选择了神经元数量为16、32、64、128、256、512、1 024的隐藏层进行实验,结果如图 5、图 6所示。随着隐藏层的神经元数量的增加,所有数据集上的AUC和AUPR值先迅速增加,然后缓慢下降。当隐藏层的神经元数量小于128时,DAES-DTI处于欠拟合的状态,无法通过测试数据达到最佳效果;而当隐藏层的神经元数量大于1 024时,DAES-DTI处于过拟合状态,即模型过于复杂,难以从高度稀疏的交互矩阵中学习到丰富、有效的特征。这表明选择适当的隐藏层的大小实际上是在欠拟合状态和过拟合状态之间进行权衡。(Drug-)DAE的神经元数量大于512后,DAES-DTI在4个数据集上的AUC值均不同程度下降,AUPR值在3个数据集上变化不大但在GPCRs数据集上明显下降;(Target-)DAE的神经元数量大于512后,DAES-DTI在3个数据集(Ion channels、GPCRs、Nuclear receptors)上的AUC值和2个数据集(GPCRs和Nuclear receptors)上的AUPR值均不同程度下降,而在Enzymes数据集上的AUC值和2个数据集(Enzymes和Ion channels)上的AUPR值变化不明显。因此,DAES-DTI在与基线模型的比较实验中设定隐藏层的神经元数量为512。
|
| 图 5 不同隐藏层大小的(Drug-)DAE对DAES-DTI性能的影响 Fig. 5 Effect of (Drug-)DAE with different hidden layer sizes on DAES-DTI performance |
|
| 图 6 不同隐藏层大小的(Target-)DAE对DAES-DTI性能的影响 Fig. 6 Effect of (Target-)DAE with different hidden layer sizes on DAES-DTI performance |
3.2 与较先进模型的性能比较
表 3和表 4分别给出了各模型在4个基准数据集上所获得的AUC和AUPR值。与各基线模型相比,DAES-DTI(Drug)在4个基准数据集上获得的AUC和AUPR值明显优于所比较的PPAEDTI、AutoDTI++、CMF、Bi-PSSM、ESBoost、CNNDTI、NFSPDTI和EFMSDTI等8个较先进模型所得的,并与另一先进模型aSDAE获得的相当(文献[32]中仅给出了两位有效小数,通过小数点后第3位四舍五入取得,因而无法精确比较)。这表明DAES-DTI(Drug)能较有效、准确地预测药物-靶标相互作用。各基线模型在Nuclear receptors数据集上的性能表现基本上是4个基准数据集中最差的,这可能是因为Nuclear receptors数据集包含的药物和靶标的数量最少,以致模型能学习到的有用特征信息也较少,从而让模型的性能表现较差。此外,将每种药物的嵌入向量和与其相互作用的所有靶标的表征集合作为输入的DAES-DTI,即DAES-DTI (Drug),在Enzymes、Ion channels、GPCRs 3个数据集上的AUC和AUPR性能都优于将每种靶标的嵌入向量和与其相互作用的所有药物的表征集合作为输入的DAES-DTI,即DAES-DTI (Target);而在Nuclear receptors数据集上,DAES-DTI (Target)的表现优于DAES-DTI (Drug)。这可能是由于靶标分子结构比小分子结构更复杂,更难提取对药物-靶标相互作用有影响的靶标分子特征信息,在这种情况下,DAES-DTI (Target)在靶标样本数较少的Nuclear receptors数据集上通过将每种靶标的潜在特征都融入到DAE的药物-靶标对特征学习中,使模型可获得更丰富的、对药物-靶标相互作用预测有影响的靶标分子特征信息。
| 模型 Model |
Enzymes | Ion channels | GPCRs | Nuclear receptors |
| PPAEDTI | 95.56 | 95.35 | 90.15 | 82.01 |
| AutoDTI ++ | 0.90 | 0.91 | 0.86 | 0.87 |
| CMF | 0.960 5 | 0.976 6 | 0.957 5 | 0.920 4 |
| Bi-PSSM | 0.948 0 | 0.989 0 | 0.872 0 | 0.869 0 |
| ESBoost | 0.968 9 | 0.936 9 | 0.933 2 | 0.928 5 |
| CNNDTI | 0.980 2 | 0.975 4 | 0.951 3 | 0.896 5 |
| NFSPDTI | 0.98 | 0.99 | 0.98 | 0.99 |
| EFMSDTI | 0.984 4 | 0.971 3 | 0.981 7 | 0.944 5 |
| aSDAE | 0.99 | 0.99 | 0.98 | 0.90 |
| DAES-DTI (Drug) | 0.990 6 | 0.992 3 | 0.984 2 | 0.949 2 |
| DAES-DTI (Target) | 0.988 5 | 0.972 4 | 0.983 5 | 0.955 4 |
| Note: the original results of PPAEDTI are obtained from the 5-fold cross-test.The best results of the comparative methods on the corresponding datasets are highlighted in bold. | ||||
| 模型 Model |
Enzymes | Ion channels | GPCRs | Nuclear receptors |
| PPAEDTI | - | - | - | - |
| AutoDTI ++ | 0.82 | 0.90 | 0.85 | 0.84 |
| CMF | 0.851 4 | 0.933 3 | 0.902 4 | 0.900 3 |
| Bi-PSSM | 0.546 0 | 0.390 2 | 0.282 0 | 0.411 0 |
| ESBoost | 0.686 8 | 0.480 7 | 0.500 5 | 0.791 0 |
| CNNDTI | 0.918 8 | 0.948 7 | 0.908 1 | 0.903 4 |
| NFSPDTI | 0.87 | 0.87 | 0.69 | 0.91 |
| EFMSDTI | 0.923 4 | 0.956 2 | 0.911 7 | 0.904 4 |
| aSDAE | 0.97 | 0.98 | 0.94 | 0.93 |
| DAES-DTI (Drug) | 0.965 7 | 0.974 1 | 0.943 1 | 0.917 9 |
| DAES-DTI (Target) | 0.935 7 | 0.973 5 | 0.933 1 | 0.935 3 |
| Note: the original literature presented PPAEDTI did not provide results for the AUPR values.The best results of the comparative methods on the corresponding datasets are highlighted in bold. | ||||
3.3 非线性相似度计算方法的有效性实验
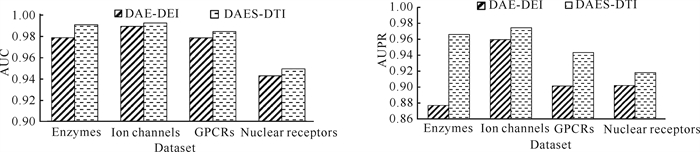
相似度计算能获取药物-药物和靶标-靶标关系的额外信息。为了探究相似度计算方法对DAES-DTI的有效性,本研究首先将DAES-DTI与其去除非线性相似度计算部件后的DTI预测模型(本研究称之为DAE-DTI)进行对比;接着,令λd=λt,并设置它们的取值为{0,10-1,10-2,10-3,10-4,10-5,10-6},并以在DAE-DTI中加入CMF模型[24]的线性相似度计算方法作为对照组,与在DAE-DTI中加入本研究提出的非线性计算方法的模型进行比较(以AUPR作为评价指标)。如图 7所示,无论用AUC还是用AUPR作为评价指标,DAES-DTI去除非线性相似度计算部件后,在所有数据集上的DTI预测性能均显著低于原DAES-DTI。这说明本研究提出的非线性相似度计算在模型中具有积极的意义,对提高模型的性能是有效的。由表 5可知,在4个基准数据集上,与在DAE中加入了CMF模型的线性相似度计算方法比较,DAES-DTI几乎都能获得更高的性能。上述结果表明,本研究提出的非线性相似度计算方法有更强的从相似信息中提取有用信息的能力。
|
| 图 7 非线性相似度计算部件对模型性能的影响 Fig. 7 Effect of nonlinear similarity calculation components on model performance |
| 数据集 Dataset |
相似度计算方法 Similarity calculation method |
AUPR | ||||||
| λ=0 | λ=10-1 | λ=10-2 | λ=10-3 | λ=10-4 | λ=10-5 | λ=10-6 | ||
| Enzymes | CMF | 0.963 2 | 0.964 7 | 0.960 1 | 0.965 8 | 0.963 7 | 0.967 6 | 0.968 1 |
| Our method | 0.963 2 | 0.967 3 | 0.963 2 | 0.966 5 | 0.970 8 | 0.968 9 | 0.968 2 | |
| Ion channels | CMF | 0.969 2 | 0.970 3 | 0.970 7 | 0.971 2 | 0.972 2 | 0.973 1 | 0.972 2 |
| Our method | 0.969 2 | 0.970 6 | 0.971 6 | 0.972 6 | 0.972 3 | 0.974 1 | 0.974 6 | |
| GPCRs | CMF | 0.932 7 | 0.933 3 | 0.934 2 | 0.928 1 | 0.930 2 | 0.933 4 | 0.936 1 |
| Our method | 0.932 7 | 0.931 6 | 0.934 4 | 0.939 2 | 0.940 8 | 0.942 9 | 0.944 5 | |
| Nuclear receptors | CMF | 0.912 1 | 0.918 0 | 0.912 1 | 0.913 4 | 0.910 1 | 0.915 3 | 0.915 4 |
| Our method | 0.912 1 | 0.913 5 | 0.912 9 | 0.913 7 | 0.918 5 | 0.918 3 | 0.917 9 | |
因此,图 7和表 5的结果表明了本研究提出的非线性相似度计算方法对改进DTI预测模型是有效的。此外,由表 5可知,当正则化系数λ的取值小于10-4时对实验结果略有改善,当λ的取值大于10-4时对实验结果产生不利影响。因此,为了获得良好的性能,选择一个合适的正则化系数值是至关重要的。
4 结论本研究提出了DAES-DTI新模型来预测药物-靶标相互作用。DAES-DTI模型使用去噪自编码器的无监督深度学习方法来学习药物-靶标对特征信息,然后融入一种非线性相似度计算方法计算所得的药物-药物、靶标-靶标相似性信息,最后用它们进行药物-靶标相互作用预测。在Enzymes、Ion channels、GPCRs和Nuclear receptors 4个基准数据集上与9个较先进的相关模型的比较实验结果表明,本研究所提出的模型显著优于所比较的PPAEDTI、AutoDTI++、CMF、Bi-PSSM、ESBoost、CNNDTI、NFSPDTI和EFMSDTI等8个较先进模型,并与另一先进模型aSDAE相当。
未来,研究计划通过添加靶标的氨基酸序列等一些额外的信息来扩展DAES-DTI模型,同时拟使用DAES-DTI模型来开发与癌症药物重新定位相关的真实案例研究,并通过实验验证模型的选定预测,以证明结果的临床相关性。
| [1] |
李扬. 基于集成学习方法的药物-靶标相互作用预测及应用[D]. 西安: 西京学院, 2019.
|
| [2] |
MULLARD A. New drugs cost US $2.6 billion to develop[J]. Nature Reviews Drug Discovery, 2014, 13(12): 877. |
| [3] |
ROSES A D. Pharmacogenetics in drug discovery and development: a translational perspective[J]. Nature Reviews Drug Discovery, 2008, 7(10): 807-817. DOI:10.1038/nrd2593 |
| [4] |
ASHBURN T T, THOR K B. Drug repositioning: identifying and developing new uses for existing drugs[J]. Nature Reviews Drug Discovery, 2004, 3(8): 673-683. DOI:10.1038/nrd1468 |
| [5] |
KURUVILLA F G, SHAMJI A F, STERNSON S M, et al. Dissecting glucose signalling with diversity-oriented synthesis and small-molecule microarrays[J]. Nature, 2002, 416(6881): 653-657. DOI:10.1038/416653a |
| [6] |
KEISER M J, ROTH B L, ARMBRUSTER B N, et al. Relating protein pharmacology by ligand chemistry[J]. Nature Biotechnology, 2007, 25(2): 197-206. DOI:10.1038/nbt1284 |
| [7] |
CHENG A C, COLEMAN R G, SMYTH K T, et al. Structure-based maximal affinity model predicts small-molecule druggability[J]. Nature Biotechnology, 2007, 25(1): 71-75. DOI:10.1038/nbt1273 |
| [8] |
ZHU S F, OKUNO Y, TSUJIMOTO G, et al. A probabilistic model for mining implicit 'chemical compound-gene' relations from literature[J]. Bioinformatics, 2005, 21(Suppl 2): ii245-ii251. |
| [9] |
MOUSAVIAN Z, MASOUDI-NEJAD A. Drug-target interaction prediction via chemogenomic space: learning-based methods[J]. Expert Opinion on Drug Metabolism & Toxicology, 2014, 10(9): 1273-1287. |
| [10] |
WU Z R, CHENG F X, LI J, et al. SDTNBI: an integrated network and chemoinformatics tool for systematic prediction of drug-target interactions and drug repositioning[J]. Briefings in Bioinformatics, 2017, 18(2): 333-347. |
| [11] |
TANG D Y, CAO D, ZHAO J. Predicting drug-target interaction using support vector machine and invasive tumor growth optimization[J]. International Journal of Hybrid Information Technology, 2017, 10(9): 41-50. DOI:10.14257/ijhit.2017.10.9.04 |
| [12] |
XIAO X, MIN J L, WANG P, et al. ICDI-PseFpt: identify the channel-drug interaction in cellular networking with PseAAC and molecular fingerprints[J]. Journal of Theoretical Biology, 2013, 337: 71-79. DOI:10.1016/j.jtbi.2013.08.013 |
| [13] |
CHEN T, GUESTRIN C. XGBoost: a scalable tree boo- sting system[C]//Proceedings of the 22nd ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. New York: Association for Computing Machinery, 2016: 785-794.
|
| [14] |
SHIM J, HONG Z Y, SOHN I, et al. Prediction of drug-target binding affinity using similarity-based convolutional neural network[J]. Scientific Reports, 2021, 11(1): 4416. DOI:10.1038/s41598-021-83679-y |
| [15] |
WANG L, YOU Z H, CHEN X, et al. RFDT: a rotation forest-based predictor for predicting drug-target interactions using drug structure and protein sequence information[J]. Current Protein & Peptide Science, 2018, 19(5): 445-454. |
| [16] |
ZHANG J, ZHU M C, CHEN P, et al. DrugRPE: random projection ensemble approach to drug-target interaction prediction[J]. Neurocomputing, 2017, 228: 256-262. DOI:10.1016/j.neucom.2016.10.039 |
| [17] |
ZHOU J B, LI S L, HUANG L, et al. Distance-aware molecule graph attention network for drug-target binding affinity prediction[Z/OL]. arXiv Preprint, 2020: 1-12[2022-08-15]. https://arxiv.org/pdf/2012.09624.
|
| [18] |
YOU J Y, MCLEOD R D, HU P Z. Predicting drugtarget interaction network using deep learning model[J]. Computational Biology and Chemistry, 2019, 80: 90-101. DOI:10.1016/j.compbiolchem.2019.03.016 |
| [19] |
JIN G X, WONG S T C. Toward better drug repositioning: prioritizing and integrating existing methods into efficient pipelines[J]. Drug Discovery Today, 2014, 19(5): 637-644. DOI:10.1016/j.drudis.2013.11.005 |
| [20] |
BAGHERIAN M, SABETI E, WANG K, et al. Machine learning approaches and databases for prediction of drug-target interaction: a survey paper[J]. Briefings in Bioinformatics, 2021, 22(1): 247-269. DOI:10.1093/bib/bbz157 |
| [21] |
LAN K, WANG D T, FONG S, et al. A survey of data mining and deep learning in bioinformatics[J]. Journal of Medical Systems, 2018, 42(8): 139. DOI:10.1007/s10916-018-1003-9 |
| [22] |
CHENG Z J, YAN C, WU F X, et al. Drug-target interaction prediction using multi-head self-attention and graph attention network[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2022, 19(4): 2208-2218. DOI:10.1109/TCBB.2021.3077905 |
| [23] |
THAFAR M A, OLAYAN R S, ASHOOR H, et al. DTiGEMS+: drug-target interaction prediction using graph embedding, graph mining, and similarity-based techniques[J]. Journal of Cheminformatics, 2020, 12(1): 44. DOI:10.1186/s13321-020-00447-2 |
| [24] |
LEE I, NAM H. Identification of drug-target interaction by a random walk with restart method on an interactome network[J]. BMC Bioinformatics, 2018, 19(Suppl 8): 208. |
| [25] |
LI X S, MA D C, REN Y, et al. Large-scale prediction of drug-protein interactions based on network information[J]. Current Computer-Aided Drug Design, 2022, 18(1): 64-72. DOI:10.2174/1573409917666210315094213 |
| [26] |
XUAN P, HU K, CUI H, et al. Learning multi-scale heterogeneous representations and global topology for drug-target interaction prediction[J]. IEEE Journal of Biomedical and Health Informatics, 2022, 26(4): 1891-1902. DOI:10.1109/JBHI.2021.3121798 |
| [27] |
XU X Q, XUAN P, ZHANG T G, et al. Inferring drug-target interactions based on random walk and convolutional neural network[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2022, 19(4): 2294-2304. DOI:10.1109/TCBB.2021.3066813 |
| [28] |
SUN C, CAO Y K, WEI J M, et al. Autoencoder-based drug-target interaction prediction by preserving the consistency of chemical properties and functions of drugs[J]. Bioinformatics, 2021, 37(20): 3618-3625. DOI:10.1093/bioinformatics/btab384 |
| [29] |
YE Q, ZHANG X L, LIN X L. Drug-target interaction prediction via graph auto-encoder and multi-subspace deep neural networks[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2022, 20(5): 2647-2658. |
| [30] |
SUN C, XUAN P, ZHANG T G, et al. Graph convolutional autoencoder and generative adversarial network-based method for predicting drug-target interactions[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2022, 19(1): 455-464. DOI:10.1109/TCBB.2020.2999084 |
| [31] |
LI J, ZHANG C, LI Z W, et al. GCMCDTI: graph convolutional autoencoder framework for predicting drug-ktarget interactions based on matrix completion[J]. Journal of Bioinformatics and Computational Biology, 2022, 20(5): 2250023. DOI:10.1142/S0219720022500238 |
| [32] |
SAJADI S Z, ALI ZARE CHAHOOKI M, TAVAKOL M, et al. Matrix factorization with denoising autoencoders for prediction of drug-target interactions[J]. Molecular Diversity, 2023, 27(3): 1333-1343. DOI:10.1007/s11030-022-10492-8 |
| [33] |
LI Y C, YOU Z H, YU C Q, et al. PPAEDTI: personalized propagation auto-encoder model for predicting drug-target interactions[J]. IEEE Journal of Biomedical and Health Informatics, 2023, 27(1): 573-582. DOI:10.1109/JBHI.2022.3217433 |
| [34] |
YAMANISHI Y, ARAKI M, GUTTERIDGE A, et al. Prediction of drug-target interaction networks from the integration of chemical and genomic spaces[J]. Bioinformatics, 2008, 24(13): i232-i240. DOI:10.1093/bioinformatics/btn162 |
| [35] |
SAJADI S Z, ALI ZARE CHAHOOKI M, GHARAGHANI S, et al. AutoDTI++: deep unsupervised learning for DTI prediction by autoencoders[J]. BMC Bioinformatics, 2021, 22(1): 204. DOI:10.1186/s12859-021-04127-2 |
| [36] |
ZHENG X D, DING H, MAMITSUKA H, et al. Collaborative matrix factorization with multiple similarities for predicting drug-target interactions[C]//GHANI R, SENATOR T E, BRADLEY P, et al. Proceedings of the 19th ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. New York: Association for Computing Machinery, 2013: 1025-1033.
|
| [37] |
MOUSAVIAN Z, KHAKABIMAMAGHANI S, KAV-OUSI K, et al. Drug-target interaction prediction from PSSM based evolutionary information[J]. Journal of Pharmacological and Toxicological Methods, 2016, 78: 42-51. DOI:10.1016/j.vascn.2015.11.002 |
| [38] |
RAYHAN F, AHMED S, SHATABDA S, et al. iDTI-ESBoost: identification of drug target interaction using evolutionary and structural features with boosting[J]. Scientific Reports, 2017, 7(1): 17731. DOI:10.1038/s41598-017-18025-2 |
| [39] |
HU S S, ZHANG C L, CHEN P, et al. Predicting drug-target interactions from drug structure and protein sequence using novel convolutional neural networks[J]. BMC Bioinformatics, 2019, 20(Suppl 25): 689. |
| [40] |
刘芳. 基于网络模型的药物靶标相互作用预测方法研究[D]. 长沙: 湖南大学, 2021.
|
| [41] |
ZHANG Y Y, WU M J, WANG S D, et al. EFMSDTI: drug-target interaction prediction based on an efficient fusion of multi-source data[J]. Frontiers in Pharmacology, 2022, 13: 1009996. DOI:10.3389/fphar.2022.1009996 |



