肿瘤异质性是指恶性肿瘤在生长过程中,经过多次分裂增殖,由于子细胞的改变导致肿瘤细胞生长速度、侵袭能力和药敏性等多方面存在差异的现象[1]。异质性是导致不同组织功能差异化的关键因素,在细胞生理调控过程中发挥着重要的作用。近年来,随着组学技术的迅速发展,科学研究能够逐步揭示肿瘤异质性的部分特征,为癌症的分子分型、诊断和治疗提供了重要的理论基础,也为癌症患者的精准医疗提供了关键的技术手段。传统的批量转录组(Bulk RNA-seq)通常是针对整个器官或组织测序,反映的是样本所有细胞的整体基因表达水平,无法捕捉到细胞间基因表达的异质性。单细胞转录组(Single-cell transcriptomics)虽然可以揭示细胞个体间异质性,但是却也受技术限制而无法获得细胞的空间信息,阻碍了细胞空间位置与功能关系的进一步研究。为获取带有空间信息的高通量转录组数据,诞生了新的技术手段“空间转录组(Spatial Transcriptomics,ST)技术”。
空间转录组技术大概分为微解剖基因表达技术、原位杂交技术、原位测序技术、原位捕获技术4类[2]。微解剖基因表达技术主要依赖于显微切割方法,直接捕获组织中特定空间位置的细胞,但该技术只能确定特定类型细胞或组织区域的空间位置,无法确定各个细胞的具体空间位置,相关研究有Kruse等[3]提出的Tomo-seq、Chen等[4]提出的Geo-seq、Nichterwitz等[5]提出的LCM-seq等。原位杂交技术利用标记探针与目标转录本进行互补杂交,从而达到可视化的目的,但其本质上基于光学图像技术,容易受衍射极限的影响,相关研究有Chen等[6]提出的smFISH、Eng等[7]提出的seqFISH和seqFISH+、Xia等[8]提出的MERFISH等。原位测序技术利用微米或纳米级DNA球放大信号测序,但该技术受细胞固有位置限制,仅仅能区分有限数量的转录本,相关研究有Gyllborg等[9]提出的HybISS、Lee等[10]提出的FISSEQ、Alon等[11]提出的Exseq、Fürth等[12]提出的INSTA-seq等。原位捕获技术利用带有空间条形码的特殊引物来捕获具有空间位置信息的转录本, 如2016年St-hl等[13]在《Science》杂志上发表的论文中首次提出的“空间转录组技术”。空间转录组技术将空间位置识别序列(空间条形码)、捕获引物等固定于捕获芯片区域(Spot),将芯片与组织切片贴附,经过染色、成像、透化等操作捕获mRNA,再经过逆转录生成带有空间编码的cDNA并扩增建库,然后进行高通量测序,最终获得切片与芯片接触部位细胞内包含空间位置信息的转录组数据。随后,Rodriques等[14]于2019年推出Slide-seq。2020年,耶鲁大学Liu等[15]推出DBiT-seq。2021年美国密歇根大学的Cho等[16]推出基于Illumina测序的空间转录组技术Seq-Scope。同年,我国深圳华大生命科学研究院Chen等[17]推出国内自主研发的空间转录组技术——Stereo-seq,该技术利用DNA纳米球(DNA Nano Ball,DNB)制备空间编码阵列,通过芯片捕获组织中的mRNA,并通过唯一条形码还原空间位置,实现了高通量、超高分辨率的组织原位测序。空间转录组技术的发展使得对转录信息和空间信息的总体无偏差访问成为可能,然而大样本转录组数据带来了新的存储、计算问题,突破算法运算瓶颈对转录组数据进行深度解析应用具有重要的意义。本研究基于Stereo-seq测序数据,研究一种针对空间转录组的快速定位算法。针对容错处理时间复杂度较高导致算法定位耗时的不足,本研究提取空间条形码的部分碱基作为分类依据,分块读取解压序列数据,设计高效的FASTQ序列解析算法,从而提高算法的定位速度。
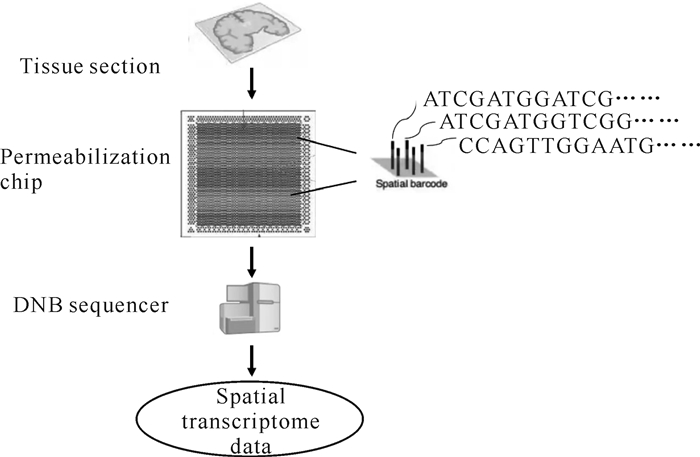
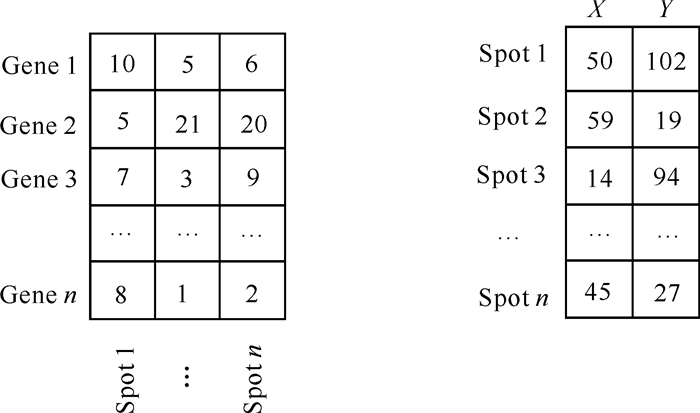
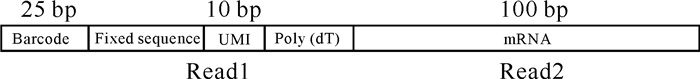
1 相关概念 1.1 Stereo-seqStereo-seq通过空间条形码获取转录本测序的过程如图 1所示。首先,将组织标本切片放置于载玻片上,通过生化处理后将组织放到测序芯片上并采用标记探针捕获mRNA;然后,将释放出来的mRNA与带有空间条形码的寡核苷酸结合逆转录成cDNA;最后,对带有空间条形码的cDNA进行测序并生成空间转录组数据,生成的空间转录组数据如图 2所示。采用双端测序构建文库,其结构如图 3所示,读段1(Read1)包含25 bp的空间条形码、10 bP的唯一分子标识符(UMI)等部分,读段2(Read2)为100 bp的mRNA。为方便计算机处理序列数据,碱基序列通常采用含有A(腺嘌呤)、C(胞嘧啶)、G(鸟嘌呤)、T(胸腺嘧啶)、N(未识别或无法识别的碱基)字符的文本串S来表示,如S=ATCGATGGATCG……。
|
| 图 1 空间转录组技术Stereo-seq测序流程 Fig. 1 The sequencing process of spatial transcriptome technology Stereo-seq |
|
| 图 2 空间转录组数据示例 Fig. 2 Example of spatial transcriptome data |
|
| 图 3 Stereo-seq测序文库结构 Fig. 3 Stereo-seq library structure |
1.2 FASTQ格式
在计算过程中,处理的基因序列一般较短,长度通常在几十至几百碱基。FASTQ是一种存储生物信息学测序数据的文件格式,保存生物序列及其对应的碱基质量分数。以本研究实验数据集的Read1序列为例,其格式示例如图 4所示。

|
| 图 4 FASTQ格式示例 Fig. 4 Example of FASTQ format |
FASTQ格式通常包含以下4行。
第1行:序列ID,以@开始,后面跟着序列名称和测序仪器输出文件名;
第2行:读段,由A、C、T、G、N字符组成;
第3行:以+开头,之后加其他描述信息(可选);
第4行:碱基质量值,以ASCII码表示,每个字符代表一个碱基的质量Q,假设测序误差概率为Pr,则质量Q的计算如式(1)所示:
| $ Q=-10 \times \lg \left(P_r\right) \text { 。} $ | (1) |
为减少近似匹配的计算开销,目前主要有3类映射(Mapping)索引被广泛应用于生物信息比对:后缀数组(Suffix Array,SA)、FM-index(Full-text Minute index,一种用于高效搜索和压缩全文数据的索引结构)、哈希索引。
对于一个文本串S,SA以字典序存储了该文本串的所有位置。对于模式串P,所有以P为前缀的位置都被连续存储于区间SA[m, n],该区间包含了所有模式串P在文本串S中的匹配位置[18]。SA能够快速查找模式串的所有匹配位置,但其空间开销较大。
FM-index是一种集合BWT[19](Burrows-Wheeler Transform,一种数据预处理算法)和一些小型辅助数据结构的全文本压缩索引,包含了BWT(S)、采样数据和一个简化SA,能够高效地统计模式串出现的数量并定位该模式串出现的所有位置。为减少空间消耗,FM-index仅存储部分信息,每隔一定距离设立一个采样位置(checkpoint)。当定位模式串P时,由于事先存储了采样位置,P的采样位置可以通过检索数组直接得到,非采样位置LF(i)则需要多次执行LF-mapping操作才能计算出具体位置,具体计算如式(2)所示,其中C[BWT(S)[i]]表示字典序小于BWT(S)[i]的碱基数目,rankBWT(S)[i](BWT(S), i)表示返回BWT(S)[i]对应碱基在BWT(S)[0, i-1]区间出现的次数[18]。FM-index需要支持两个基本操作:计数、定位,即索引结果需要返回匹配成功的次数和所有匹配成功的下标位置。FM-index能够以较小的存储开销达到良好的性能,但是其受限于耗时的定位操作。尽管选取了采样位置存储部分信息的策略,但是非采样位置的还原仍需要执行大量的计算步骤,并且计算次数取决于采样策略与采样距离,当相邻两步操作的访问非连续时,定位算法的运行时间会显著增加。
| $ \begin{aligned} & L F(i)=C[B W T(S)[i]]+ \\ & \operatorname{rank}_{B W T(S)[i](B W T(S), i)} 。\end{aligned} $ | (2) |
哈希索引将短序列映射到参考序列的候选位置,被广泛应用于序列定位算法中,如minimap2[20]。目前,只有少部分算法采用了SA,在序列定位比对中应用更广泛的是FM-index和哈希索引[21]。以人类基因(约3×109 bp)为例,采用SA需要超过12 GB的空间开销,而采用FM-index仅需要约3 GB空间开销。SA与哈希索引的空间开销相近,但哈希索引有更高的查询效率。FM-index与哈希索引相比,FM-index在空间开销上具有巨大的优势,但FM-index定位耗时,更适合于小字符集数据的快速匹配,而哈希索引定位操作的时间复杂度为常数级。哈希索引是生物信息领域最流行的索引技术[21],哈希数据结构可以有效地处理大规模数据的查询,并且可以在多个计算单元上进行,具有良好的计算并行效率,因此本研究采用此索引结构。
2 方法 2.1 数据处理框架及其优化生产者-消费者模型作为一种经典的并发编程模型,被广泛应用于生物信息计算领域,其典型的工作模型如图 5所示。生产者读取输入的转录组序列数据,并以数据块形式分发到队列中,各消费者按照先进先出(FIFO)调度原则处理数据块。这种简单模型的性能主要取决于消息队列大小和两者之间的效率平衡关系。具体来说,当生产者的生产效率优于消费者的处理效率时,消费者来不及处理任务,那么通过限制消息队列大小的方式阻塞生产者,消费者依然能够执行数据处理。然而,当生产者效率劣于消费者时,会导致只有小部分消费者执行任务、大部分消费者处于空闲的情况,即消费者处于“饥饿”状态。这种效率失衡的模式,无法满足生物信息计算这种读写(I/O)密集程序的效率需求。

|
| 图 5 典型生产者-消费者模型 Fig. 5 Typical producer-consumer model |
多线程开发可以有效地利用多核平台的优势来提升性能。然而,对于I/O型生物信息计算,其性能瓶颈可能会出现在数据的读取和解析上。尽管目前的硬盘读写速度越来越快,但是经过并行优化后的程序处理速度可能会明显超过硬盘的读取速度,从而导致应用程序从计算瓶颈转变为I/O瓶颈。即使继续提高计算性能,运行速度也可能不会有明显的提升。因此,在多线程开发中,需要特别关注I/O密集型生物信息计算的性能,以确保应用程序的整体性能得到有效提升。
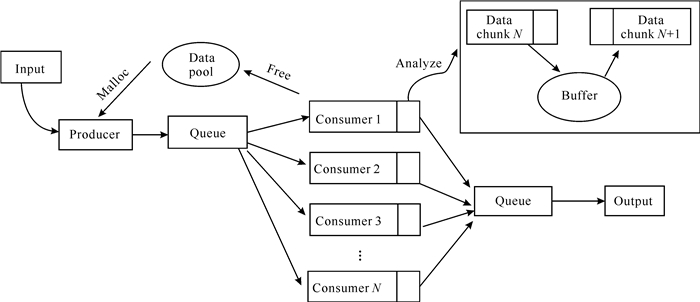
针对上述问题,本研究在经典的生产者-消费者多线程模型上改进了一个针对生物序列信息处理的I/O框架,如图 6所示。为了实现快速解析,生产者将原始文件格式化为相互独立的内存块,并使用一个具有高并发性能的数据池来有效管理这些数据块,这样可以最大程度地减少内存占用,避免频繁的内存分配,从而提高整体性能。首先,该框架把序列解析交由消费者完成,有效地缓解了生产者的负载压力,消除了生产者的性能瓶颈,提升了生产效率与程序并行度。然而,在程序执行过程中,频繁地分配与释放内存不仅可能会产生内存碎片,而且也会导致性能下降。因此,为避免频繁创建与销毁内存产生的额外开销,该框架通过数据池来管理资源。当读取序列数据时,生产者向数据池申请内存空间,如果没有空余的内存,则阻塞生产者。同理,解析序列数据完毕后,消费者释放内存空间。然后,将这些数据块放入一个共享的队列,并在访问时进行加锁,以防止由于数据竞争而引发的并发错误。最后,增加一个额外的输出队列以确保输入输出顺序一致性。数据池通过vectorA与vectorB两个动态数组来实现内存管理,其中,vectorA记录可用的数据块,vectorB记录已分配的数据块。读取数据时通过malloc函数向数据池申请空间,若有可用的数据块则加入vectorA,若无可用的数据块则进行等待直到vectorA有新的可用数据块。处理完数据后,通过free函数释放空间,同时将数据从vectorB中删除并加入vectorA中。
|
| 图 6 数据处理框架 Fig. 6 Data processing framework |
在解析序列数据时,从文件中逐个读取固定大小的数据块,并从每个数据块的末端开始向后遍历,以找到第一条完整序列的末尾位置。然后,将剩余的序列数据放入缓冲区填充下一个数据块。重复这个步骤,直至处理完所有的序列数据。这种解析策略不需要逐条读取解析序列来确定位置,仍然可以保证每个数据块末端都包含完整的一条序列,不会将一条序列切割并分布在两个不同的数据块中,这可以防止由序列不完整引起的数据处理错误。然而,如果数据块设置得过大,可能会导致线程之间的负载不平衡和较大的内存占用;如果设置得太小,则可能会产生大量的非连续内存访问,从而增加运行时间。因此,为了兼顾高效性和鲁棒性,需要设置足够大的数据块大小。序列解析算法具体实现如下。
输入:数据块指针chunkPtr、文件指针filePtr
输出:FASTQ数据块
① 处理后的数据块大小chunkSize < -0
② 每次读取的初始数据块大小size < -4M
③ function ReadChunk(chunkPtr)
④ 从缓冲区读取数据至数据块
⑤ readSize < -(size-chunkSize)
⑥ chunkPtr < -Read(filePtr, readSize)
⑦ chunkEnd < -(size-bufferSize)
⑧ 更新chunkEnd位置
⑨ chunkSize < -(size-chunkEnd)
⑩ 将剩余部分数据放入缓冲区
⑪ end function
2.2 空间定位算法及优化空间转录组测序数据的定位原理是利用25 bp的空间条形码序列来标记空间位置和测序读段,随后借助空间条形码序列进行哈希映射,将测序得到的读段定位回原来的位置。空间定位算法如下。
输入:read1(空间条形码,UMI)、read2(mRNA)两个FASTQ格式压缩文件,mask(空间条形码,position)HDF5格式文件
输出:定位成功且带有空间信息的FASTQ格式文件
① 根据mask文件,以空间条形码为key、position为value,构建哈希散列表
② 解压FASTQ压缩文件
③ for read in FASTQ文件
④ 提取read中的空间条形码序列
⑤ num < -空间条形码中含字符N的数目
⑥ if num>1 then
⑦ continue
⑧ endif
⑨ if num==1 then
⑩ 将空间条形码中N随机替换成A、T、C、G四种碱基之一
⑪ endif
⑫ else
⑬ 根据空间条形码索引哈希散列表
⑭ if找不到空间位置信息then
⑮ 将空间条形码中所有碱基随机替换成另外三种之一
⑯ 执行步骤⑬
⑰ endif
⑱ if找不到空间位置信息或有多个位置信息匹配then
⑲ continue
⑳ endif
㉑ return定位成功的唯一空间位置信息
㉒ endif
㉓ 将定位成功的空间条形码序列与空间位置信息输出FASTQ文件
㉔ end for
然而,当面对大规模转录组数据时,这种直接将空间条形码作为哈希键、坐标位置作为哈希值的键值对策略运行效率不高、定位速度较慢,其原因在于哈希容器数据结构通常采用一定倍率的扩容机制,扩容时会重新分配一个高倍容量的哈希容器,将值从较小的哈希容器移动到更大的哈希容器,然后释放原先内存。为进一步提升定位速度,本研究结合2.1节所述数据处理框架并采用分治法思想进行优化,将哈希容器进行拆分,从而减少内存峰值,而且这种拆分还有利于并发处理。同时,提取空间条形码序列前8位碱基并按照“A-00、C-01、G-10、T-11”编码方式编码,从而节省了1/4的空间开销,将空间条形码转换成16位数值n,按照n mod 8对mask文件和FASTQ文件进行分类,如图 7所示。算法的底层实现调用了C++标准库的std: : hash默认哈希散列函数FNV-1a,使用线性探测法解决冲突。内存使用情况为$O\left(\frac{\text { size }}{\text { load_factor }} \times(\text { sizeof }(\text { value_type })+1)\right)$。其中,value_type表示数据类型,sizeof(value_type)为该数据类型的大小,size是哈希容器中值的数量,装填因子$\text { load_factor }=\frac{\text { size }}{\text { bucket count }} \text { (bucket_count }$表示桶数组数量),大小在0.437 5(调整大小后)至0.875 0(调整大小前)之间变化。调整大小时额外内存使用量为$O\left({\frac{{{\rm{ size }}}}{{0.4375}} \times \left({{\rm{ sizeof }}\left({{\rm{ value }}\_{\rm{type }}} \right) + 1} \right) \times } \right.{\rm{0}}{\rm{.0625}}$),额外内存使用量对应于旧的桶数组,即扩容后新桶数组的1/2。假设哈希值均匀分布,当分成8个子映射时,0.062 5等于0.5除以8。
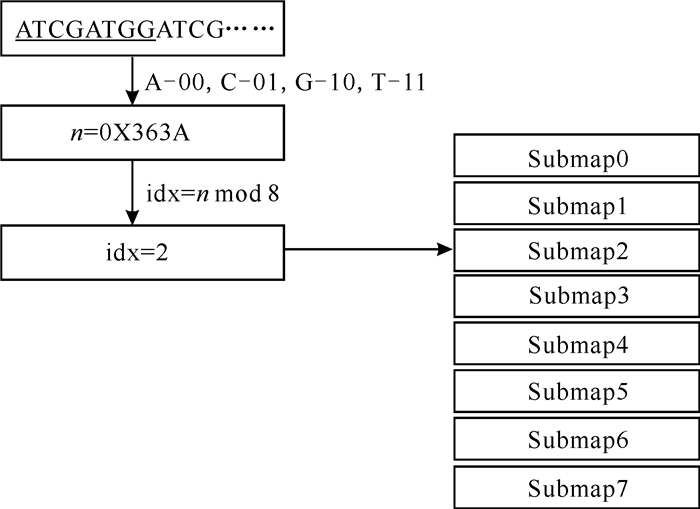
|
| 图 7 索引优化 Fig. 7 Index optimization |
3 仿真实验与结果 3.1 实验环境与数据集
为验证实验效果,本研究数据集测序样本为老鼠的脑组织SS200000135TL_D1(https://github.com/STOmics/SAW/tree/main/Test_Data)。其中,定位芯片上所有点的空间条形码序列信息都被编制成一个叫“mask”的文件,mask文件名为SS200000135TL_D1.barcodeToPos.h5,大小为4.42 GB。FASTQ格式数据集分别选用了大小为738 MB的E100026571_L01_20M_extract_read_1.fq.gz、大小为1.71 GB的E100026571_L01_20M_extract_read_2.fq.gz,均包含了21 497 957条序列。实验在ubuntu22.04.2LTS 64位操作系统环境下运行,硬件平台为Intel Ice Lake (2.7 GHz/3.3 GHz),64核处理器,内存为470 GB。
3.2 测试结果由于本研究框架主要针对FASTQ格式压缩数据,因此其处理效率在一定程度上受解压速度的影响,尤其是在处理大规模生物序列压缩文件时,解压过程可能会非常耗时。基于上述问题,本研究首先尝试在框架中引入zlib软件库来支持FASTQ格式压缩文件解压,zlib的优势在于其能够以较低的性能代价获得较高的解压速度,但对于大规模序列而言其效率表现依然不够优秀。为进一步提高大规模序列压缩文件的处理效率,在框架中引入高性能解压缩库isa-l替代zlib,实验测试两者在框架中的性能差异,结果如图 8所示。zlib具备更好的压缩率,但代价是性能较低,而isa-l则能够以牺牲部分压缩率的代价获取更高的处理性能。
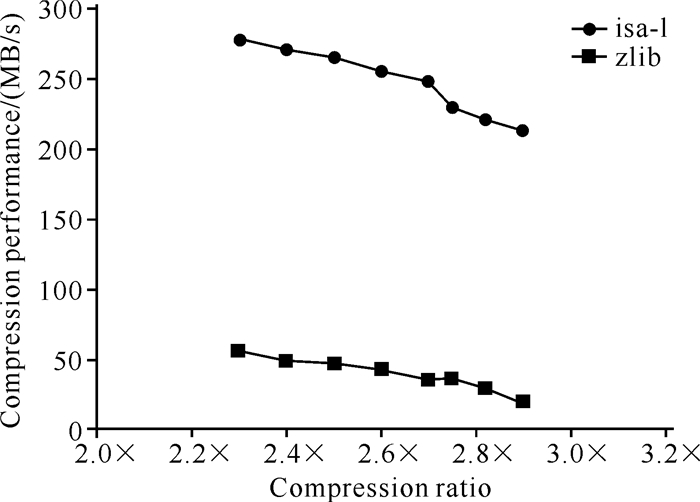
|
| "×" means "multiple". 图 8 算法在解压缩库isa-l与软件库zlib下的性能比对 Fig. 8 Performance comparison of algorithms under decompression library isa-l and software library zlib |
此外,为了实现并行哈希定位,引入高性能开源库Parallel-hashmap重写映射表,在多线程环境中实现更高的性能。它通过将数据划分为多个桶,并在每个桶上使用单独的线程进行操作,从而允许多个线程同时访问和修改数据,在减少了锁竞争的同时提高并发性能,而且还提升了定位速度,图 9(a)和(b)分别给出了单线程和多线程下使用Parallel-hashmap带来的速度提升测试。
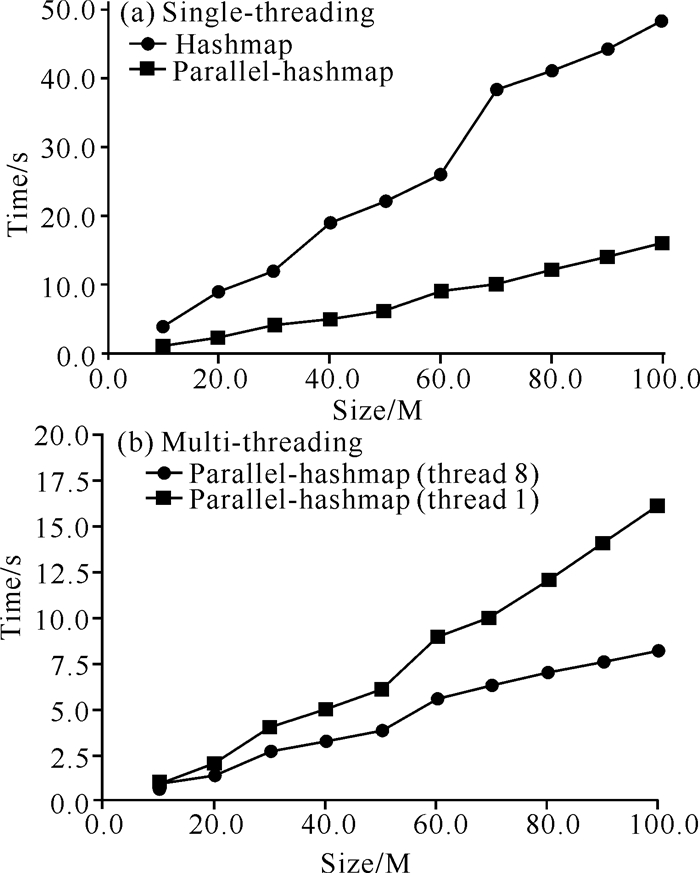
|
| 图 9 性能测试 Fig. 9 Performance test |
最后,对本研究所提的空间定位算法进行速度测试,结果表明本研究方案能够通过空间条形码序列成功定位,在未经过数据处理的情况下,算法运行时间为403 s,经过本研究所提方案优化后定位算法耗时由403 s下降到173 s,速度提升1.33倍。
4 结论细胞之间的关系以及它们在组织样本中的相对位置对理解疾病病理学至关重要,如何实现空间转录组快速定位对肿瘤诊断研究具有重大意义。实验表明,本研究方案显著提升了空间转录组的定位速度。由于测序技术限制,序列数据并非百分百准确,目前的容错策略是将空间条形码序列碱基进行替换,这种容错策略时间复杂度较高,且内存开销大。在本研究基础上,未来的工作可以从以下3点入手:①考虑改进目前的容错方式以降低时间复杂度;②进一步对软件进行优化,提升哈希表查找效率,以及压缩哈希表减少存储空间;③借鉴或探索设计其他索引数据结构,实现大规模转录组空间快速定位及性能优化。
| [1] |
张思影, 陈峰. 肿瘤空间异质性影像学定量评价进展[J]. 中华放射肿瘤学杂志, 2017, 26(12): 1451-1456. |
| [2] |
肖宇彬, 张子旭, 王玉珠, 等. 时空转录组研究进展[J]. 植物学报, 2023, 58(2): 214-232. |
| [3] |
KRUSE F, JUNKER J P, VAN OUDENAARDEN A, et al. Tomo-seq: a method to obtain genome-wide expression data with spatial resolution[J]. Methods in Cell Biology, 2016, 135: 299-307. |
| [4] |
CHEN J, SUO S, TAM P P, et al. Spatial transcriptomic analysis of cryosectioned tissue samples with Geo-seq[J]. Nature Protocols, 2017, 12(3): 566-580. DOI:10.1038/nprot.2017.003 |
| [5] |
NICHTERWITZ S, BENITEZ J A, HOOGSTRAATEN R, et al. LCM-seq: a method for spatial transcriptomic profiling using laser capture microdissection coupled with PolyA-based RNA Sequencing[J]. Methods in Molecular Biology, 2018, 1649: 95-110. |
| [6] |
CHEN J X, MCSWIGGEN D, VNAL E. Single molecule fluorescence in situ hybridization (smFISH) analysis in budding yeast vegetative growth and meiosis[J]. Journal of Visualized Experiments, 2018(135): e57774. |
| [7] |
ENG C L, LAWSON M, ZHU Q, et al. Transcriptome-scale super-resolved imaging in tissues by RNA seqFISH[J]. Nature, 2019, 568(7751): 235-239. DOI:10.1038/s41586-019-1049-y |
| [8] |
XIA C L, FAN J, EMANUEL G, et al. Spatial transcriptome profiling by MERFISH reveals subcellular RNA compartmentalization and cell cycle-dependent gene expression[J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(39): 19490-19499. |
| [9] |
GYLLBORG D, LANGSETH C M, QIAN X Y, et al. Hybridization-based in situ sequencing (HybISS) for spatially resolved transcriptomics in human and mouse brain tissue[J]. Nucleic Acids Research, 2020, 48(19): e112. DOI:10.1093/nar/gkaa792 |
| [10] |
LEE J H, DAUGHARTHY E R, SCHEIMAN J, et al. Fluorescent in situ sequencing (FISSEQ) of RNA for gene expression profiling in intact cells and tissues[J]. Nature Protocols, 2015, 10(3): 442-458. DOI:10.1038/nprot.2014.191 |
| [11] |
ALON S, GOODWIN D R, SINHA A, et al. Expansion sequencing: spatially precise in situ transcriptomics in intact biological systems[J]. Science, 2021, 371(6528): eaax2656. DOI:10.1126/science.aax2656 |
| [12] |
FVRTH D, HATINI V, LEE J H. In situ transcrip-tome accessibility sequencing (INSTA-seq) [Z/OL]. (2019-08-06)[2023-11-10]. https://doi.org/10.1101/722819.
|
| [13] |
STÅHL P L, SALMÉN F, VICKOVIC S, et al. Visualization and analysis of gene expression in tissue sections by spatial transcriptomics[J]. Science, 2016, 353(6294): 78-82. DOI:10.1126/science.aaf2403 |
| [14] |
RODRIQUES S G, STICKELS R R, GOEVA A, et al. Slide-seq: a scalable technology for measuring genome-wide expression at high spatial resolution[J]. Science, 2019, 363(6434): 1463-1467. DOI:10.1126/science.aaw1219 |
| [15] |
LIU Y, YANG M, DENG Y, et al. High-spatial-resolution multi-omics sequencing via deterministic barcoding in tissue[J]. Cell, 2020, 183(6): 1665-1681.e18. DOI:10.1016/j.cell.2020.10.026 |
| [16] |
CHO C S, XI J Y, SI Y P, et al. Microscopic examination of spatial transcriptome using Seq-Scope[J]. Cell, 2021, 184(13): 3559-3572.e22. DOI:10.1016/j.cell.2021.05.010 |
| [17] |
CHEN A, LIAO S, CHENG M, et al. Spatiotemporal transcriptomic atlas of mouse organogenesis using DNA nanoball-patterned arrays[J]. Cell, 2022, 185(10): 1777-1792.e21. DOI:10.1016/j.cell.2022.04.003 |
| [18] |
程昊宇. 面向大规模测序数据集的序列比对算法研究[D]. 合肥: 中国科学技术大学, 2019.
|
| [19] |
BURROWS M, WHEELER D J. A block-sorting lossless data compression algorithm [R/OL]. (1994-05-10)[2023-11-21]. https://www.cs.jhu.edu/~langmea/resources/burrows_wheeler.pdf.
|
| [20] |
LI H. Minimap2: pairwise alignment for nucleotide sequences[J]. Bioinformatics, 2018, 34(18): 3094-3100. DOI:10.1093/bioinformatics/bty191 |
| [21] |
ALSER M, ROTMAN J, DESHPANDE D, et al. Technology dictates algorithms: recent developments in read alignment[J]. Genome Biology, 2021, 22(1): 249. DOI:10.1186/s13059-021-02443-7 |



