2. 玉林师范学院, 广西高校复杂系统优化与大数据处理重点实验室, 广西玉林 537000;
3. 玉林师范学院计算机科学与工程学院, 广西玉林 537000;
4. 玉林师范学院数学与统计学院, 广西玉林 537000
2. Guangxi Colleges and Universities Key Laboratory of Complex System Optimization and Big Data Processing, Yulin Normal University, Yulin, Guangxi, 537000, China;
3. School of Computer Science and Engineering, Yulin Normal University, Yulin, Guangxi, 537000, China;
4. School of Mathematics and Statistics, Yulin Normal University, Yulin, Guangxi, 537000, China
由于单细胞RNA测序(Single-cell RNA sequencing, scRNA-seq)数据有着高维性、噪声分布不均匀、稀疏性、低读取深度以及缺乏标注信息等特点,高质量的数据降维在下游分析中显得尤为重要[1]。受到技术限制和生物学变异的影响,scRNA-seq数据通常呈现出零膨胀的特征,即大部分基因表达值为0,有时表达值为0甚至占所有基因的90%以上。这些零值究竟是来源于生物变异还是技术缺陷成为了一个重要的不确定因素。这些零值并非随机分布,而是受到技术噪声、细胞类型和状态等因素的影响,呈现出不均匀性,这可能会影响数据的统计分析结果和后续的生物学解释。过去的研究表明,非负矩阵分解(Non-negative Matrix Factorization, NMF)能有效地提取基因表达的潜在特征,但在处理含噪声和稀疏数据方面仍有不足,特别是在处理稀疏度不同的数据时表现不佳[2]。为解决这些问题,需要深入研究零膨胀数据的分布,并结合NMF降维方法来处理这些数据,从而提高数据的准确性和可靠性[3, 4]。
尽管NMF能够有效地提取高维数据中的潜在低维特征,尤其在应对scRNA-seq数据中的零膨胀现象时显示出优势,但其在处理稀疏和噪声数据时,尤其在面对不同稀疏度的数据时,性能显著下降。为克服这一问题,研究人员提出了多种改进的NMF方法。鲁棒非负矩阵分解(Robust Non-negative Matrix Factorization, rNMF)因其更强的鲁棒性,能够在噪声或异常值存在的情况下,更好地提取数据的结构信息[5];rNMF通过优化目标函数,结合了平方损失函数和稀疏惩罚项,以确保分解后的矩阵具有稀疏性和鲁棒性。基于图模型非负矩阵分解(Graph Regularized Non-negative Matrix Factorization, GNMF)方法利用细胞之间的关系信息进行数据分解[6];GNMF通过图正则化保持细胞之间的相似性,并借助NMF提取潜在特征,从而在保留数据结构的同时,有效识别细胞亚型。鲁棒图正则化非负矩阵分解(Robust Graph Regularized Non-negative Matrix Factorization,rGNMF)相较于GNMF,在优化目标函数时还考虑了降低对异常值的敏感性,对数据中的噪声和异常值具有更强的抵抗能力[7]。概率非负矩阵分解(Probabilistic Non-negative Matrix Factorization,pNMF)通过将NMF与概率图模型相结合,建模数据的生成过程,并利用EM算法等方法进行参数估计。pNMF能够更好地处理数据中的不确定性和噪声,从而提高插补的准确性和稳健性[8]。
为了处理稀疏的单细胞数据,基于NMF的质谱代谢组学数据缺失值插补方法[9]利用NMF模型进行低秩近似,并将零值作为优化目标之一来优化NMF模型参数,该缺失值插补方法在处理大规模数据时效果显著,但在处理高度不均匀的数据分布时效果不够理想。基于低秩近似的无标记质谱肽段插补(Mass Spectrometry Imputation, MSImpute)方法利用NMF的低秩逼近和样本相似性来处理质谱数据,充分考虑了数据稀疏性和噪声影响,适用于大规模质谱数据,但其在运算速度和结果稳定性方面仍存在不足[10]。此外,将NMF与深度学习技术结合,利用深度学习网络的非线性特性可以更好地拟合数据的复杂结构,从而解决大规模数据集的内存溢出问题, 提高聚类效果,但是存在鲁棒性欠缺的问题[11]。一些半监督学习方法利用已知信息指导数据降维、整合标记和未标记数据,从而提高模型性能和鲁棒性[12]。
应用基于NMF的分解方法处理单细胞数据时,必须考虑数据的分布特征以确保方法的有效性和适用性。许多研究假设基因表达量服从伽玛-泊松(Gamma-Poisson)分布,且已有研究表明,这一假设能够较好地拟合scRNA-seq数据,解释其分布特征并提供准确的统计推断。这一理论基础来源于scRNA-seq数据的两个主要特征——技术噪声和生物学变异[13, 14]。技术噪声在低表达水平下更为显著,此时基因表达量的变异主要由测序技术的随机误差所导致,泊松分布适合描述这种情况,并且泊松分布方差与均值相等的特性与低表达基因的噪声特性相吻合。在高表达水平下,生物学变异成为主要变异来源,技术噪声的影响较小并且基因表达量更接近连续值,此时用伽玛分布描述更为合适,伽玛分布的灵活性能够帮助捕捉高表达水平下的广泛变异。因此,结合伽玛和泊松分布的伽玛-泊松分布模型能够综合描述低、高表达水平下的基因表达数据,提供更准确的统计推断和数据特征描述[15, 16]。
综上所述,这些方法各有优点和局限性,应根据具体的数据特征来确定适合的方法。深度学习增强的NMF适用于处理复杂的数据模式和关系,半监督学习的NMF适用于利用有限的标记数据进行降维,而基于概率图模型的NMF适用于处理数据的不确定性和噪声。虽然传统NMF方法在处理scRNA-seq数据时存在局限性,但通过引入正则化,结合图模型,与深度学习和概率图模型的结合,能够有效应对单细胞数据的稀疏性、噪声和计算复杂性等问题。为了克服传统NMF方法在处理高稀疏数据时的不足,并提升对单细胞数据稀疏性、噪声和计算复杂性的处理能力,本文提出一种改进的单细胞非负矩阵分解算法GPNMF。
1 相关工作 1.1 NMF分解方法在scRNA-seq数据中,存在零膨胀(Zero-inflation)现象,即大量基因表达值为零。因此,需要对传统的NMF公式进行改进,才能更好地揭示scRNA-seq数据的结构和动态表达模式。具体来说,对NMF的原始公式使用Frobenius范数来度量数据的重构误差,并假设数据噪声来自高斯分布,如公式(1)所示:
| $ \min\limits _{\mathbf{W}, \mathbf{H}}\|\mathbf{X}-\mathbf{W} \mathbf{H}\|_{\mathbf{F}}^2, \text { s. t. }, \mathbf{W} \geqslant 0 \quad \mathbf{H} \geqslant 0, $ | (1) |
其中,X是输入的数据矩阵,W和H是分解得到的非负矩阵,$\|\mathbf{X}-\mathbf{W H}\|_{\mathbf{F}}^{2}$表示Frobenius范数。
scRNA-seq数据中大量的零值不一定是噪声,也可能是由于实验技术和生物学因素导致的。因此,需要调整NMF公式以更好地适应这种零膨胀的数据特点。一种改写方式是引入一个二值矩阵 M,用于掩盖数据中的零值[17]。该二值矩阵的元素如公式(2)所示:
| $ \mathbf{M}_{i j}=\left\{\begin{array}{c} 1, x_{i j}>0 \\ 0, \text { otherwise } \end{array} 。\right. $ | (2) |
通过该二值矩阵 M,可以修改NMF的目标函数,其能够更好地处理scRNA-seq数据中的零值。改写后的目标函数如公式(3)所示:
| $ \min\limits _{\mathbf{W}, \mathbf{H}}\|\mathbf{M} \odot \mathbf{X}-\mathbf{M} \odot \mathbf{W} \mathbf{H}\|_{\mathbf{F}}^{2}, \text { s. t. }, \mathbf{W} \geqslant 0 \quad \mathbf{H} \geqslant 0 , $ | (3) |
其中,$\odot$表示元素级别的乘法运算。这样就可以利用NMF的模型结构和零膨胀特性,充分描述scRNA-seq数据中的零值,从而提高数据的完整性和后续分析的可信度。
基于二值矩阵的NMF方法通过掩盖scRNA-seq数据中的零值,能够更好地应对零膨胀问题,从而精确捕捉数据的潜在结构。然而,选择合适的掩盖矩阵 M可能存在困难,尤其在高度稀疏的数据中,零值标记可能不准确,影响模型效果。与此相比,传统的NMF方法基于Frobenius范数,简单且适用于噪声较低的情况,但它未能有效处理scRNA-seq数据中的零膨胀现象,可能导致数据建模不准确。因此,基于二值矩阵的方法更适合零膨胀数据,而传统方法则在数据噪声较少时表现较好。
1.2 GNMF分解方法在scRNA-seq数据分析中,捕捉细胞之间的局部几何结构和全局拓扑关系是揭示细胞异质性和谱系的重要任务。GNMF[6]通过引入图正则化项改进标准NMF,从而能够更好地保持数据的局部邻域结构。GNMF假设数据点之间的相似性可用由邻接矩阵 S定义的图表示。GNMF的目标函数如公式(4)所示:
| $ \min\limits _{\mathbf{w}, \mathbf{H}}\|\mathbf{X}-\mathbf{W} \mathbf{H}\|_{\mathbf{F}}^{2}+\alpha \sum\limits_{i, j} \mathbf{S}_{i, j}\left\|\mathbf{W}_{i}-\mathbf{W}_{j}\right\|^{2} , $ | (4) |
其中,X是scRNA-seq数据矩阵,S是反映数据点间相似性的邻接矩阵,α是正则化参数。正则项确保了相似的数据点在低维表示中保持接近,从而更好地反映细胞之间的关系。在具体应用中,邻接矩阵 S可以通过k近邻图或高斯相似性函数构建,如公式(5)所示:
| $ \mathbf{S}_{i j}=\exp \left(-\frac{\left\|X_{i}-X_{j}\right\|^{2}}{2 \sigma^{2}}\right) , $ | (5) |
式中,Xi和Xj为不同的数据点,σ为标准差。
GNMF在单细胞分析中的应用包括细胞类型分类、细胞谱系推断和动态变化分析等,其优势在于能够在降维过程中充分利用数据的局部结构信息,使得降维后的表示更加与生物学相关。
1.3 rGNMF分解方法虽然GNMF方法能够有效地捕捉scRNA-seq数据的局部几何结构和全局拓扑关系,但其对数据中的噪声和异常值的处理能力有限。为了增强GNMF的鲁棒性,rGNMF方法被提出[7]。rGNMF通过引入处理噪声和异常值的机制,增强了模型的鲁棒性。
rGNMF的目标函数在标准GNMF的基础上加入了对噪声和异常值的处理项,其形式如公式(6)所示:
| $ \begin{aligned} & \min _{\mathbf{W}, \mathbf{H}, \mathbf{E}}\|\mathbf{X}-\mathbf{W H}-\mathbf{E}\|_{\mathbf{F}}^2+\alpha \sum\limits_{i, j} \mathbf{S}_{i j}\left\|\mathbf{W}_i-\mathbf{W}_j\right\|^2+ \\ & \beta\|\mathbf{E}\|_{2, 1}, \end{aligned} $ | (6) |
其中,E表示噪声和异常值的矩阵,α和β是正则化参数。目标函数中的第3项$\|\mathbf{E}\|_{2, 1}$是E的L2, 1范数,用于增强对异常值的鲁棒性。该范数定义如公式(7)所示:
| $ \|\mathbf{E}\|_{2, 1}=\sum\limits_{i}^{n}\left\|\mathbf{E}_{i, :}\right\|_{2} , $ | (7) |
其中,$\left\|\mathbf{E}_{i, : }\right\|_{2}$ 是矩阵$\mathbf{E}$ 第$i$ 行的$L_{2, 1}$ 范数,通过引入矩阵$\mathbf{E}$ 来显式建模数据中的噪声和异常值,并采用$L_{2, 1}$ 范数来减少其影响,从而提高降维结果的鲁棒性。具体的优化过程通常采用交替优化策略 [7],即交替优化$\mathbf{W}$,$\mathbf{H}$ 和$\mathbf{E}$ 以逐步逼近目标函数的最优值。
2 方法 2.1 基于伽玛-泊松分布和图正则化的NMF方法(GPNMF)本文方法的目标是通过最大化观测数据的似然来学习模型的参数。假设一个观测数据矩阵 X,其维度为(ns, nf),其中ns是样本数,即单细胞数据中的细胞数;nf是特征数,即单细胞数据中的基因数。使用矩阵分解来联合构建细胞和基因的表示,在低维空间中分别是维度为(ns, nc)的矩阵 W和维度为(nc, nf)的矩阵 H,其中nc是潜在空间的维度。
假设观测数据X服从伽玛-泊松分布,并且根据NMF模型,将 WH视为模型对数据的重构Xpred。伽玛-泊松分布是对计数数据建模的一种常见方法,通过将泊松分布的参数化结合伽玛分布的先验信息来表示,从而可以更灵活地处理数据中的过度离散和稀疏现象。观测值Xi的概率密度函数如公式(8)所示:
| $ f\left(X_{i} ; \alpha, \beta\right)=\frac{\beta^{\alpha}}{\Gamma(\alpha)} \cdot \frac{X_{i}^{\alpha-1}}{(1+\beta)^{\alpha+X_{i}}} \cdot \frac{1}{X_{i}!} , $ | (8) |
其中α和β分别是伽玛-泊松分布的形状参数和尺度参数,$\Gamma(\cdot)$是Gamma函数,Xi表示第i个观测值。接下来,考虑该分布的负对数似然函数$-\log \left[f\left(X_{i}\right.\right.$;$\alpha, \beta)]$。简化过程如公式(9)所示:
| $ \begin{aligned} & \quad-\log \left[f\left(X_i ; \alpha, \beta\right)\right]=-\log \left[\frac{\beta^\alpha}{\Gamma(\alpha)} \cdot\right. \\ & \left.\frac{X_i^{\alpha-1}}{(1+\beta)^{\alpha+X_i}} \cdot \frac{1}{X_{i}!}\right]=-\log \left[\frac{\beta^\alpha}{\Gamma(\alpha)}\right]- \\ & \log \left[\frac{X_i^{\alpha-1}}{(1+\beta)^{a+X_i}}\right]-\log \left(\frac{1}{X_{i}!}\right)=\log \left[\frac{\Gamma(\alpha)}{\beta^\alpha}\right]- \\ & \log \left[\frac{X_i^{\alpha-1}}{(1+\beta)^{\alpha+X_i}}\right]+\log \left[X_{i}!\right)=\log \left(\frac{\Gamma(\alpha)}{\beta^\alpha}\right]-[(\alpha- \\ & \left.1) \log \left(X_i\right)-\left(\alpha+X_i\right) \log (1+\beta)\right]+\log \left(X_{i}!\right)= \\ & -\log \left[\frac{\Gamma(\alpha)}{\beta^\alpha}\right]+(\alpha-1) \log \left(X_i\right)-(\alpha+ \\ & \left.X_i\right) \log (1+\beta)-\log \left(X_{i}!\right) 。\end{aligned} $ | (9) |
在伽玛-泊松分.布参数估计中,形状参数α和尺度参数β对概率密度函数的形状和尺度有着重要的影响。这些参数通常通过最大似然估计(Maximum Likelihood Estimation,MLE)或贝叶斯估计(Bayesian Estimation,BE)等方法来单独估计。为了简化计算过程,本文提出一种方法,将这些参数替换为模型的预测值$X_{\text {pred }_{i}}$,即$\alpha=X_{\text {pred }_{i}}, \beta=X_{\text {pred }_{i}}$。虽然直接用预测值替代参数并不完全符合严格的统计推断,但在实际应用中,将预测值直接用于形状参数和尺度参数可以简化模型的计算复杂度。这种方法避免了在每次迭代中重新估计多个参数,从而加快了模型的训练过程。在统计建模和机器学习中,损失函数通常需要衡量模型预测值与实际观测值之间的差异。尽管简化了参数估计过程,NMF等模型通常在损失函数中会最小化预测值$X_{\text {pred }_{i}}$ 和实际观测值之间的差异,这使得$X_{\text {pred }_{i}}$ 在一定程度上反映了数据的分布特征,包括形状和尺度,此时负对数似然函数如公式(10)所示:
| $ \begin{aligned} & \quad-\log \left[f\left(X_i ; X_{\text {pred }_i}\right)\right]=-\log \left[\frac{X_{\text {pred }_i}^{X_{\text {pred }_i}}}{\Gamma\left(X_{\text {pred }_i}\right)}\right]+ \\ & \left(X_{\text {pred }_i}-1\right) \log \left(X_i\right)-\left(X_{\text {pred }_i}+X_i\right) \log \left(1+X_{\text {pred }_i}\right)- \\ & \log \left(X_{i}!\right)=-X_{\text {pred }_i} \log \left(X_{\text {pred }_i}\right)+\log \left[\Gamma\left(X_{\text {pred }_i}\right)\right]+ \\ & \left(X_{\text {pred }_i}-1\right) \log \left(X_i\right)-\left(X_{\text {pred }_i}+X_i\right) \log \left(1+X_{\text {pred }_i}\right)- \\ & \log \left(X_{i}!\right) 。\end{aligned} $ | (10) |
负对数似然函数提供了一种基于概率模型的误差度量,它可以直接用于构建损失函数。然而,简单的负对数似然函数可能不足以捕捉所有模型误差的特点,因此引入了NMF模型预测值$X_{\text {pred }}^{i}$与观测值$X_{i}$之间的误差$\Delta_{i}=X_{i}-X_{\text {pred }_{i}}$,并将其与负对数似然函数中的对数项$\left(X_{\text {pred }_{i}}+X_{i}\right) \log \left(1+X_{\text {pred }_{i}}\right)$结合起来。这种结合可以确保损失函数既考虑了基于概率模型的误差(对数项),又考虑了实际观测误差(误差项)。
对数项$\left(X_{\text {pred }_{i}}+X_{i}\right) \log \left(1+X_{\text {pred }_{i}}\right)$ 是公式(10)负对数似然函数中的一部分,可以近似处理为
| $ X_{\text {pred }_{i}} \log \left(\frac{X_{\text {pred }_{i}}}{X_{i}+\varepsilon}\right) , $ | (11) |
其中ε是一个极小的正值,以防止 WH中出现零元素,从而避免在计算对数时出现无穷大值。将公式(11)和$\Delta_{i}$ 结合起来构建损失函数,函数的平方和用于度量误差的影响。损失函数如公式(12)所示:
| $ {\rm{ Loss}}{{\rm{ }}_{{\rm{temp }}}} = {\rm{ }}\sum\limits_{i = 1}^n {{{\left[ {\left( {{X_i} - {X_{{\rm{pred}}{{\rm{ }}_i}}}} \right) + {X_{{\rm{pred}}{{\rm{ }}_i}}}\log \left( {\frac{{{X_{{\rm{pred}}{{\rm{ }}_i}}}}}{{{X_i} + \varepsilon }}} \right)} \right]}^2}} {\rm{ }} 。$ | (12) |
为了让模型在训练过程中学习到样本之间的拓扑结构信息,提高模型的鲁棒性和泛化能力,通过在公式(12)的损失函数中引入图正则化项,模型可以更好地保持相似样本之间的连续性,从而减少过拟合的风险并提高性能,图正则化项在NMF中用于约束分解结果,以保留数据的局部和全局结构。计算邻接矩阵是图正则化项的基础,因为它反映了数据点之间的相似性或连接程度。因此,使用高斯核函数和k个最近邻居构建邻接矩阵 A,可以得到邻接矩阵A的计算过程如公式(13)所示:
| $ \mathbf{A}_{i j}=\left\{\begin{array}{c} \exp \left(-\frac{\left\|x_i-x_j\right\|^2}{2 \sigma^2}\right), x_j \in N\left(x_i\right) \\ 0, \text { otherwise } \end{array}\right. , $ | (13) |
其中,$\left\|x_{i}-x_{j}\right\|^{2}$ 表示细胞$i$ 和细胞$j$ 之间特征向量的欧式距离的平方,$N\left(x_{i}\right)$ 表示$x_{i}$ 的$k$ 个最近邻居,假设$\mathbf{D}$ 是度矩阵,拉普拉斯算矩阵$\mathbf{L}=\mathbf{D}-\mathbf{A}$,$\operatorname{Tr}(\cdot)$ 表示矩阵的迹,则图正则化项如公式(14)所示:
| $ \begin{gathered} \mathbf{R}=\frac{1}{2} \sum\limits_{i, j} \mathbf{A}_{i j}\left\|h_{i}-h_{j}\right\|^{2}=\sum\limits_{i} \mathbf{D}_{i i} h_{i}^{\mathrm{T}} h_{i}- \\ \sum\limits_{i j} \mathbf{A}_{i j} h_{i}^{\mathrm{T}} h_{j}=\operatorname{Tr}\left(\mathbf{H} \mathbf{D} \mathbf{H}^{\mathrm{T}}\right)-\operatorname{Tr}\left(\mathbf{H} \mathbf{A} \mathbf{H}^{\mathrm{T}}\right)=\operatorname{Tr}\left(\mathbf{H L} \mathbf{H}^{\mathrm{T}}\right), \end{gathered} $ | (14) |
其中,$\mathbf{H}$ 是NMF分解中的基矩阵。具体来说,NMF将非负矩阵$\mathbf{X}$ 分解为两个非负矩阵$\mathbf{W}$ 和$\mathbf{H}$,在图正则化的上下文中,$\mathbf{H}$ 矩阵的每一列$h_{i}$ 代表数据点$i$在新的低维空间中的表示。通过引入图正则化项$\operatorname{Tr}\left(\mathbf{H L H}^{\mathrm{T}}\right)$,模型能够保持数据点在原始空间中的邻近关系,确保相似的数据点在低维表示中也
| $ \begin{gathered} \text { Loss }_{\text {final }}= \\ \sum\limits_{i=1}^n\left[\left(X_i-X_{\text {pred }_i}\right)+X_{\text {pred }_i} \log \left(\frac{X_{\text {pred }_i}}{X_i+\varepsilon}\right)\right]^2+\lambda R 。\end{gathered} $ | (15) |
鲁棒性图正则化方法采用了与rGNMF分解方法相同的更新规则,更新规则 Q、W和H分别如公式(16)-(18)所示:
| $ \begin{aligned} & \mathbf{Q}_{i i}= \\ & \frac{1}{\left\|X_i-(\mathbf{W H})_i+(\mathbf{W H})_i \cdot \log \left[\frac{X_i}{(\mathbf{W H})_{\mathrm{i}}}\right]\right\|_2}, \end{aligned} $ | (16) |
| $ \mathbf{W}_{i j} \leftarrow \mathbf{W}_{i j} \frac{\left(\mathbf{X Q H}^{\mathrm{T}}\right)_{i j}}{\left(\mathbf{W H Q H}^{\mathrm{T}}\right)_{i j}+\varepsilon}, $ | (17) |
| $ \mathbf{H}_{i j} \leftarrow \mathbf{H}_{i j} \frac{\left[\left(\mathbf{W}^{\mathrm{T}} \mathbf{X} \mathbf{Q}+2^\lambda \mathbf{H A}\right)_{i j}\right]}{\left[\left(\mathbf{W}^{\mathrm{T}} \mathbf{W} \mathbf{H Q}+2^\lambda \mathbf{H D}\right)_{i j}\right]+\varepsilon}, $ | (18) |
其中,W 是权重矩阵;$\mathbf{X}$ 是原始数据矩阵,表示节点之间的关系矩阵;$\mathbf{H}$ 是待学习的矩阵,表示图数据分析中节点的特征表示;在更新$\mathbf{W}$ 和$\mathbf{H}$ 时,$\varepsilon$ 是一个极小的正值,用于避免除零错误。在更新$\mathbf{Q}$ 时,使用矩阵的范数来计算$\mathbf{Q}$ 的对角元素。图正则化项$R=$ $\operatorname{Tr}\left(\mathbf{H L H}^{\mathrm{T}}\right)$ 用于计算图拉普拉斯矩阵$\mathbf{L}=\mathbf{D}-\mathbf{A}$ 及其相关的矩阵,$\lambda$ 是正则化权重参数,$\operatorname{Tr}(\cdot)$ 表示矩阵的迹,即矩阵对角线上元素的总和。
2.2 算法流程算法通过将数据建模为概率分布,结合非负矩阵分解和图正则化,能够更好地挖掘数据的结构信息,对处理具有拓扑结构的数据集具有很好的效果,具体算法流程如算法1所示。
算法1 GPNMF算法
输入:原始数据矩阵 X,初始权重矩阵W,初始特征矩阵H,损失函数L,图的邻接矩阵 A,图的度矩阵D
输出:更新后的权重矩阵 W和特征矩阵H
① 设置损失函数L,图的邻接矩阵 A,图的度矩阵D
② 对 X进行对数变换,然后对每一行进行归一化处理
③ 奇异值分解的结果来初始化矩阵 W和H
④ 初始化迭代次数iteration为1,最大迭代次数max_iter为100,损失函数的相对误差范围tol为1e-5
⑤ 初始化前次迭代损失prev_loss其初始值根据损失函数的数值范围设定,以确保计算稳定性
⑥ while (iteration < max_iter and (prev_loss-curr_loss) / prev_loss > tol),执行以下步骤:
a.将前一次迭代的损失prev_loss更新为当前损失curr_loss
b.利用公式(16)更新矩阵 Q,进行归一化
c.利用公式(17)通过更新矩阵 W
d.利用公式(18)通过更新矩阵 H
e.利用公式(15)计算当前损失curr_loss,以逼近原始数据矩阵 X
f.迭代次数iteration加1
若迭代次数达到最大迭代次数max_iter,则循环结束
3 实验与结果分析 3.1 数据集为了验证算法的准确性,选取20个数据集进行实验分析,其中包括human_kidney[18]、GSE75748[19]以及Young[20]等。这些数据集来源于不同的实验和研究,涵盖了多个生物体系和组织类型,具有丰富的细胞谱系信息,具体信息如表 1所示。
| 数据集 Dataset | 平台 Platform |
类别数 Number of classes |
细胞数量 Number of cells |
基因数量 Number of genes |
稀疏度/% Sparsity/% |
| 10X_PBMC | 10X | 8 | 4 271 | 16 653 | 92.23 |
| Adam | Drop-seq | 8 | 3 660 | 23 797 | 92.33 |
| CITE_CBMC | 10X | 7 | 2 881 | 21 143 | 85.92 |
| HumanLiver | 10X | 7 | 2 881 | 21 143 | 85.92 |
| Macosko mouse | Drop-seq | 39 | 14 653 | 11 422 | 85.92 |
| Muraro | CEL-seq2 | 9 | 2 122 | 19 046 | 73.02 |
| Bladder | 10X | 4 | 2 500 | 23 341 | 86.94 |
| 10X_Muscle | 10X | 6 | 3 909 | 23 341 | 93.57 |
| Spleen | 10X | 5 | 9 552 | 23 341 | 94.34 |
| Diaphragm | Smart-seq2 | 5 | 870 | 23 341 | 91.35 |
| QS_Muscle | Smart-seq2 | 6 | 1 090 | 23 341 | 89.47 |
| QS_Lung | Smart-seq2 | 11 | 1 676 | 23 341 | 89.08 |
| QS_Trachea | Smart-seq2 | 4 | 1 350 | 23 341 | 85.48 |
| Romanov | Fluidigm C1 | 7 | 2 881 | 21 143 | 85.92 |
| Shekhar mouse | 10X | 7 | 2 881 | 21 143 | 85.92 |
| Young | 10X | 11 | 5 685 | 33 658 | 94.70 |
| human_kidney | 10X | 11 | 5 685 | 25 215 | 85.92 |
| mouse_ES | Droplet | 4 | 2 717 | 20 670 | 65.76 |
| worm_neuron | sci-RNA-seq | 10 | 4 186 | 11 955 | 85.92 |
| GSE75748 | Fluidigm C1 | 6 | 758 | 19 189 | 54.68 |
3.2 对比实验
本文对这些数据集进行NMF方法的基准测试,对降维的特征进行k-Means聚类,计算的指标包括调整兰德指数(Adjusted Rand Index, ARI)、归一化互信息(Normalized Mutual Information, NMI)、纯度(Purity)和准确率(Accuracy, ACC),这些指标能够客观地评价算法的准确性和性能。将本文方法GPNMF与其他常用的NMF方法(如NMF、rNMF、GNMF、rGNMF和GP_rGNME)进行比较。为了公平比较,5种方法均采用奇异值分解(SVD)的结果来初始化权重矩阵 W和特征矩阵H,不对比监督或半监督的NMF方法。低维空间nc设置为基因数的平方根取整,对所有带正则项的NMF方法,一律设置正则化项权重λ=1,GNMF的图正则化项采用k近邻方法构造邻接矩阵,k近邻值设置为8。
为了评估NMF方法在20个数据集上的性能,对数据进行log(1+x)和归一化处理,每种方法的ARI和NMI值见表 2。GPNMF在多个数据集上表现突出,特别是在10X_PBMC(0.55)、Adam(0.49)、10X_Muscle(0.96)、Spleen(0.86)等数据集中显示了最高的ARI值,表明该算法在这些数据集上能更准确地进行数据聚类。相对而言,传统的NMF和rNMF方法的表现较为一致且接近,但普遍低于GNMF和GPNMF。同时,表 3展示了相应的Purity和ACC值。由于Purity指标衡量了真实类别和预测类别之间的最大交集,并且不考虑类别的大小,同时,考虑到scRNA-seq数据中类别大小的不平衡,本文未观察到GPNMF与其他NMF方法在Purity值上的显著差异。
| 数据集 Dataset |
调整兰德指数ARI | 归一化互信息NMI | |||||||||
| NMF | rNMF | GNMF | rGNMF | GPNMF | NMF | rNMF | GNMF | rGNMF | GPNMF | ||
| 10X_PBMC | 0.47 | 0.48 | 0.45 | 0.44 | 0.55 | 0.50 | 0.50 | 0.51 | 0.51 | 0.56 | |
| Adam | 0.42 | 0.42 | 0.48 | 0.56 | 0.49 | 0.58 | 0.57 | 0.67 | 0.70 | 0.67 | |
| CITE_CBMC | 0.47 | 0.48 | 0.45 | 0.44 | 0.55 | 0.50 | 0.50 | 0.51 | 0.51 | 0.56 | |
| HumanLiver | 0.47 | 0.48 | 0.45 | 0.44 | 0.55 | 0.50 | 0.50 | 0.51 | 0.51 | 0.56 | |
| Macosko mouse | 0.47 | 0.48 | 0.45 | 0.44 | 0.55 | 0.50 | 0.50 | 0.51 | 0.51 | 0.56 | |
| Muraro | 0.88 | 0.90 | 0.87 | 0.87 | 0.87 | 0.83 | 0.84 | 0.83 | 0.83 | 0.83 | |
| Bladder | 0.75 | 0.75 | 0.76 | 0.76 | 0.76 | 0.79 | 0.79 | 0.80 | 0.80 | 0.80 | |
| 10X_Muscle | 0.79 | 0.79 | 0.95 | 0.95 | 0.96 | 0.84 | 0.85 | 0.94 | 0.94 | 0.95 | |
| Spleen | 0.50 | 0.51 | 0.87 | 0.60 | 0.86 | 0.57 | 0.57 | 0.82 | 0.66 | 0.82 | |
| Diaphragm | 0.97 | 0.97 | 0.96 | 0.96 | 0.96 | 0.95 | 0.95 | 0.92 | 0.92 | 0.93 | |
| QS_Muscle | 0.60 | 0.60 | 0.65 | 0.66 | 0.65 | 0.76 | 0.76 | 0.82 | 0.82 | 0.82 | |
| QS_Lung | 0.61 | 0.60 | 0.77 | 0.77 | 0.59 | 0.79 | 0.79 | 0.83 | 0.83 | 0.80 | |
| QS_Trachea | 0.88 | 0.87 | 0.87 | 0.87 | 0.87 | 0.81 | 0.78 | 0.85 | 0.85 | 0.85 | |
| Romanov | 0.47 | 0.48 | 0.45 | 0.44 | 0.55 | 0.50 | 0.50 | 0.51 | 0.51 | 0.56 | |
| Shekhar mouse | 0.47 | 0.48 | 0.45 | 0.44 | 0.55 | 0.50 | 0.50 | 0.51 | 0.51 | 0.56 | |
| Young | 0.32 | 0.35 | 0.54 | 0.53 | 0.44 | 0.52 | 0.54 | 0.68 | 0.67 | 0.59 | |
| human_kidney | 0.47 | 0.48 | 0.45 | 0.44 | 0.55 | 0.50 | 0.50 | 0.51 | 0.51 | 0.56 | |
| mouse_ES | 0.57 | 0.76 | 0.95 | 0.95 | 0.95 | 0.70 | 0.80 | 0.92 | 0.92 | 0.92 | |
| worm_neuron | 0.47 | 0.48 | 0.45 | 0.44 | 0.55 | 0.50 | 0.50 | 0.51 | 0.51 | 0.56 | |
| GSE75748 | 0.60 | 0.60 | 0.58 | 0.60 | 0.65 | 0.72 | 0.72 | 0.70 | 0.71 | 0.76 | |
| Note: data in bold represent the optimal results. | |||||||||||
| 数据集 Dataset |
纯度Purity | 准确率ACC | |||||||||
| NMF | rNMF | GNMF | rGNMF | GPNMF | NMF | rNMF | GNMF | rGNMF | GPNMF | ||
| 10X_PBMC | 0.80 | 0.80 | 0.81 | 0.80 | 0.84 | 0.65 | 0.66 | 0.63 | 0.63 | 0.68 | |
| Adam | 0.65 | 0.65 | 0.73 | 0.75 | 0.73 | 0.64 | 0.64 | 0.68 | 0.67 | 0.69 | |
| CITE_CBMC | 0.80 | 0.80 | 0.81 | 0.80 | 0.84 | 0.65 | 0.66 | 0.63 | 0.63 | 0.68 | |
| HumanLiver | 0.80 | 0.80 | 0.81 | 0.80 | 0.84 | 0.65 | 0.66 | 0.63 | 0.63 | 0.68 | |
| Macosko mouse | 0.80 | 0.80 | 0.81 | 0.80 | 0.84 | 0.65 | 0.66 | 0.63 | 0.63 | 0.68 | |
| Muraro | 0.92 | 0.93 | 0.91 | 0.91 | 0.91 | 0.92 | 0.93 | 0.91 | 0.91 | 0.91 | |
| Bladder | 0.97 | 0.97 | 0.97 | 0.97 | 0.97 | 0.74 | 0.74 | 0.79 | 0.79 | 0.79 | |
| 10X_Muscle | 0.90 | 0.90 | 0.98 | 0.98 | 0.98 | 0.82 | 0.82 | 0.98 | 0.98 | 0.98 | |
| Spleen | 0.93 | 0.93 | 0.98 | 0.96 | 0.98 | 0.72 | 0.72 | 0.95 | 0.82 | 0.95 | |
| Diaphragm | 0.99 | 0.99 | 0.98 | 0.98 | 0.98 | 0.99 | 0.99 | 0.98 | 0.98 | 0.98 | |
| QS_Muscle | 0.91 | 0.91 | 0.95 | 0.95 | 0.95 | 0.72 | 0.72 | 0.71 | 0.73 | 0.71 | |
| QS_Lung | 0.91 | 0.89 | 0.90 | 0.90 | 0.89 | 0.70 | 0.67 | 0.74 | 0.74 | 0.61 | |
| QS_Trachea | 0.94 | 0.92 | 0.95 | 0.95 | 0.95 | 0.94 | 0.92 | 0.95 | 0.95 | 0.95 | |
| Romanov | 0.80 | 0.80 | 0.81 | 0.80 | 0.84 | 0.65 | 0.66 | 0.63 | 0.63 | 0.68 | |
| Shekhar mouse | 0.80 | 0.80 | 0.81 | 0.80 | 0.84 | 0.65 | 0.66 | 0.63 | 0.63 | 0.68 | |
| Young | 0.61 | 0.64 | 0.73 | 0.72 | 0.63 | 0.48 | 0.52 | 0.63 | 0.61 | 0.58 | |
| human_kidney | 0.80 | 0.80 | 0.81 | 0.80 | 0.84 | 0.65 | 0.66 | 0.63 | 0.63 | 0.68 | |
| mouse_ES | 0.77 | 0.88 | 0.98 | 0.98 | 0.98 | 0.77 | 0.80 | 0.98 | 0.98 | 0.98 | |
| worm_neuron | 0.80 | 0.80 | 0.81 | 0.80 | 0.84 | 0.65 | 0.66 | 0.63 | 0.63 | 0.68 | |
| GSE75748 | 0.75 | 0.75 | 0.74 | 0.75 | 0.78 | 0.69 | 0.69 | 0.73 | 0.73 | 0.76 | |
| Note: data in bold represent the optimal results. | |||||||||||
综合来看,GPNMF在多个数据集上表现出优异的聚类能力,显示了其在处理不同类型数据集时的鲁棒性和稳定性。rGNMF也展现了较好的性能,尤其是在处理复杂数据集时。传统的NMF和rNMF方法虽然在某些数据集上表现尚可,但总体来说略逊于GNMF和GPNMF。通过这次实验可以得出以下结论:改进的GPNMF方法在多种实际数据集上的表现更加优越,适用于更广泛的数据分析场景。
3.3 数据可视化外周血单核细胞(Peripheral Blood Mononuclear Cells, PBMC)数据集是由10x Genomics公司提供的公开数据集,专为scRNA-seq而设计。该数据集收集了人类外周血单核细胞的RNA表达数据,旨在揭示不同类型细胞之间的转录组差异和细胞类型的多样性。本文通过散点图对数据集中的各簇进行可视化,并使用残差-相似度(Residue-Similarity,RS)图来分析降维后特征的聚类情况[21]。
经过NMF降维处理的散点图直观展现了数据结构,将原始高维数据转换到易于理解的二维空间。每个点代表一个样本,其位置由其两个主要特征的数值决定。通过散点图可以清晰地观察样本在这两个关键特征上的分布情况,并探索可能存在的聚类结构或分布规律。从图 1可以明显看出,由GPNMF所得到的散点图中,离群点较少,而且聚类结构非常明显。相比之下,其他算法所得到的散点图离群点较多,聚类结构不够明显。这种对比分析显示了GPNMF在数据降维和聚类方面的优异性,为后续数据分析和模式识别提供了有力支持。
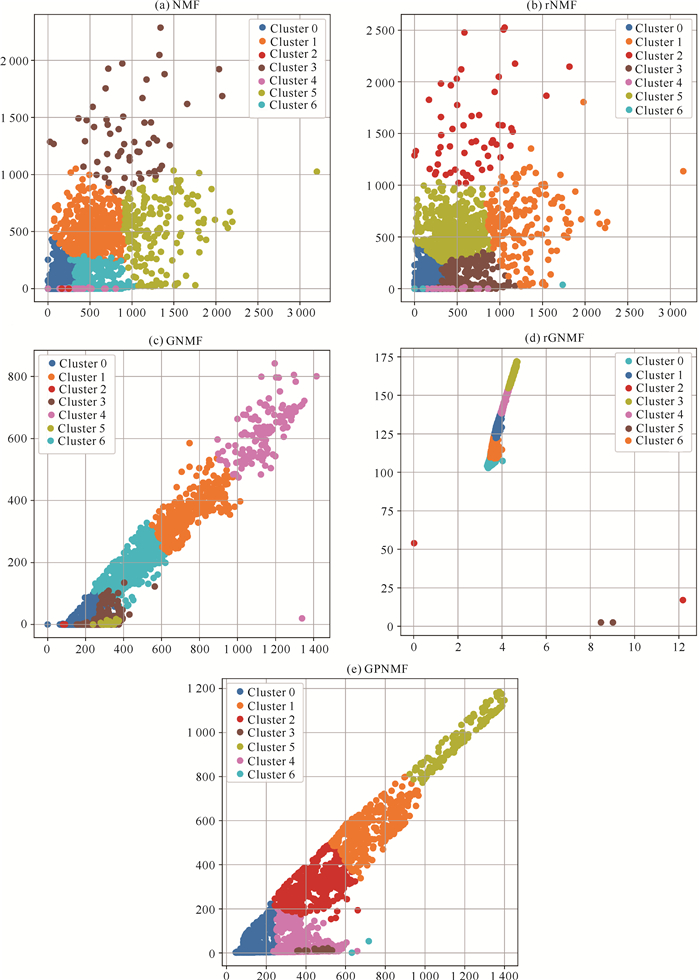
|
| 图 1 PBMC数据集特征散点图 Fig. 1 Scatter plot of PBMC dataset |
RS图是一种用于衡量模型重构误差的方法,可以帮助改进NMF模型处理高噪声和稀疏数据的不足[22]。残差分数(R分数)表示原始数据样本和重建数据样本之间的差异,分数越低越好。相似度分数(S分数)通常表示每个数据点与某个参考点(如聚类中心或重建数据点)之间距离的最小值,而不是表示相似度的最小值。这个参考点可以是重建的数据点,也可以是聚类中心,具体取决于应用场景和算法。S分数的具体意义和计算方式会根据具体的数据分析任务和算法而有所不同,因此不能简单地根据分数的高低来判断。本文中S分数表示每个原始数据样本与其最近重建数据样本之间距离的最小值。如图 2所示,在RS图的比较分析中,X轴表示每个样本的R分数,Y轴表示每个样本的S分数。样本根据k-Means聚类算法预测的细胞类型进行着色,使用聚类结果获得预测类别,并采用匈牙利算法寻找聚类预测标签与真实细胞类型标签之间的最佳映射。综合来看,不同算法生成的RS图中,GPNMF的R分数平均值较低,且呈现较窄的分布,S分数跨度很大且分布均匀,可能暗示着数据集在聚类或降维后,数据点之间的距离差异较大,但重建模型在大多数数据点上的效果良好。
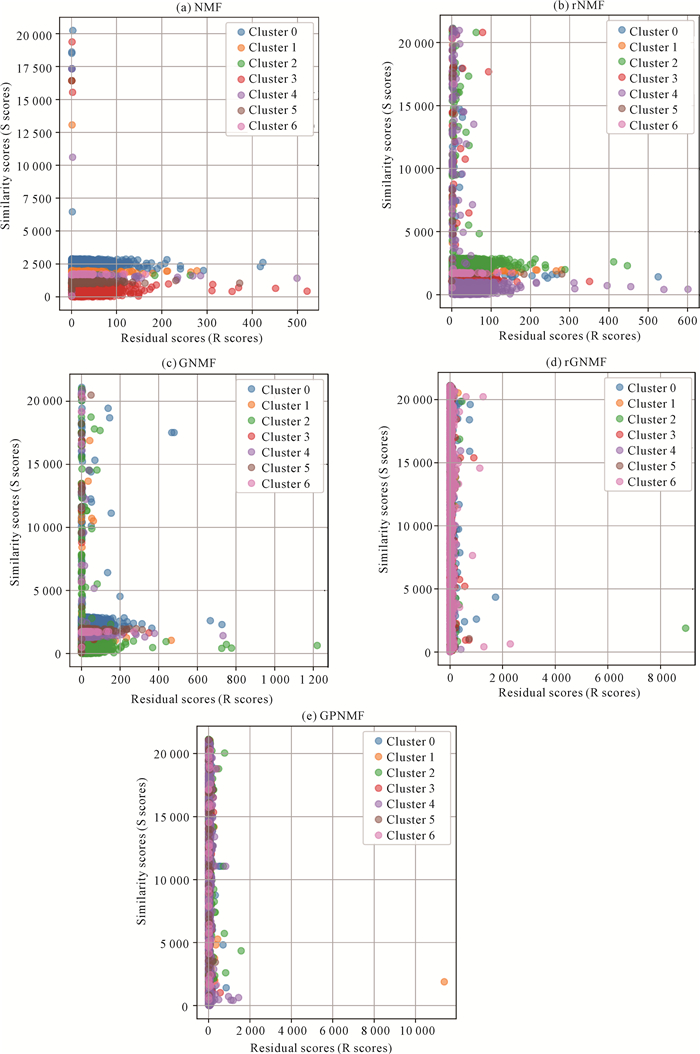
|
| 图 2 10X_PBMC数据集RS图 Fig. 2 RS plot of 10X_PBMC dataset |
3.4 轨迹推断分析
胰腺内分泌细胞在胰腺结构与功能中扮演着关键角色,其真实轨迹状态的描述对理解其生物学功能和调控机制至关重要。本文选择胰腺数据集[23]中第15天的内分泌细胞进行深入分析。内分泌细胞主要包括胰岛素产生的β细胞、胰高血糖素产生的α细胞等,它们在胰腺中的分布与功能状态随时间和环境条件的变化而变化。生物学上,这些细胞从幼年阶段的增殖和分化,到成熟后的功能表达和分泌调节,都展现出明显的生理和代谢变化。因此,所用的胰腺数据集需要进行预处理,包括去除低质量细胞、标准化和归一化等步骤,以确保数据质量。胰腺数据集涵盖了胰腺中多种细胞类型的单细胞转录组数据,主要包括内分泌和外分泌细胞。内分泌细胞在胰腺中的真实轨迹状态可以通过它们在不同发育阶段和环境条件下的转录组特征来描述。例如,胰岛素产生的β细胞在胰腺内的分化过程中可能表现出特定的基因表达模式,反映其从幼年期到成熟期的功能逐渐成熟和增强; 而胰高血糖素产生α细胞则可能在不同的代谢状态下调节其分泌功能,对血糖水平的调节具有重要意义。这些细胞类型的轨迹状态分析,有助于揭示胰腺内分泌系统的功能变化及其在疾病发展中的潜在作用。图 3展示了胰腺细胞在第15天的真实轨迹推断,反映了内分泌细胞在特定发育阶段的分化和动态过程。图 3表明了细胞从未分化状态向成熟细胞类型转变的路径,揭示了细胞分化的起点、分叉点和终点,提供了对细胞发育顺序和分化节点的详细见解。
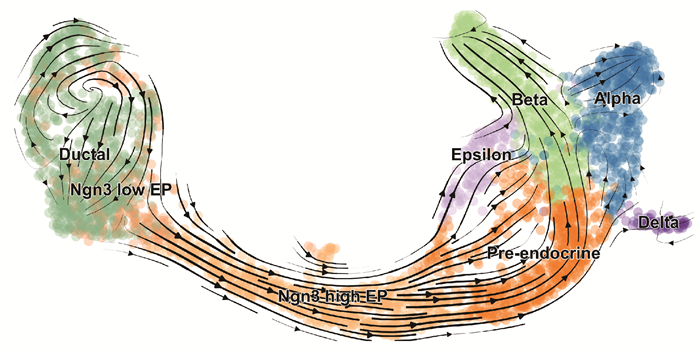
|
| 图 3 胰腺细胞第15天真实轨迹推断 Fig. 3 Real trajectory inference of pancreas cell on day 15 |
有关轨迹推断的实验,首先对5种不同NMF方法的解释方差比率(Explained Variance Ratio,EVR)指标[24]和均方根误差(RMSE)指标[25]进行计算。EVR用于衡量降维后数据保留原始数据变异性的程度。正值表示模型解释了部分数据的方差,而负值则表示模型在解释方差时出现了问题。RMSE用于衡量降维后的数据与原始数据之间的差异。RMSE越小,表示降维后数据与原始数据越接近。在此次实验中,5种NMF方法的EVR和RMSE分别均为0.57和0.004 6,差异很小,说明它们在保留原始数据变异性和数据重构方面具有较好的效果。虽然结合图正则化和稀疏正则化的GNMF、rGNMF和GPNMF方法在理论上有所改进,但实际结果并未显著优于传统的NMF和rNMF方法。
其次对胰腺数据集中第15天的内分泌数据进行轨迹推断分析可视化,评估不同NMF方法生成的轨迹图像的性能,并详细分析其在细胞排列、流向和聚集情况等方面的表现。具体步骤如下: 应用NMF将原始scRNA-seq数据从高维空间映射到低维空间。NMF生成的低维 H矩阵提供了细胞在低维空间中的坐标。然后通过scvelo工具包,将NMF得到的低维坐标用于生成轨迹图[26], 从而比较不同NMF方法生成的低维坐标对轨迹推断的影响。在本实验中,scvelo工具包设置随机模式计算速度向量,从而能够直观地展示和分析scRNA-seq数据中的轨迹信息,揭示细胞的发育路径和动态变化过程。
如图 4所示,在胰腺细胞第15天的轨迹数据集中,起点通常是神经内胚层细胞(Neurogenin3 positive cells,NgN3),这些细胞是胰腺内胚层细胞的前体细胞; 终点则是成熟的胰岛细胞(如Beta细胞和Delta细胞)和胰腺导管细胞(Ductal cells)。5种NMF方法都能正常表示这些细胞类型的轨迹推断结果。传统的NMF和rNMF得到的细胞排列较为均匀,显示出不同细胞类型之间的渐变过渡,但部分区域的细胞聚集较为密集; 轨迹流向整体较为流畅,在某些区域存在回流现象,可能不完全符合生物学上的预期路径; 细胞聚集情况较为分散,虽然能够区分出不同的细胞类型,但界限不够清晰。GNMF得到的细胞排列较为均匀,显示出较好的细胞类型过渡,但在某些区域细胞聚集过于密集; 轨迹流向整体较为顺畅,但在某些区域存在较多的分叉,可能影响对细胞运动路径的理解; 细胞聚集情况合理,不同细胞类型之间的界限清晰,但某些细胞类型的分布可能过于紧密。rGNMF和GP_rGNMF得到的细胞类型渐变过渡更为明显,与GNMF相比有所改进; 轨迹流向非常顺畅,符合生物学上的预期路径; 细胞聚集情况非常合理,不同细胞类型之间的界限清晰,细胞类型的分布较为均匀。观察5种NMF方法细胞聚集成团的情况,整体上能够准确区分不同类型的细胞。然而,部分轨迹的分叉并没有得到良好体现。例如,α细胞和β细胞在轨迹图中无法清晰分离。
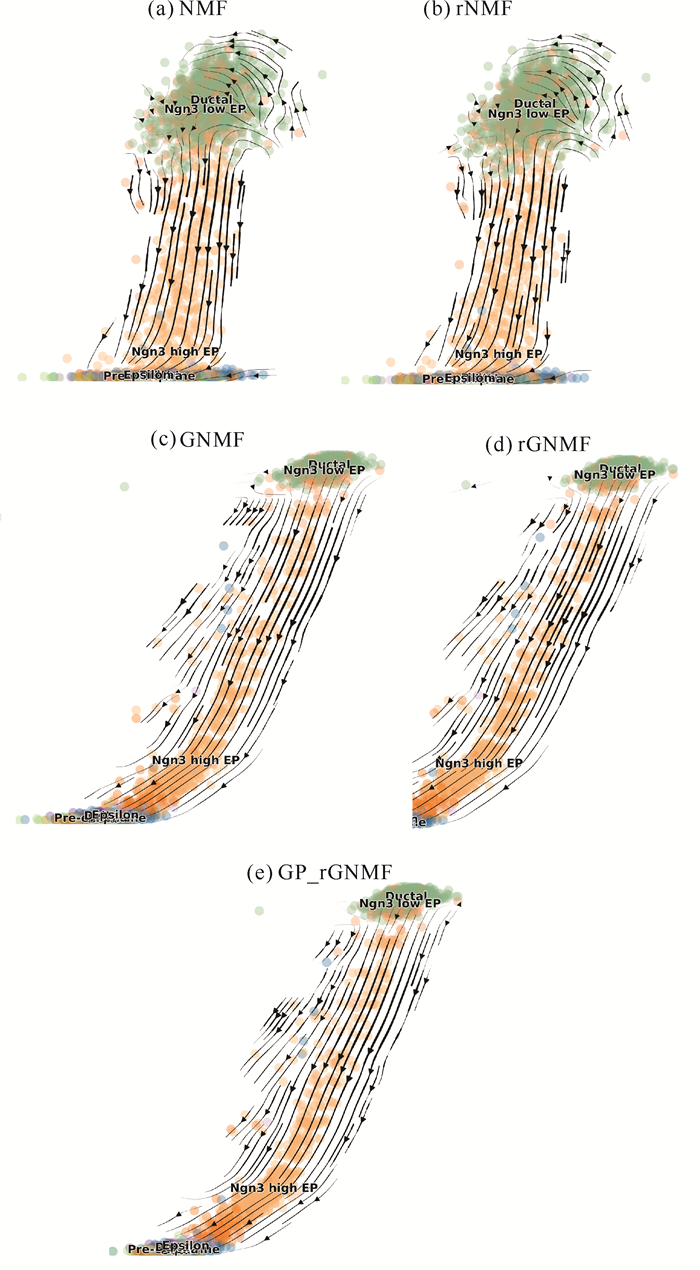
|
| 图 4 5种NMF方法的胰腺第15天轨迹推断图 Fig. 4 Trajectory inference plots of pancreas on day 15 by five NMF methods |
4 讨论
在高丢失率(> 70%)的情况下使用GPNMF对scRNA-seq数据进行降维,这种高丢失率在基于液滴的测序技术中很常见,例如10x Genomics技术,它正逐渐成为scRNA-seq的主导技术。传统的NMF方法未充分考虑单细胞数据的零膨胀特征,而伽玛-泊松分布是一种能够处理高丢失率和零膨胀现象的概率分布,特别适用于scRNA-seq数据。伽玛分布作为泊松分布的先验,能够在一定程度上捕捉数据的稀疏性和多样性。GPNMF方法采用伽玛-泊松分布对基因表达建模,更好地反映了数据的潜在结构。另外,GPNMF结合了图正则化,通过构建细胞-细胞相互作用图,在一定程度上捕捉了细胞间的局部相似性和全局结构。图拉普拉斯正则化项帮助保持细胞间的拓扑关系,从而在降维过程中保留更多的生物学信息。实验结果显示,GPNMF在ARI和NMI指标上表现优异,相较于其他NMF方法,在所有数据集中达到了最高或接近最高的ARI和NMI值,这表明其在聚类准确性和类别信息保留方面具有显著优势。ARI和NMI分别衡量聚类结果与真实标签的一致性和信息量。GPNMF在这两个指标上表现出的优势,说明其在处理高丢失率和零膨胀数据方面具有较强的适应性和鲁棒性。尽管GPNMF在理论上具有优越性,但其假设数据符合伽玛-泊松分布,可能在某些情况下存在偏差。scRNA-seq数据中的零值可能源于技术噪声,而非真实的生物学信号。未来研究可以进一步探索如何更好地建模零膨胀现象,或结合其他概率分布以提高模型的灵活性和适应性。
5 结束语本文提出一种单细胞非负矩阵分解算法GPNMF,基于伽玛-泊松分布和图正则化,对scRNA-seq数据表现出良好的性能和应用前景。为了进一步提高NMF方法在单细胞数据分析中的效果和应用价值,未来可从以下方向对该算法进行优化。首先,探索无需参数设定的GNMF方法,解决当前方法在k近邻参数选择上的不确定性和主观性,这有助于更好地挖掘单细胞数据的拓扑结构,提高细胞类型分类和细胞谱系推断的准确性。其次,研究新的矩阵初始化方法,以更有效地初始化NMF模型参数,进而加速模型收敛并提高降维结果的稳定性。合适的初始化方法对于NMF算法的性能至关重要,尤其在处理高维稀疏数据时。最后,探索更合适的核函数以构造拉普拉斯邻接矩阵,从而更准确地反映细胞间的相互作用和关联关系。拉普拉斯邻接矩阵的构建对图正则化NMF算法的效果有着关键影响,因此寻找更合适的核函数可以进一步提高算法的性能。这些改进将有助于更好地揭示单细胞数据的潜在特征,提取更具解释性的特征,并推动单细胞数据分析方法的发展和应用。随着这些研究方向的探索和发展,基于概率分布的GNMF方法在单细胞数据分析领域将展现出更广阔的应用前景。
| [1] |
LÄHNEMANN D, KÖSTER J, SZCZUREK E, et al. Eleven grand challenges in single-cell data science[J]. Genome Biology, 2020, 21(1): 31. DOI:10.1186/s13059-020-1926-6 |
| [2] |
JIANG H, WANG M N, HUANG Y A, et al. Graph-Regularized non-negative matrix factorization for single-cell clustering in scRNA-Seq data[J]. IEEE Journal of Biomedical and Health Informatics, 2024, 28(8): 4986-4994. DOI:10.1109/JBHI.2024.3400050 |
| [3] |
HICKS S C, TOWNES F W, TENG M, et al. Missing data and technical variability in single-cell RNA-sequencing experiments[J]. Biostatistics, 2018, 19(4): 562-578. DOI:10.1093/biostatistics/kxx053 |
| [4] |
LIU W X, ZHENG N N, YOU Q B. Nonnegative matrix factorization and its applications in pattern recognition[J]. Chinese Science Bulletin, 2006, 51(1): 7-18. DOI:10.1007/s11434-005-1109-6 |
| [5] |
KONG D G, DING C, HUANG H, et al. Robust nonnegative matrix factorization using L21-norm [C]//Proceedings of the 20th ACM International Conference on Information and Knowledge Management. New York: ACM, 2011: 673-682.
|
| [6] |
XIAO Q, LUO J W, LIANG C, et al. A graph regularized non-negative matrix factorization method for identifying microRNA-disease associations[J]. Bioinformatics, 2018, 34(2): 239-248. DOI:10.1093/bioinformatics/btx545 |
| [7] |
SHU Z Q, LONG Q H, ZHANG L P, et al. Robust graph regularized NMF with dissimilarity and similarity constraints for ScRNA-seq data clustering[J]. Journal of Chemical Information and Modeling, 2022, 62(23): 6271-6286. DOI:10.1021/acs.jcim.2c01305 |
| [8] |
DURIF G, MODOLO L, MOLD J E, et al. Probabilistic count matrix factorization for single cell expression data analysis[J]. Bioinformatics, 2019, 35(20): 4011-4019. DOI:10.1093/bioinformatics/btz177 |
| [9] |
XU J J, WANG Y S, XU X N, et al. NMF-based ap-proach for missing values imputation of mass spectrometry metabolomics data[J]. Molecules, 2021, 26(19): 5787. DOI:10.3390/molecules26195787 |
| [10] |
HEDIYEH-ZADEH S, WEBB A I, DAVIS M J. MSI-mpute: imputation of label-free mass spectrometry peptides by low-rank approximation [EB/OL]. (2020-08-13)[2024-03-23]. https://doi.org/10.1101/2020.08.12.248963.
|
| [11] |
SI T, HOPKINS Z, YANEV J, et al. A novel f-divergence based generative adversarial imputation method for scRNA-seq data analysis[J]. PLoS One, 2023, 18(11): e0292792. DOI:10.1371/journal.pone.0292792 |
| [12] |
QIU Y S, YAN C, ZHAO P, et al. SSNMDI: a novel joint learning model of semi-supervised non-negative matrix factorization and data imputation for clustering of single-cell RNA-seq data[J]. Briefings in Bioinformatics, 2023, 24(3): bbad149. DOI:10.1093/bib/bbad149 |
| [13] |
AHLMANN-ELTZE C, HUBER W. glmGamPoi: fitting Gamma-Poisson generalized linear models on single cell count data[J]. Bioinformatics, 2021, 36(24): 5701-5702. DOI:10.1093/bioinformatics/btaa1009 |
| [14] |
ZAPPIA L, PHIPSON B, OSHLACK A. Splatter: simulation of single-cell RNA sequencing data[J]. Genome Biology, 2017, 18(1): 174. DOI:10.1186/s13059-017-1305-0 |
| [15] |
HUANG M, WANG J S, TORRE E, et al. SAVER: gene expression recovery for single-cell RNA sequencing[J]. Nature Methods, 2018, 15(7): 539-542. DOI:10.1038/s41592-018-0033-z |
| [16] |
GRVN D, LYUBIMOVA A, KESTER L, et al. Single-cell messenger RNA sequencing reveals rare intestinal cell types[J]. Nature, 2015, 525: 251-255. DOI:10.1038/nature14966 |
| [17] |
ELYANOW R, DUMITRASCU B, ENGELHARDT B E, et al. netNMF-sc: leveraging gene–gene interactions for imputation and dimensionality reduction in single-cell expression analysis[J]. Genome Research, 2020, 30(2): 195-204. DOI:10.1101/gr.251603.119 |
| [18] |
WEI N N, NIE Y T, LIU L, et al. Secuer: ultrafast, scalable and accurate clustering of single-cell RNA-seq data[J]. PLoS Computational Biology, 2022, 18(12): e1010753. DOI:10.1371/journal.pcbi.1010753 |
| [19] |
CHU L F, LENG N, ZHANG J, et al. Single-cell RNA-seq reveals novel regulators of human embryonic stem cell differentiation to definitive endoderm[J]. Genome Biology, 2016, 17(1): 173. DOI:10.1186/s13059-016-1033-x |
| [20] |
PAN W Q, LONG F N, PAN J. ScInfoVAE: interpretable dimensional reduction of single cell transcription data with variational autoencoders and extended mutual information regularization[J]. BioData Mining, 2023, 16(1): 17. DOI:10.1186/s13040-023-00333-1 |
| [21] |
HOZUMI Y, WANG R, WEI G W. CCP: correlated cl-ustering and projection for dimensionality reduction [EB/OL]. (2022-06-08)[2024-03-23]. https://arxiv.org/abs/2206.04189.
|
| [22] |
FENG H S, WEI G W. Virtual screening of drugbank database for hERG blockers using topological Laplacian-assisted AI models[J]. Computers in Biology and Medicine, 2023, 153: 106491. DOI:10.1016/j.compbiomed.2022.106491 |
| [23] |
BARON M, VERES A, WOLOCK S L, et al. A single-cell transcriptomic map of the human and mouse pancreas reveals inter- and intra-cell population structure[J]. Cell Systems, 2016, 3(4): 346-360.e4. DOI:10.1016/j.cels.2016.08.011 |
| [24] |
ZOU H, HASTIE T, TIBSHIRANI R. Sparse principal component analysis[J]. Journal of Computational and Graphical Statistics, 2006, 15(2): 265-286. DOI:10.1198/106186006X113430 |
| [25] |
WILLMOTT C J, MATSUURA K. Advantages of the mean absolute error (MAE) over the root mean square error (RMSE) in assessing average model performance[J]. Climate Research, 2005, 30: 79-82. DOI:10.3354/cr030079 |
| [26] |
BERGEN V, LANGE M, PEIDLI S, et al. Generalizing RNA velocity to transient cell states through dynamical modeling[J]. Nature Biotechnology, 2020, 38: 1408-1414. DOI:10.1038/s41587-020-0591-3 |



