2. 广西图像图形与智能处理重点实验室, 广西桂林 541004
2. Guangxi Key Laboratory of Image and Graphic Intelligent Processing, Guilin, Guangxi, 541004, China
随着医学影像技术的快速发展,共振成像(MR)、计算机断层扫描(CT)、超声、正电子发射断层扫描(PET)等成为医疗机构开展疾病诊断、手术计划制定、预后评估和随访必不可少的设备,因而产生了大量的医学影像数据。医学图像处理是分析医学图像的首要步骤,使得图像更加直观、清晰,提高诊断效率,因此国内外都十分重视医学影像处理研究[1-4]。医学图像分割是图像处理的重要环节和主要难点,也是制约三维重建等技术应用的瓶颈性问题。细胞核的分割结果是病理学家进行癌症分类和评级的重要依据。截至目前,病理图像细胞核分割问题存在的主要难点包括:细胞图像复杂且细胞种类繁多,每种细胞处于的生长阶段不同,其形态却有可能相同; 来自多中心的细胞病理图像受染色不均、光照条件不一致等的影响较大,灰度值容易发生变化[5, 6]; 细胞图像经常重叠,没有明显的边界; 细胞的大小变化很大,细胞核形态各异[7]。
为解决病理图像细胞核分割的难题,研究人员借助计算机技术辅助实现细胞核的自动分割,这具有重要的医学价值。传统的分割方法包括基于阈值的分割和分水岭分割算法。阈值分割法[8]是一种基于区域的图像分割技术。龙建武等[9]提出自适应阈值分割与Canny边缘检测算子相结合的方法,并基于高斯径向基函数,利用边缘检测算子检测到的边缘点灰度均值作为分割阈值来提取目标,该方法能够检测出模糊的小目标。分水岭分割算法是另一种比较经典的、基于数学形态学的分割方法[10, 11],具体分为排序和淹没两个过程,其主要缺点是图像中的噪声等会导致过度分割。
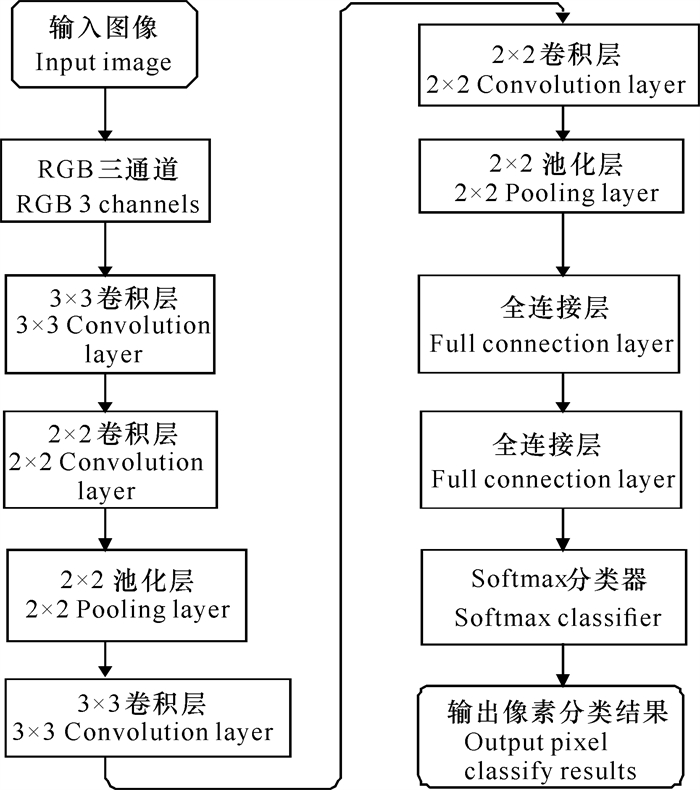
近年来,随着深度学习技术的不断发展,越来越多的细胞分割采用深度学习方法,其性能往往优于传统机器学习方法,是当前主流的细胞核分割算法[12-15]。例如,图 1所示为基于卷积神经网络(CNN)的细胞分割网络结构图[16-18],该网络包含3个卷积层。首先将预处理后的原始图像输入到卷积网络的第一层,使用3×3的卷积核进行特征提取;然后将结果输入到第二层网络,进行2×2的卷积操作后激活并进行最大池化,接着进入第三、第四层网络;最后使用全连接层和Softmax函数作为分类器,输出各个像素的分类结果。李宗民等[19]采用一种基于生成对抗网络的分割模型(该网络结合对抗网络GAN和自编码器[20]),设计一种针对细胞分割的Cell-GAN, 然后基于训练学习到的细胞形态学知识来对细胞图像的信息进行判断,并引入单个细胞核指导因子,用于分割细胞的精确定位。韩文忠等[21]使用全卷积网络(FCN)进行磁共振成像(MRI)膀胱图像的分割,相比于CNN,该网络完全舍弃了全连接层,且能够接收任意尺寸的输入,实现了端到端的训练。
|
| 图 1 基于CNN的细胞分割流程图 Fig.1 Flowchart of CNN-based cell segmentation |
重叠、图像对比度低以及杂质干扰是制约细胞图像分割的主要问题。传统方法比较依赖于参数的调节,往往只能针对特定的细胞环境,在复杂环境中的分割效果不好。尽管深度学习实现了在复杂环境下的较好分割,但是这种优势仅局限于单细胞分割,无法有效地分割重叠细胞图像。为此,本研究采用深度学习的思想,提出一种基于U-Net[22]网络和残差网络(ResNet)[23]的新型语义分割模型,拟在保证细胞核分割准确率较高的前提下,实现相互接触和重叠细胞核图像的精确分割。
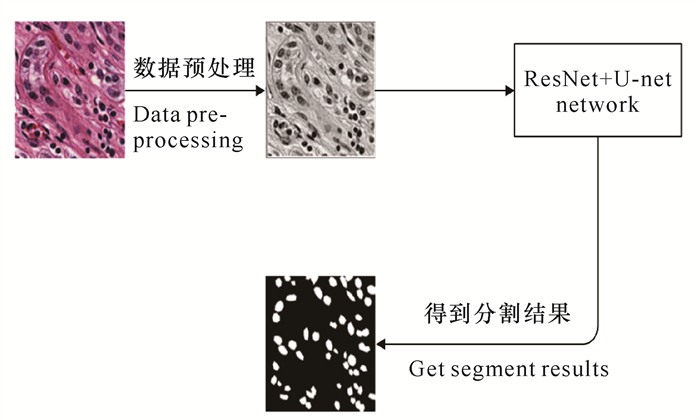
1 模型与方法基于U-Net[22]网络和残差网络(ResNet)[23],本研究提出一种新型语义分割模型。该模型利用不同的残差模块提取多尺度的信息。该模型的数据处理流程如下:首先对数据集进行数据预处理,得到灰度图;然后采用随机划分图像块(Image patch)的方式,将划分的图片连同其标签,送入ResNet+U-Net模型进行若干轮次的训练,保存最佳的模型权重;最后,在测试集上对模型进行测试,将测试集图片送入网络,得到最终的细胞核分割结果(图 2)。
|
| 图 2 数据处理流程 Fig.2 Flowchart of data processing |
1.1 数据预处理
图像预处理可以实现图像的简化、增强和消除无关信息等效果,从而提升特征抽取和图像分割的准确度和精确度。零相位分量分析白化(Zero-phase Component Analysis Whitening,ZCA白化)是一种图像预处理方法,其主要目的是防止数据冗余和维数爆炸。经ZCA白化处理后,不同维度的数据之间具有相关性低、方差相同的特点,从而使数据尽可能接近原始的输入数据。因此,本研究采用ZCA白化进行图像数据的预处理,具体过程如下。
对于含有m个样本的数据集{x(1), x(2), …, x(m)},假设每个样本的维度是n, 即x(i)∈Rn, i=1, …, m。在处理过程中,首先对数据进行亮度和对比度的归一化,对每个像素点x(i)减去图像的灰度均值,再除以标准差(为避免分母为0,标准差加上很小的常数e=0.001)。然后使用公式(1)计算训练样本的协方差矩阵:
| $ \sum = \frac{1}{m}\sum\limits_{i = 1}^m {\left( {{x^{(i)}}} \right){{\left( {{x^{(i)}}} \right)}^{\rm{T}}}} 。$ | (1) |
求出数据集的协方差矩阵后,对其进行奇异值分解(SVD),得到特征矩阵U。利用UTx得到数据经过旋转后的结果xrio。再将xrio中的每一维除以
| $ x{\;_{{\rm{ZC}}\;{\rm{Awhite}}}} = Ux{\;_{{\rm{PC}}\;{\rm{Awhite}}}}。$ | (2) |
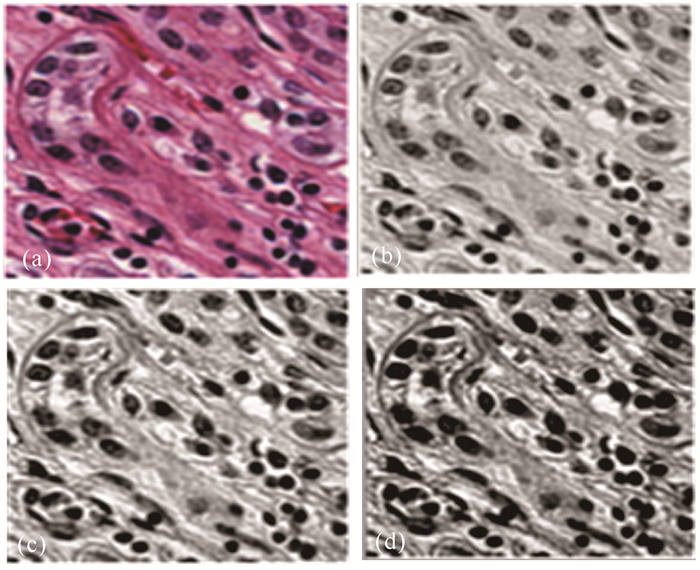
通过ZCA白化使得数据x映射到xZCAwhite空间,特征向量各维度方差相等,数据的重要程度得到统一。从图 3可以看出,ZCA白化之后细胞核和背景的对比度提升最大,这有利于后续网络的特征提取。
|
| (a)原始细胞核切片,(b)PCA方法预处理结果,(c)对比度受限均衡化方法预处理结果,(d)本文ZCA白化的预处理结果 (a) Original nuclei slice, (b) Result of PCA preprocessing, (c) Result of contrast constrained equalization preprocessing, (d) ZCA preprocessing result 图 3 预处理前后图像对比 Fig.3 Image comparison before and after preprocessing |
1.2 基于U-Net和ResNet的网络结构
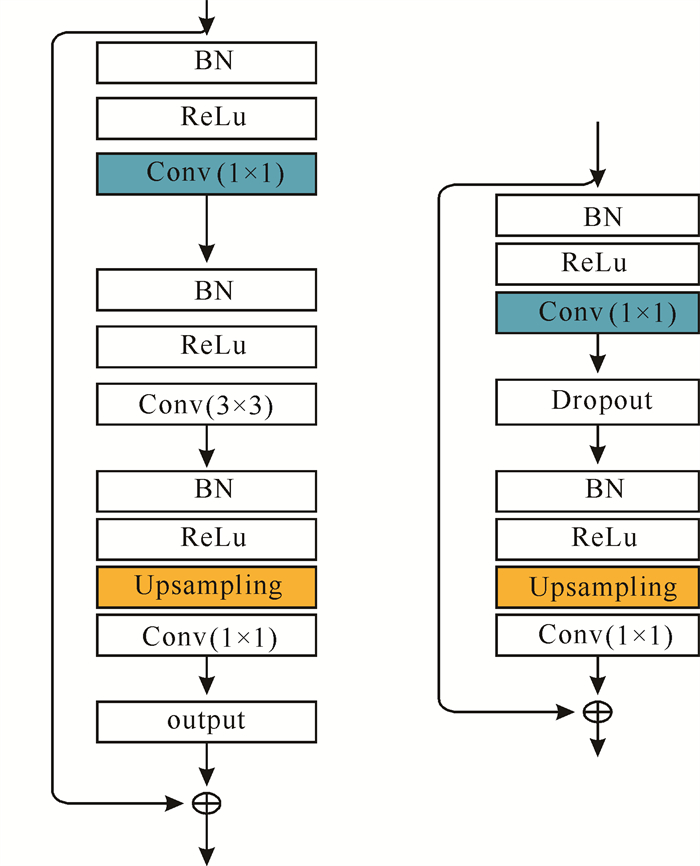
传统的Res-block通常采用两个3×3 Conv对特征图进行卷积,然后和原始输入特征进行短路连接(Short cut connection)。本研究新型细胞核分割网络模型是在U-Net和ResNet网络及两种不同的残差模块结构Res-block 1和Res-block 2的基础上提出的。图 4是Res-block 1和Res-block 2的具体结构,蓝色表示特征编码部分的卷积单元,黄色表示特征解码过程的上采样单元。Res-block 1包括3个卷积模块,依次是1×1 Conv、3×3 Conv和1×1 Conv。使用两层1×1 Conv的目的主要是为了减少计算参数数量: 先用一个1×1 Conv进行降维,使得第二层较大的卷积核个数相对较少,从而减少卷积操作参数的计算量,然后再用一个1×1 Conv进行升维。Res-block 1残差模块采用基于瓶颈(Bottle neck)的结构进行特征编码,同时集合数据正则化处理(BN层)缓解梯度消失。相比于传统的残差模块,Res-block 1具有较强的特征聚合能力,同时参数的计算量显著减少,能够促进模型进行更深层次的特征提取。而Res-block 2的结构相对简单,包括两个1×1 Conv的卷积模块和相应的BN、ReLu模块。Res-block 2在特征解码阶段的上样操作中,采用dropout函数防止模型的过拟合,同时借助跳跃连接(Skip connection)实现特征的融合,有利于分割边缘细节精度的提升。
|
| 图 4 Res-block 1(左)和Res-block 2(右)残差模块结构 Fig.4 Residual modules diagrams of Res-block 1 (left)and Res-block 2 (right) |
图 5展示了本研究基于ResNet和U-Net进行细胞核分割的网络模型,该模型保留了U-Net的基本结构,包括收缩路径和扩张路径两部分。相比于传统U-Net大小单一的卷积核,新模型通过堆叠多个残差模块,利用步长卷积替换传统的池化层(Pooling layer),使不同大小的卷积核实现了多尺度和深层次的特征提取,同时减少了采样过程空间细节信息的丢失。另外,在上采样阶段,新模型通过跳跃连接多尺度的特征融合,将对应收缩路径的高分辨率信息叠加到上采样层,进一步提高医学图像复杂边缘的分割精度。
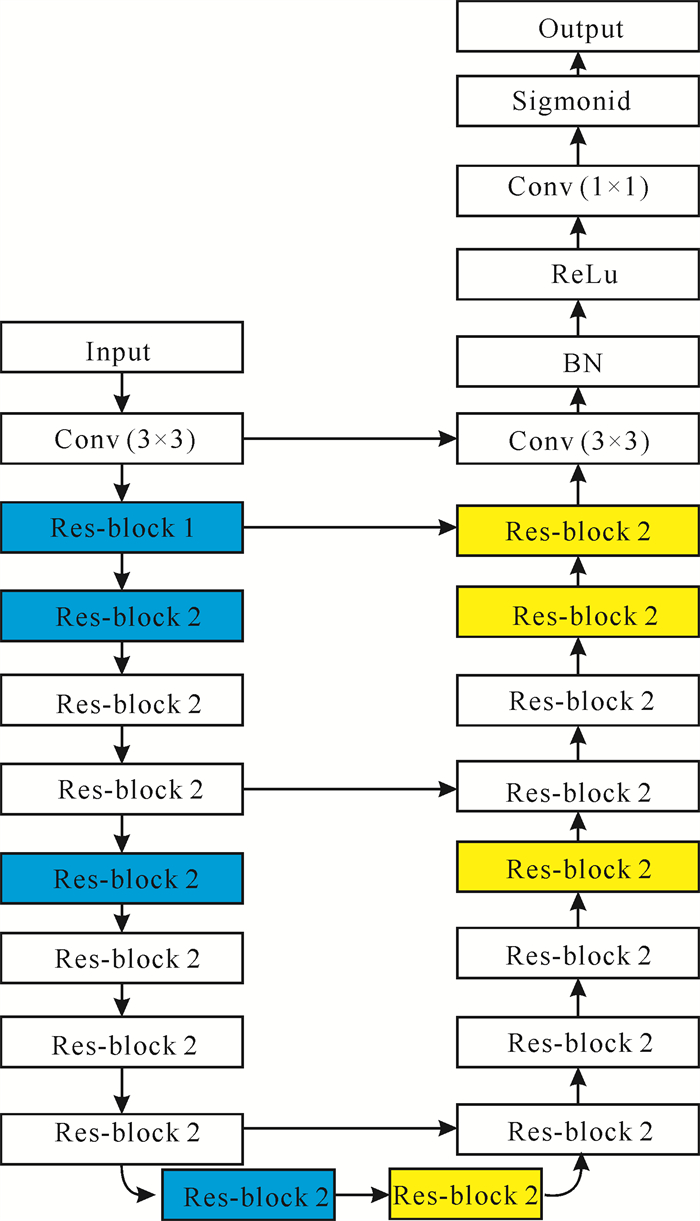
|
| 图 5 网络模型结构 Fig.5 Structure of network model |
在每个残差模块中,新模型都用BN对数据进行归一化处理。BN的基本原理是使用一定规范化方法[24],将网络每一维的输入处理成均值为0、方差为1的标准正态分布,从而加快神经网络的收敛速度,缩短训练时间。具体的算法步骤如下:
输入.训练网络N的参数NBNtr, K维的特征
输出.BN规范化的输出网络结果NBNinf
Step 1.for k=1,…,K do
Step 1.1.将转换函数
Step 1.2.对于训练网络的每一维特征,根据步骤step 1.1,将每一维的x(k)都转化为y(k)。
Step 2.训练NBNinf,得到最优化的训练参数。
Step 3.for k=1, …, K do
Step 3.1.对于包含多个样本的batch B, 用样本的均值和方差代替总体的均值和方差。具体计算方法为
| $ \begin{array}{*{20}{l}} {E\left[ x \right] = {E_B}\left[ {{\mu _B}} \right], }\\ {{\rm{var}}\left[ x \right] = \frac{m}{{m - 1}}{E_B}\left[ {\sigma _B^2} \right]}。\end{array} $ |
其中:E[x]和var[x]分别表示总体数据的均值和方差,μB和σB2分别为样本的均值和方差,EB[μB]和EB[σB2]分别表示样本均值的期望和样本方差的期望,m表示样本的数量。
Step 3.2.运用计算得到的期望和方差,计算出预测值
| $ y = \frac{\alpha }{{\sqrt {{\rm{var}}\left[ x \right] + \varepsilon } }} + \left( {\beta - \frac{{\alpha E\left( x \right)}}{{\sqrt {{\rm{var}}\left[ x \right] + \varepsilon } }}} \right), $ |
其中:E[x]和var[x]分别表示总体数据的均值和方差;α为缩放因子,取值为1.01;β为平移因子,取值为0.99;ε为调节因子,取值为0.001。
训练阶段参数的学习过程如公式(3)所示。表 1为本研究网络模型的参数情况,包含各个模块处理数据的输入和输出大小,以及各模块的使用次数。
| 层名 Layer name |
模块名称 Module name |
输出图片大小 Size of output image |
输出通道数 Number of output channels |
使用次数 Using times |
| DownSample1 | Conv (3×3) | 512×512 | 32 | 1 |
| DownSample2 | Res-block 1 | 256×256 | 32 | 1 |
| DownSample3 | Res-block 2 | 128×128 | 128 | 3 |
| DownSample4 | Res-block 2 | 64×64 | 256 | 8 |
| DownSample5 | Res-block 2 | 32×32 | 512 | 10 |
| Across | Res-block 2 | 32×32 | 1 024 | 3 |
| UpSample1 | Res-block 1 | 64×64 | 512 | 10 |
| UpSample2 | Res-block 2 | 128×128 | 256 | 8 |
| UpSample3 | Res-block 2 | 256×256 | 128 | 3 |
| UpSample4 | Res-block 1 | 512×512 | 32 | 1 |
| UpSample5 | Conv (3×3) | 512×512 | 32 | 1 |
| Classifier | Conv (1×1) | 512×512 | 1 | 1 |
| $ \begin{array}{*{20}{l}} {{\mu _B} \leftarrow \frac{1}{m}\sum\limits_{i = 1}^m {{x_i}, } {\rm{ }}}\\ {\sigma _B^2 \leftarrow \frac{1}{m}\sum\limits_{i = 1}^m {{{\left( {{x_i} - {\mu _B}} \right)}^2}} , }\\ {{{\hat x}_i} \leftarrow \frac{{{x_i} - {\mu _B}}}{{\sigma _B^2 + \varepsilon }}, }\\ {{y_i} = \alpha {{\hat x}_i} + \beta \equiv {\rm{B}}{{\rm{N}}_{\alpha \beta }}\left( {{x_i}} \right)} \end{array} $ | (3) |
式(3)中:μB和σB2分别为样本的均值和方差,
本研究使用交叉熵(Cross entropy)来衡量模型语义分割的精度,使用Dice loss来度量分割细胞核和ground-truth在大小、形态等方面的近似性。Cross entropy和Dice loss分别按公式(4)和公式(5)[25]计算,通过这两种损失函数的评价,可以直观地反映出本研究所提出模型的分割质量。
| $ \begin{array}{l} \;\;\;{\rm{Cross}}\;{\rm{entropy}} = \sum\limits_{i = 1}^n {{y_i}} {\rm{log}}\left( {{{\hat y}_i}} \right) + \\ \left( {1 - {y_i}} \right){\rm{log}}\left( {1 - {{\hat y}_i}} \right), \end{array} $ | (4) |
| $ {\rm{Dice}}\;{\rm{loss}} = - {\rm{ }}\frac{{2\sum\limits_{i = 1}^n {{{\hat y}_i}} {y_i}}}{{\sum\limits_{i = 1}^n {{{\hat y}_i}} + \sum\limits_{i = 1}^n {{y_i}} }}, $ | (5) |
其中,n表示样本的个数,yi表示第i个样本的实际标签,
学习率对于模型训练是非常重要的,实验设定初始学习率为0.001,每30个epoch衰减率为0.7。Batch size也是非常重要的量,而较小的Batch size会使得网络训练得更快,因此本实验中设定Batch size为16。
实验所用的两个细胞核数据集为ISBI2018 Cell和MoNuSeg。其中,MoNuSeg包含30个病人的病理切片,每张切片大小是1 000×1 000 dpi,包含乳腺、肝脏、肾脏、前列腺、胸腺、直肠、胃等7种不同器官的细胞核,分为14张训练集和16张测试集。其中, 训练集包含7种器官,每个器官随机选取两张切片。ISBI2018 Cell是包含30个病人的120张切片的多器官细胞核数据集,由国际医学影像顶级会议ISBI收集。该数据集中的每张切片大小为565×448 dpi,都进行了标准染色(苏木精-伊红染色),且其细胞种类比MoNuSeg更丰富。
为验证Res-block 1和Res-block 2在减少训练时间、提高细胞核分割准确率方面的有效性,本实验基于MoNuSeg数据集,分别从训练时间和分割质量两个角度对新模型进行消融实验。同时,使用MoNuSeg数据集验证所提模型在其他数据集上的泛化能力。
2 结果与分析相比于传统的Res-block,Res-block 1 + Res-block 2可以显著减少U-Net的参数量,在数据集相同、训练轮数相同的前提下,训练和测试的时间是最少的,Dice loss和Cross-entropy也是最小的,在Precision、Recall ratio、F1-score等方面是最高的(表 2,3)。
| 模型名称 Model name |
卷积参数量 Parameter number (m) |
训练时间 Train time (h) |
测试时间 Test time (h) |
| U-Net+Traditional Res-block | 7.75 | 2.3 | 1.2 |
| U-Net+Res-block 1 | 4.12 | 1.8 | 0.9 |
| U-Net+Res-block 2 | 4.97 | 1.9 | 1.0 |
| U-Net+Res-block 1+Res-block 2 | 4.84 | 1.9 | 0.9 |
| 模型名称 Model name |
Dice loss |
交叉熵 Cross-entropy |
精度 Preci-sion |
召回率 Recallratio |
F1分数 F1-score |
| U-Net+Tradi-tional Res-block | 1.528 | 15.2 | 0.921 | 0.881 | 0.898 |
| U-Net+Res-block 1 | 1.123 | 13.1 | 0.937 | 0.890 | 0.909 |
| U-Net+Res-block 2 | 1.089 | 12.6 | 0.934 | 0.883 | 0.907 |
| U-Net+Res-block 1+Res-block 2 | 1.069 | 12.0 | 0.942 | 0.891 | 0.913 |
根据图 6的实验结果来看,新模型算法在分割质量上要优于其他深度学习方法和传统的分水岭算法,相比于U-Net+Attention也有一定提高。如图 7所示,对于目标细胞核和背景对比度差异较大的细胞分割问题,新模型能使目标细胞核之间极少发生直接接触,也没有大面积的粘连现象,这与U-Net+Attention分割结果没有明显的差异。但是,在处理一些存在细胞核间相互粘连的分割问题时,新模型仍然能较好地实现细胞的独立分割,并将不同的细胞核很好地区分开来,展现出比U-Net+Attention更好的性能。这是因为本研究的Res-block 1和Res-block 2实现了更加丰富的特征提取和融合,同时多层次的卷积结构以及BN操作又增加了模型对不同形态细胞核的适应能力,所以分类器能充分利用上下文信息,对前景和背景像素做出更加准确的预测。

|
| 图 6 不同算法在ISBI2018 Cell数据集上的分割质量比较 Fig.6 Comparison of segmentation quality of different algorithms towards ISBI2018 Cell dataset |
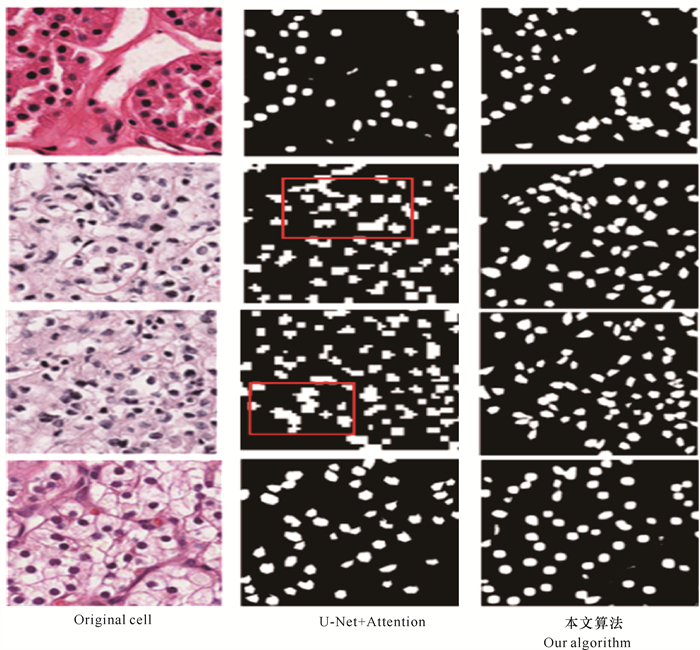
|
| 图 7 本文算法和U-Net+Attention比较 Fig.7 Comparison of our algorithm with U-Net+Attention |
本文模型的算法采用Res-block 1和Res-block 2的改进残差结构,提升了分类器分类的准确率和置信度,是一种高效的分割模型。如表 4所示,相比于其他深度学习的细胞核分割算法,本研究算法在准确率方面有所提升,在precision和F1-score上均是最高的。此外,基于Res-block 1和Res-block 2的改进U-Net模型,本研究算法的交叉熵(Cross-entropy)和Dice loss数都较小,实现了更加丰富的多尺度特征提取和融合,分割结果的假阳性和假阴性更低,更接近真实的细胞核形态。
| 方法 Methods |
Dice loss |
交叉熵 Cross-entropy |
精度 Precision |
召回率 Recallratio |
F1分数 F1-score |
| VGG-16 | 1.303 | 25.3 | 0.819 | 0.765 | 0.796 |
| 分水岭算法 Watershed algorithms |
1.098 | 13.2 | 0.935 | 0.890 | 0.911 |
| CNN3 | 1.311 | 19.5 | 0.836 | 0.855 | 0.893 |
| FCN-8S | 1.109 | 14.1 | 0.917 | 0.887 | 0.901 |
| U-Net+Attention | 1.069 | 12.0 | 0.942 | 0.891 | 0.913 |
| 本文算法 Our algorithm |
1.027 | 9.890 | 0.963 | 0.884 | 0.917 |
本研究使用MoNuSeg数据集验证所提模型在其他数据集上的泛化能力。实验结果的可视化效果如图 8所示, 其中第一列为原始病理图像切片,第二列为对应的细胞核分割的label,第三列为本研究算法的分割结果。从图 8可以看出,本研究模型在其他细胞核数据集也取得较好的分割结果。
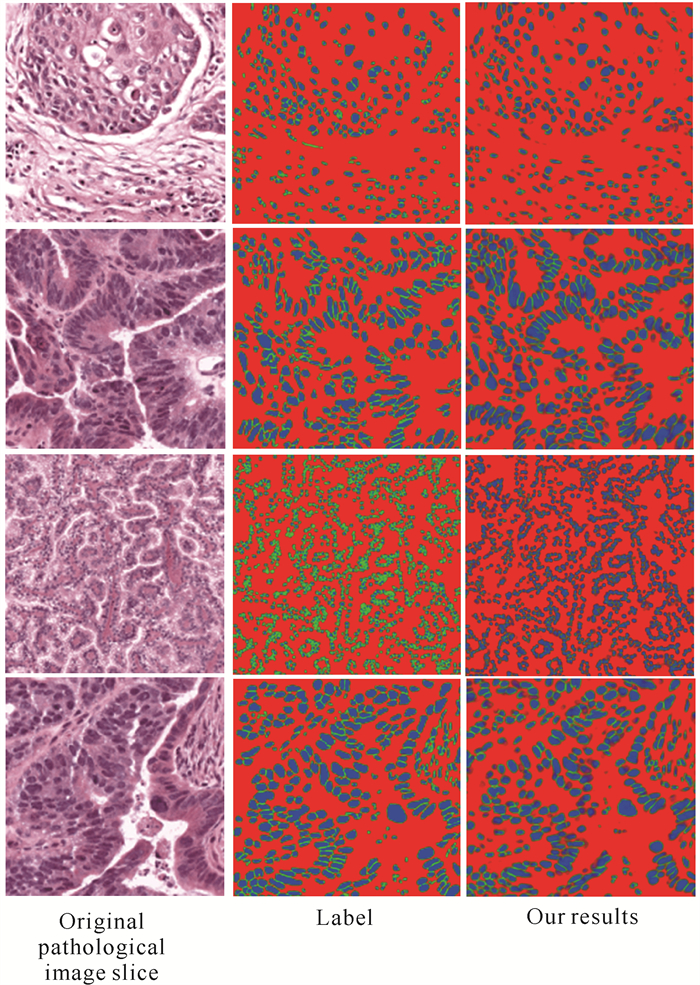
|
| 图 8 本文算法在MoNuSeg数据集的测试结果 Fig.8 Test results of our algorithm on the MoNuSeg dataset |
3 结论
本研究基于深度学习的方法,结合广泛用于医学图像分割的U-Net网络以及ResNet网络中的残差模块,提出一种新型的细胞核分割模型,实现了细胞的精确分割。与其他的深度学习方法相比,新模型在面对细胞间存在大量接触、细胞和背景对比度较小的分割实例时,分割的可视化效果更好,准确率更高。但是,由于来自不同医学中心的细胞核数据集中的细胞核形态和大小等差异很大,因此如何设计出更加具有鲁棒性的细胞核分割算法,并提高模型的泛化能力,是未来需要深入研究的问题。
| [1] |
LEE H G, LEE S C. Morphological multi-cell discrimination for robust cell segmentation[J]. IEEE Access, 2020, 8: 49837-49847. DOI:10.1109/ACCESS.2020.2980561 |
| [2] |
DING Z, FU Y. Robust transfer metric learning for image classification[J]. IEEE Transactions on Image Processing, 2017, 26(2): 660-670. DOI:10.1109/TIP.2016.2631887 |
| [3] |
王晓飞, 庞全. 基于圆形约束快速水平集的原生质体细胞分割[J]. 中国图像图形学报, 2013, 18(1): 55-61. |
| [4] |
YU L, SHENG T. Asymmetric GAN for unpaired ima-ge to image translation[J]. IEEE Transactions on Image Processing, 2019, 28(12): 5881-5896. DOI:10.1109/TIP.2019.2922854 |
| [5] |
庞春颖, 刘记奎, 韩立喜. 改进FCM和LFP相结合的白细胞图像分类[J]. 中国图像图形学报, 2013(5): 66-72. |
| [6] |
ZHANG H, ZHU H, LING X. Polar coordinate sampl-ing-based segmentation of overlapping cervical cells using attention U-Net and random walk[J]. Neurocomputing, 2020, 383: 212-223. DOI:10.1016/j.neucom.2019.12.036 |
| [7] |
OYEBODE K O, DU S, VAN WYK B J, et al. Investigating the relevance of graph cut parameter on interactive and automatic cell segmentation[J]. Computational and Mathematical Methods in Medicine, 2018. |
| [8] |
BATENBURG K J, SIJBERS J. Optimal threshold selection for tomogram segmentation by projection distance minimization[J]. IEEE Transactions on Medical Imaging, 2008, 28(5): 676-686. |
| [9] |
龙建武, 申铉京, 臧慧, 等. 高斯尺度空间下估计背景的自适应阈值分割算法[J]. 自动化学报, 2014(8): 1773-1782. |
| [10] |
王娅. 血液红细胞图像自适应标记分水岭分割算法[J]. 中国图像图形学报, 2017, 22(12): 1779-1787. |
| [11] |
谢凤英, 姜志国, 周付根. 基于数学形态学的免疫细胞图象分割[J]. 中国图像图形学报, 2002(11): 18-21. |
| [12] |
李彦冬, 郝宗波, 雷航. 卷积神经网络研究综述[J]. 计算机应用, 2016, 36(9): 2508-2515. |
| [13] |
CHENG G, ZHOU P, HAN J. Duplex metric learning for image set classification[J]. IEEE Transactions on Image Processing, 2018, 27(1): 281-292. DOI:10.1109/TIP.2017.2760512 |
| [14] |
COL A D, VALDIVIA P, PETRONETO F, et al. Wavelet-based visual analysis of dynamic networks[J]. IEEE Transactions on Visualization & Computer Graphics, 2018, 24(8): 2456-2469. |
| [15] |
WANG R, KAMATA S I. Nuclei segmentation of cervical cell images based on intermediate segment qualifier[C]//2018 24th International Conference on Pattern Recognition (ICPR), 2018.
|
| [16] |
LEE H, EUM S, KWON H. ME R-CNN: Multi-expert R-CNN for object detection[J]. IEEE Transactions on Image Processing, 2019, 29: 1030-1044. |
| [17] |
王强, 李孝杰, 陈俊. Supplement卷积神经网络的图像分类方法[J]. 计算机辅助设计与图形学学报, 2018, 23(11): 502-533. |
| [18] |
SUN Y, XUE B, ZHANG M, et al. Completely automated CNN architecture design based on blocks[J]. IEEE Transactions on Neural Networks and Learning Systems, 2020, 31(4): 1242-1254. DOI:10.1109/TNNLS.2019.2919608 |
| [19] |
李宗民, 徐希云, 刘玉杰, 等. 条件随机场像素建模与深度特征融合的目标区域分割算法[J]. 计算机辅助设计与图形学学报, 2018, 30(6): 29-36. |
| [20] |
LI X, DONG Y, PEERS P, et al. Modeling surface appearance from a single photograph using self-augmented convolutional neural networks[J]. ACM Transactions on Graphics, 2017, 36(4): 1-11. |
| [21] |
韩文忠, 康莉, 江静婉, 等. 深度全卷积网络对MRI膀胱图像的分割[J]. 信号处理, 2019, 35(3): 443-450. |
| [22] |
LI S, ZHANG J, RUAN C, et al. Multi-stage attention-U-Net for wireless capsule endoscopy image bleeding area segmentation[C]//2019 IEEE International Conference on Bioinformatics and Biomedicine (BIBM). IEEE, 2019: 818-825.
|
| [23] |
卢飞. 基于区域与深度残差网络的图像语义分割[D]. 赣州: 江西理工大学, 2019.
|
| [24] |
HENRY A, JEFF O, JOHN S, et al. PixelBNN: Augmenting the pixelCNN with batch normalization and the presentation of a fast architecture for retinal vessel segmentation[J]. Journal of Imaging, 2019, 5(2): 26-34. DOI:10.3390/jimaging5020026 |
| [25] |
NIU Y, QIN L, WANG X. Structured graph regularized shape prior and cross-entropy induced active contour model for myocardium segmentation in CTA images[J]. Neurocomputing, 2019, 35(7): 215-230. |



