2. 山东省林草种质资源中心,山东济南 250014
2. Center for Forest and Grass Genetic Resources of Shandong Province, Jinan, Shandong, 250014, China
野生玫瑰(Rosa rugosa)为蔷薇科(Rosaceae)蔷薇属(Rosa)植物,具有重要的生态价值、经济价值和育种价值,在国内主要分布在辽宁、吉林以及山东部分地区。Yang等[1]研究表明,野生玫瑰的濒危是人类活动的结果,大部分野生玫瑰种呈现出生长缓慢、繁殖力低、遗传多样性减少等负面趋势。2021年野生玫瑰被列为国家二级重点保护野生植物[2],珲春市的野生玫瑰种群被划入吉林珲春东北虎国家级自然保护区,山东烟台市的野生玫瑰被划入烟台沿海防护林省级自然保护区[3]。十几年前,长白山和上海建立了3处野生玫瑰基因保存林,但规模较小,收集的异质基因有限[4]。可见,对野生玫瑰的研究与保护亟待深入,并且需要新的思路及多方努力。
迁地保护是收集和保存野生植物资源,尤其是珍稀濒危物种种质资源的重要方式[5],对生境受到严重破坏的物种,有必要采取该方式进行保护,以维护生物多样性和生态平衡。张凯强等[6]对杭州植物园迁地保护的夏蜡梅(Calycanthus chinensis)进行研究,为夏蜡梅这一濒危植物的保护和利用提供科学依据。邓丽丽等[7]对叉叶苏铁(Cycas bifida)进行迁地保护研究,发现迁地保护可有效提高其种群遗传多样性,为其种质资源的保护及管理提供理论指导。可见,迁地保护对于恢复种群遗传多样性、保护濒危野生植物资源具有重要作用。
目前对野生玫瑰的研究主要集中在资源分布[8]、果实[9, 10]、育种[11, 12]和核心种质[13]等方面。本课题组自2019年起开展野生玫瑰迁地保护工作,但未曾对其保护成效进行评估。迁地保护种群是人为建立的小型独立种群,存在采样不全、重复引进、资源不清或种植条件差等问题,因此经过迁地保护的濒危植物同样面临遗传多样性下降或灭绝的危险[14]。为此,本研究以玫瑰野生种群和迁地保存种群为研究对象,采用SSR分子标记技术分析其遗传多样性和遗传结构状况,并对迁地保护的成效进行评估,以期为该物种的有效保护提供科学依据。
1 材料与方法 1.1 材料于2023年4月对分布于辽宁大连、吉林珲春、山东烟台、山东威海4个地区的玫瑰野生种群(DLYS、HCYS、YTYS、WHYS)和迁地保存种群(DLQD、HCQD、YTQD、WHQD)进行采样(表 1),摘取当年生嫩叶作为提取DNA的样品,硅胶干燥保存。
| 种群 Population |
采集地 Collection site |
经度 Longitude |
纬度 Latitude |
数量 Quantity |
| DLYS | Dalian, Liaoning Province | 121°94′N | 40°10′E | 15 |
| HCYS | Hunchun, Jilin Province | 130°34′N | 42°27′E | 15 |
| YTYS | Yantai, Shandong Province | 121°61′N | 37°46′E | 15 |
| WHYS | Weihai, Shandong Province | 122°32′N | 37°40′E | 15 |
| QD | Shandong provincial agricultural high-tech park | 117°15′N | 36°17′E | 60 |
| Note: QD includes 4 ex situ conservation populations, with 15 samples for each population and a total of 60. | ||||
1.2 方法 1.2.1 DNA提取
采用植物基因组DNA提取试剂盒DC104(南京诺唯赞生物科技股份有限公司)提取样品DNA。
1.2.2 反应体系和扩增程序采用优化扩增反应体系:总体积20 μL,其中浓度40 ng/μL的模板DNA 1 μL,浓度10 μmol/L的正、反向引物各1 μL,2×Taq Master Mix酶10 μL,ddH2O 7 μL。扩增程序:95 ℃预变性3 min;95 ℃变性15 s,45-55 ℃退火(依引物不同而异)15 s,72 ℃延伸60 s,35个循环;72 ℃彻底延伸5 min。
1.2.3 扩增产物检测PCR扩增在Biometra Tone 96G型基因扩增仪(德国耶拿分析仪器股份公司)上进行。反应结束后,送往济南天一生物科技有限公司进行高效毛细管电泳检测。
1.2.4 统计分析对8对SSR引物扩增后的产物检测得到的基因型进行统计分析。利用GenAlEx 6.5软件[15]分析各个种群的等位基因数(Na)、有效等位基因数(Ne)、Shannon′s多样性指数(I)、观测杂合度(Ho)、期望杂合度(He)及近交系数(Fis),并根据分析数据进行主成分分析(Principal Component Analysis, PCoA)。利用POPGENE 1.32软件[16]计算8个种群的Nei′s遗传距离及相似系数。利用MEGA 7.0软件[17]基于遗传距离构建非加权组平均法(UPGMA)聚类图。
使用Structure 2.3软件[18]对8个玫瑰种群进行分析,并根据遗传成分差异分析种群遗传结构。遗传学组数(K)设置为1-8,每个K值分别运行10次。运行结果代入公式m(|L(K+1)-2L(K)+L(K-1)|)/s[L(K)]计算ΔK值,分析出最佳K值。公式中m代表求平均值,L(K)代表运行结果中返回的第K次运行的LnP(D)值,s代表标准差。
2 结果与分析 2.1 种群遗传多样性基于8对SSR引物对120个玫瑰个体进行SSR分析,共扩增出29条条带,预期产物长度为89-187 bp。平均每对引物扩增出的条带数为3.6条(表 2)。
| 引物编号 Primer No. |
引物序列 Primer sequence |
荧光类型 Fluorescence type |
预期产物长度/bp Expected product length/bp |
退火温度/℃ Annealing temperature/℃ |
扩增条带数 Number of amplified bands |
| Ro02 | GAGAAAATCAAGTCGCC CTCCAAACACTCCAGCA | FAM | 93 | 56 | 2 |
| Ro04 | CTTCACCTTCTTGGGAG TGGCTTGTTTGGCTATC | FAM | 89 | 55 | 3 |
| Ro15 | GTGCCCAACTCCATCGT GCTCACAGAAAACCGCC | TAMRA | 133 | 54 | 2 |
| Ro28 | GCTATTTCATCCCAACA AAGATACTCCATTCCCC | HEX | 187 | 54 | 2 |
| Ro30 | CACTGAAGCAGAAGGAA GGGAGGTGGAAGTAATC | HEX | 145 | 55 | 2 |
| SSR9 | ATTATTCAAGAAATCGATGCAGG TGTGGTGATTAACAGCATTGTTC | HEX | 149 | 57 | 8 |
| SSR14 | GTCTCTGCTGCATTCTTTCTCC ATCTCTATATGACCGATTCGCCT | TAMRA | 140 | 58 | 3 |
| SSR17 | ATATTGGACTCAACACCAGCAAT AATGGCAGATTACGAGAGATTTG | FAM | 158 | 57 | 7 |
| Note: Primers Ro02, Ro04, Ro15, Ro28 and Ro30 are designed according to the previous research of the research group; primers SSR9, SSR14 and SSR17 refer to the primers developed by Xia[19]. | |||||
遗传多样性是保护生物学研究的核心之一,对珍稀濒危物种保护方针和措施的制定均基于对物种遗传多样性的研究。由GenAlEx 6.5软件分析得知,各种群的Na为2.000-2.500,平均值为2.234;Ne为1.441 5-2.062 4,平均值为1.698 8;I平均值为0.489 9,DLQD种群最高(0.628 8),HCYS种群最低(0.392 4);Ho为0.089 3-0.150 0,平均值为0.122 7;He为0.226 0-0.359 8,平均值为0.283 5(表 3)。整体来看,HCYS的各项指标最低,即遗传多样性水平最低,DLQD的各项指标最高;辽宁大连与吉林珲春迁地保存种群遗传多样性水平分别高于其对应的野生种群。
| 种群 Population |
等位基因数 Na | 有效等位基因数 Ne | Shannon′s多样性指数 I | 观测杂合度 Ho | 期望杂合度 He | 近交系数 Fis |
| DLYS | 2.500 | 1.895 0 | 0.571 9 | 0.112 8 | 0.313 4 | 0.640 1 |
| DLQD | 2.500 | 2.062 4 | 0.628 8 | 0.089 3 | 0.359 8 | 0.751 8 |
| HCYS | 2.125 | 1.441 5 | 0.392 4 | 0.100 0 | 0.226 0 | 0.557 5 |
| HCQD | 2.375 | 1.647 8 | 0.491 5 | 0.144 0 | 0.283 4 | 0.491 9 |
| YTYS | 2.000 | 1.668 9 | 0.475 1 | 0.150 0 | 0.293 1 | 0.488 2 |
| YTQD | 2.250 | 1.629 3 | 0.463 7 | 0.150 0 | 0.258 6 | 0.419 9 |
| WHYS | 2.125 | 1.744 9 | 0.494 0 | 0.116 7 | 0.288 8 | 0.595 9 |
| WHQD | 2.000 | 1.500 6 | 0.401 5 | 0.118 5 | 0.244 8 | 0.515 9 |
| Average | 2.234 | 1.698 8 | 0.489 9 | 0.122 7 | 0.283 5 | 0.557 7 |
将野生种群与迁地保存种群分别合并,合并后的遗传多样性及结构分析结果如表 4所示。迁地保存种群等位基因数略高于野生种群,野生种群的I、Ho、He分别为0.715 7、0.120 4、0.386 5,迁地保存种群的I、Ho、He分别为0.752 3、0.127 2、0.409 3。综合分析发现,迁地保存种群的遗传多样性水平高于野生种群。野生种群与迁地保存种群的近交系数均为正值且数值较高,表明种群杂合子缺失,纯合子过剩,存在一定程度的自交或近交。
| 种群 Population |
等位基因数 Na |
Shannon′s多样性指数 I | 观测杂合度 Ho | 期望杂合度 He | 近交系数 Fis | 总遗传变异 Ht |
种群内遗传变异 Hs | 种群间遗传分化系数 Fst |
| Wild population | 3.375 | 0.715 7 | 0.120 4 | 0.386 5 | 0.688 4 | 0.383 4 | 0.280 3 | 0.268 9 |
| Ex situ conser-vation population | 3.475 | 0.752 3 | 0.127 2 | 0.409 3 | 0.689 2 | 0.410 9 | 0.286 7 | 0.302 3 |
| Total value | 3.625 | 0.742 7 | 0.123 6 | 0.400 3 | 0.691 2 | 0.399 7 | 0.283 5 | 0.290 7 |
野生种群的总遗传变异(Ht)为0.383 4,种群内遗传变异(Hs)为0.280 3,种群间遗传变异为0.103 1,种群间遗传分化系数(Fst)为0.268 9。迁地保存种群的Ht为0.410 9,Hs为0.286 7,种群间遗传变异为0.124 2,Fst为0.302 3。
野生种群和迁地保存种群的Ht为0.399 7,Hs为0.283 5,种群间遗传变异为0.116 2,Fst为0.290 7。在整体遗传多样性中,遗传变异主要存在于种群内:29.07%的遗传变异存在于种群间,70.93%的遗传变异存在于种群内。
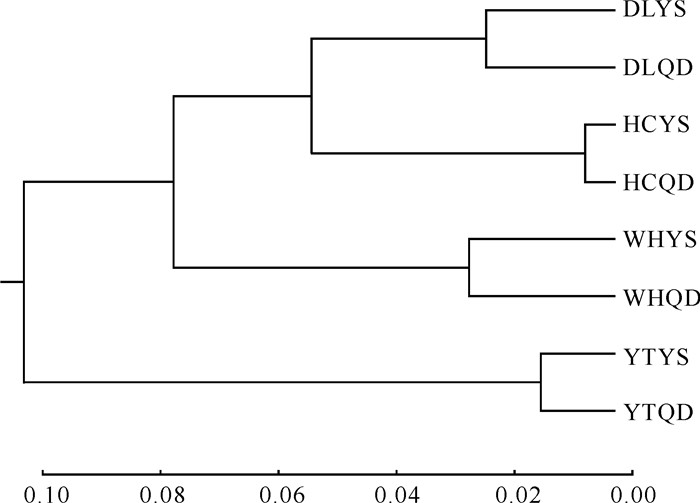
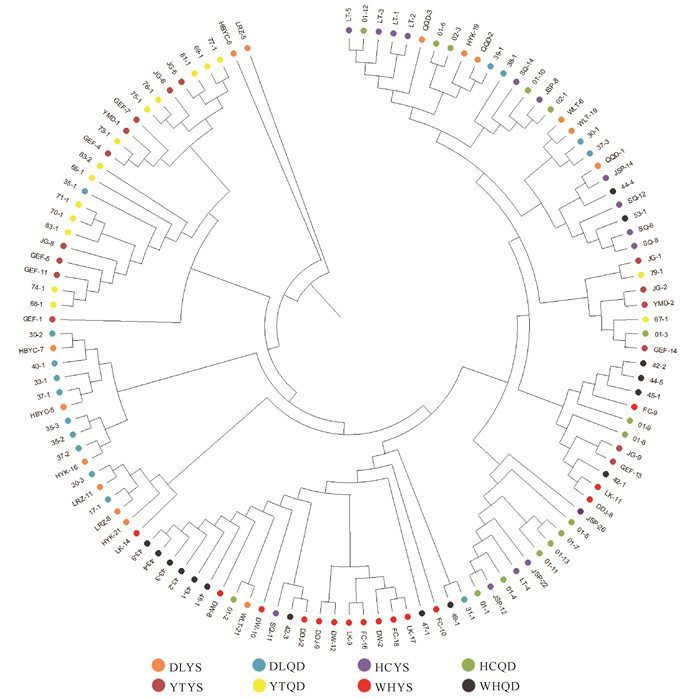
2.2 遗传结构由POPGENE 1.32软件计算得到8个玫瑰种群间的Nei′s遗传距离及相似系数(表 5)。由此分析可知,8个种群间的Nei′s遗传距离为0.013 0-0.386 6,变幅为0.373 6;遗传相似系数为0.679 4-0.987 1,变幅为0.307 7。其中,遗传距离最近的两个种群为HCYS与HCQD;遗传距离最远的两个种群为YTQD与WHQD。将分析出的遗传距离数据导入MEGA 7.0软件,构建8个玫瑰种群聚类图(图 1)以及120个玫瑰个体的遗传聚类图(图 2)。同一地区的野生种群与迁地保存种群均聚为一类;山东烟台地区的种群单独聚为一类,与其他地区种群有所区分。个体聚类与种群聚类相一致,但表现出来自不同群体的个体出现个体混合聚类模式,代表着群体间缺乏强遗传分化。
| 种群 Population |
DLYS | DLQD | HCYS | HCQD | YTYS | YTQD | WHYS | WHQD |
| DLYS | 0.941 6 | 0.917 7 | 0.901 2 | 0.766 7 | 0.711 8 | 0.778 3 | 0.866 1 | |
| DLQD | 0.060 2 | 0.836 2 | 0.819 8 | 0.783 2 | 0.739 7 | 0.765 0 | 0.817 0 | |
| HCYS | 0.085 9 | 0.178 9 | 0.987 1 | 0.800 7 | 0.714 8 | 0.796 7 | 0.878 3 | |
| HCQD | 0.104 0 | 0.198 7 | 0.013 0 | 0.797 6 | 0.702 8 | 0.791 6 | 0.859 4 | |
| YTYS | 0.265 6 | 0.244 4 | 0.222 3 | 0.226 1 | 0.963 3 | 0.794 2 | 0.738 2 | |
| YTQD | 0.339 9 | 0.301 5 | 0.335 8 | 0.352 7 | 0.037 4 | 0.739 6 | 0.679 4 | |
| WHYS | 0.250 6 | 0.267 8 | 0.227 3 | 0.233 7 | 0.230 4 | 0.301 6 | 0.943 1 | |
| WHQD | 0.143 8 | 0.202 1 | 0.129 8 | 0.151 5 | 0.303 5 | 0.386 6 | 0.058 6 |
|
| 图 1 基于种群遗传距离的聚类图 Fig.1 Clustering map based on population genetic distance |
|
| 图 2 基于个体遗传距离的遗传聚类图 Fig.2 Genetic clustering map based on individual genetic distance |
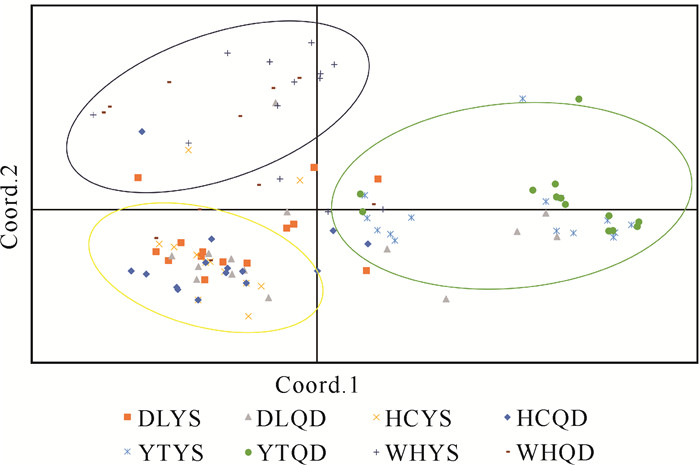
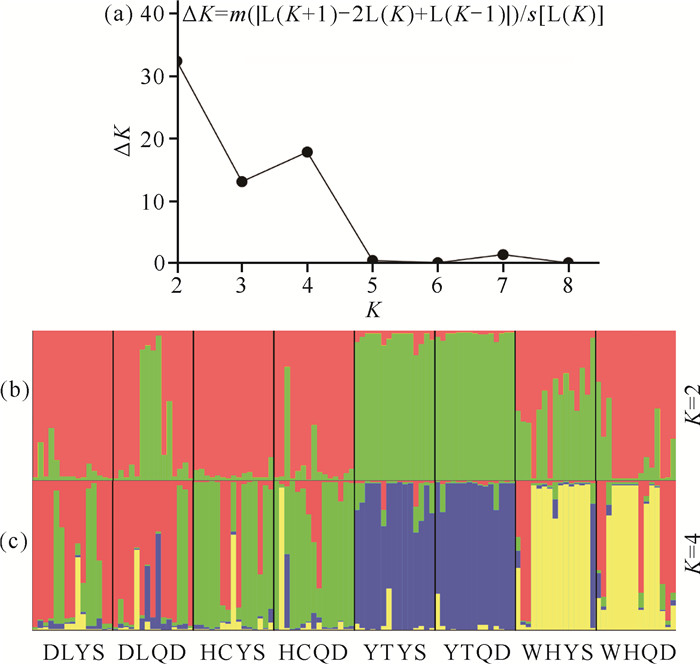
PCoA结果见图 3。烟台种群相对独立,大连种群与珲春种群遗传关系较为相近。Coord.1和Coord.2分别代表 19.91%和12.77%的总变异。Structure分析结果显示,8个玫瑰种群120个玫瑰个体最佳遗传学组数K=2[图 4(a)],此时烟台种群大部分个体分为一组,其余种群大部分个体分为一组[图 4(b)]。当K=4时,聚类情况与野生玫瑰分布地位置基本一致,出现少量离群样本[图 4(c)]。珲春种群、烟台种群和威海种群相对独立,仅有部分个体与其他地区个体进行过基因交流;珲春种群中则存在较多与外地个体交流的个体。PCoA和Structure分析8个玫瑰种群遗传结构的结果基本一致。
|
| 图 3 主成分分析结果 Fig.3 Analysis result of principal component |
|
| (a) The relationship between the number of undetermined groups K and the estimated value ΔK; (b) Population Structure analysis when K=2;(c) Population Structure analysis when K=4. 图 4 Structure分析结果 Fig.4 Structure analysis result |
3 讨论 3.1 玫瑰种群的遗传多样性
遗传多样性水平是决定种群适应环境能力和进化潜力的重要因素之一[20]。通常,珍稀濒危植物中孤立且数量较小的种群,其遗传多样性水平较低[21, 22]。物种水平上,野生玫瑰的遗传多样性水平较低(Na、I、He分别为2.234、0.489 9、0.283 5)。野生种群中,DLYS种群的遗传多样性最高,HCYS种群最低;迁地保存种群中,DLQD种群的遗传多样性最高,WHQD种群最低。由于种群间的遗传多样性不平衡,部分种群较低的遗传多样性使得整个种群的遗传多样性水平降低。将野生种群与迁地保存种群分别合并分析,结果显示迁地保存种群的遗传多样性高于野生种群。各种群观测杂合度均小于期望杂合度,说明杂合子缺失(Fis>0),群体间亲缘关系较近,存在一定程度的近交,生境片断化的遗传效应正逐渐显现[23]。
影响物种遗传多样性的因素有多种,如人为破坏、地理分布和繁殖方式等[24]。野生玫瑰在我国的分布地比较狭窄,近年来由于农田开垦、经济开发等人为原因破坏严重,种群在逐渐缩小和碎片化。同时,玫瑰种子发育存在缺陷,未经人工干预时繁殖能力低,多数通过地下根茎繁殖产生新个体[25],这可能是导致其野生种群遗传多样性水平较低的原因。玫瑰在野外通常依靠蜜蜂、蝴蝶等昆虫进行异花授粉,种群间或种群内个体存在频繁的基因交流,因此种子会拥有更高的遗传多样性[26]。本研究中,迁地保存种群为野生玫瑰的种播苗,这可能是迁地保存种群遗传多样性水平高于野生种群的原因。
3.2 玫瑰种群的遗传结构与分化植物种群的遗传构成是其在长时间演化过程中,物种的进化历史、分布范围的变化、栖息地的破碎化与种群的适应性变化、繁殖机制、基因交流、自然选择等因素相互作用的结果[27]。从Nei′s遗传距离与相似系数来看,8个玫瑰种群的遗传分化并不明显,遗传相似系数变幅为0.307 7。分化最大的两个种群YTQD与WHQD,遗传距离和相似系数分别为0.386 6与0.679 4;分化最小的两个种群HCYS与HCQD,遗传距离和相似系数分别为0.013 0与0.987 1。
PCoA和Structure分析结果相似,取样种群最佳遗传学组数K=2,即烟台地区与其他种群存在较大差异,可单独分为一组,其余种群为另一组,说明烟台种群与其他地区种群有明显分化。威海种群较为接近烟台种群,由于威海与烟台地理位置接近,推测两地区种群间存在更为频繁的基因交流,以水为媒介进行繁殖的可能性较大[28]。DLQD种群中,部分个体与其他地区个体发生过基因交流,说明对该地区的保护初见成效,遗传多样性趋于丰富。
就某一个物种来说,遗传多样性程度与其适应环境的能力直接关联[29],遗传变异水平关系到种群的适应能力乃至决定着整个种的进化与生存[20, 30]。本研究中,玫瑰野生种群的Ht、Hs、Fst分别为0.383 4、0.280 3、0.268 9,迁地保存种群的Ht、Hs、Fst分别为0.410 9、0.286 7、0.302 3,野生种群和迁地保存种群遗传变异主要存在于种群内,该结果与Xu等[31]的研究结果一致。在其他植物如广西火桐[32](Erythropsis kwangsiensis)和南方红豆杉[33](Taxus wallichiana)的迁地保护中,也出现遗传变异主要存在于种群内而种群间基因流动频率较低的现象,这可能是由于自然种群地理位置相距过大,从而限制了种群间的基因交流,增强了居群内的遗传分化[34, 35]。
4 结束语本研究采用SSR分子标记技术,分析玫瑰野生种群、迁地保存种群的遗传多样性和遗传结构状况,并对迁地保护的成效进行评估,发现野生玫瑰具有较低的遗传多样性和种群间遗传分化水平。这种遗传结构的同质性和分化不明显可能会威胁到物种的长期生存和进化潜能,而迁地保护对野生玫瑰遗传多样性恢复有较大作用。该结果可为后续野生玫瑰保护策略的制定提供科学依据,同时对其他野生植物的保护也有借鉴意义。
综合本课题组对野生玫瑰迁地保护工作的开展情况以及对其遗传多样性的分析,给出以下3个建议:首先,加强自然栖息地的保护,包括建立和升级自然保护区,以及执行严格的管理策略以维护其自然生态系统的完整性。其次,推进迁地保护工作,通过人工繁育项目来增加野生玫瑰的种群数量,同时注意保持物种的遗传多样性。最后,通过分子生物学手段深入研究野生玫瑰的生活习性、繁殖机制和生态需求,以便更精确地制定和实施保护策略。
| [1] |
YANG J H, ZHANG S P, LIU J, et al. Genetic diversity of the endangered species Rosa rugosa Thunb.in China and implications for conservation strategies[J]. Journal of Systematics and Evolution, 2009, 47(6): 515-524. DOI:10.1111/j.1759-6831.2009.00049.x |
| [2] |
靳亚琦. 不同种源玫瑰(Rosa rugosa)群体开花相关基因的遗传分化研究[D]. 济南: 山东大学, 2021.
|
| [3] |
金飞宇, 束华杰, 刘建, 等. 玫瑰种群生物学研究进展[J]. 生态学报, 2016, 36(11): 3156-3166. |
| [4] |
陈建军, 刘毅, 吴景才, 等. 野生玫瑰濒危机理的研究[J]. 吉林林业科技, 2008, 37(2): 1-6. DOI:10.3969/j.issn.1005-7129.2008.02.001 |
| [5] |
PRIMACK R B, 马克平, 蒋志刚. 保护生物学[M]. 北京: 科学出版社, 2014.
|
| [6] |
张凯强, 王浥尘, 陆婷, 等. 迁地保护区珍稀濒危植物夏蜡梅的保护研究[J]. 陕西农业科学, 2023, 69(4): 71-74. DOI:10.3969/j.issn.0488-5368.2023.04.018 |
| [7] |
邓丽丽, 唐健民, 邹蓉, 等. 广西崇左叉叶苏铁野生和迁地保护种群的遗传多样性比较分析[J]. 广西科学, 2023, 30(6): 1101-1109. |
| [8] |
夏德美, 姚洪锡. 野生玫瑰种质资源分布现状及预测[J]. 杨凌职业技术学院学报, 2023, 22(4): 13-15. |
| [9] |
周秀娟. 野生玫瑰两个品系果实品质特性的比较分析与评价[D]. 延吉: 延边大学, 2017.
|
| [10] |
何雨, 董画, 薛桂新. 野生玫瑰果类胡萝卜素含量变化及其色泽相关性分析[J]. 食品科技, 2018, 43(9): 95-99. |
| [11] |
侯秋梅, 周洪英. 玫瑰种质资源及杂交育种研究现状[J]. 贵州农业科学, 2022, 50(1): 14-22. DOI:10.3969/j.issn.1001-3601.2022.01.003 |
| [12] |
冯立国. 玫瑰野生种质资源评价及其与栽培种质亲缘关系的研究[D]. 泰安: 山东农业大学, 2007.
|
| [13] |
姜丽媛. 濒危植物野生玫瑰种质资源评价与核心种质构建[D]. 泰安: 山东农业大学, 2018.
|
| [14] |
ZHU X L, ZOU R, QIN H Z, et al. Genome-wide diversity evaluation and core germplasm extraction in ex situ conservation: a case of golden Camellia tunghinensis[J]. Evolutionary Applications, 2023, 16(9): 1519-1530. DOI:10.1111/eva.13584 |
| [15] |
PEAKALL R, SMOUSE P E. GenAlEx 6.5:genetic analysis in Excel.Population genetic software for teaching and research: an update[J]. Bioinformatics, 2012, 28(19): 2537-2539. DOI:10.1093/bioinformatics/bts460 |
| [16] |
YEH F, YANG R C, BOYLE T, et al. POPGENE, version 1.32:the user friendly software for population genetic analysis[J]. Computer Science, Biology, 1999, 64471650. |
| [17] |
UMAR S, STECHER G, TAMURA K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [18] |
PRITCHARD J K, STEPHENS M, DONNELLY P. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155(2): 945-959. DOI:10.1093/genetics/155.2.945 |
| [19] |
夏峰梅. 基于转录组信息的玫瑰SSR标记开发及野生玫瑰遗传多样性分析[D]. 泰安: 山东农业大学, 2016.
|
| [20] |
FRANKHAM R, BALLOU J D, BRISCOE D A. Introduction to conservation genetics[M]. Cambridge: Cambridge University Press, 2002: 182-183.
|
| [21] |
GAO Q B, LI Y, GENGJI Z M, et al. Population genetic differentiation and taxonomy of three closely related species of Saxifraga (Saxifragaceae) from southern Tibet and the Hengduan Mountains[J]. Frontiers in Plant Science, 2017, 8: 1325. DOI:10.3389/fpls.2017.01325 |
| [22] |
SPIELMAN D, BROOK B W, FRANKHAM R. Most species are not driven to extinction before genetic factors impact them[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(42): 15261-15264. |
| [23] |
曹毓蓉, 马永鹏, 张秀姣, 等. 基于SNP分子标记的波叶杜鹃遗传特征分析[J]. 云南大学学报(自然科学版), 2022, 44(4): 859-869. |
| [24] |
NYBOM H. Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants[J]. Molecular Ecology, 2004, 13(5): 1143-1155. DOI:10.1111/j.1365-294X.2004.02141.x |
| [25] |
陈建军, 刘毅, 吴景才, 等. 野生玫瑰濒危机理的研究[J]. 吉林林业科技, 2008, 37(2): 1-6. DOI:10.3969/j.issn.1005-7129.2008.02.001 |
| [26] |
杨鹏, 何志, 胡军和, 等. 种子传播提高生物多样性的机制[J]. 植物保护学报, 2023, 50(5): 1244-1253. |
| [27] |
SCHAAL B A, HAYWORTH D A, OLSEN K M, et al. Phylogeographic studies in plants: problems and prospects[J]. Molecular Ecology, 1998, 7(4): 465-474. DOI:10.1046/j.1365-294x.1998.00318.x |
| [28] |
王晶晶. 基于全基因组重测序的玫瑰群体结构及遗传多样性分析[D]. 北京: 中国农业科学院, 2023.
|
| [29] |
HAMRICK J L, GODT M W. Effects of life history traits on genetic diversity in plant species[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 1996, 351(1345): 1291-1298. |
| [30] |
STEFENON V M, GAILING O, FINKELDEY R. Genetic structure of plantations and the conservation of genetic resources of Brazilian pine (Araucaria angustifolia)[J]. Forest Ecology and Management, 2008, 255(7): 2718-2725. |
| [31] |
XU J J, ZANG F Q, WU Q C, et al. Analysis of the genetic diversity and molecular phylogeography of the endangered wild rose (Rosa rugosa) in China based on chloroplast genes[J]. Global Ecology and Conservation, 2021, 28: e01653. |
| [32] |
骆文华, 代文娟, 刘建, 等. 广西火桐自然种群和迁地保护种群的遗传多样性比较[J]. 中南林业科技大学学报, 2015, 35(2): 66-71. |
| [33] |
李乃伟, 贺善安, 束晓春, 等. 基于ISSR标记的南方红豆杉野生种群和迁地保护种群的遗传多样性和遗传结构分析[J]. 植物资源与环境学报, 2011, 20(1): 25-30. |
| [34] |
文菁, 范嗣刚, 李海鹏, 等. 南海4个花刺参地理群体的遗传多样性研究[J]. 水产科学, 2018, 37(3): 404-408. |
| [35] |
庞兴宸, 陈景锋, 孙芝倩, 等. 广州兰圃公园兰花种质资源迁地保护[J]. 中国野生植物资源, 2020, 39(9): 80-90. |



