随着年龄的增长,人的认知能力也会不断退化甚至引发阿尔茨海默病等认知功能性疾病,此过程亦称为认知衰老。此类认知衰老导致的认知障碍(Cognitive Impairment,CI)不仅给老年人的生活带来不便,而且产生较高的护理成本,增加家庭和社会的负担[1]。在认知衰老过程中,嗅觉扮演着重要的角色。人类嗅觉功能随年龄增长而逐渐下降[2],嗅觉阈值升高[3]。中国60岁以上老年人嗅觉障碍患病率超过60%[4]。嗅觉皮层与海马体、杏仁体、前额叶等认知相关区域密切连接,这些区域在处理嗅觉信息时也参与了记忆、情感与执行功能等生理过程的调节[5-6]。因此,在衰老过程中,嗅觉皮层和海马体等结构退化可能是嗅觉障碍与CI相互关联的基础。已有研究发现嗅觉障碍与痴呆风险呈正相关,并可作为阿尔茨海默病的早期标志[7-8]。因此,通过对嗅觉障碍相关基因的筛查,期望揭示嗅觉障碍的分子通路,并通过对关键靶点的早期干预,预防或延缓认知衰老,从而提高老年人的生活质量。
嗅觉损害和CI都是复杂表型,均受遗传因素和环境因素共同影响[9-10]。前期研究发现,广西红水河流域长寿老人不仅规避了常见的衰老相关性疾病,同时还保持了良好的嗅觉及认知功能,故长寿亦称“成功衰老”或“健康衰老”[11]。由此推测,红水河流域长寿老人能够实现健康衰老,除优越的自然环境和健康的生活方式外,可能还与嗅觉和认知方面的遗传学保护优势密切相关。长寿人群在高龄状态依旧保持了较为良好的认知功能,减少了阿尔茨海默病等认知功能性疾病的发生,为此,本研究对该流域长寿老人嗅觉相关基因OR7D4、OR7E24、OR9Q1和SNCA的7个单核苷酸多态性(Single Nucleotide Polymorphism, SNP)进行检测,包括OR7D4 rs61729907、OR7D4 rs8100858、OR7E24 rs2240927、OR7E24 rs12608694、OR9Q1 rs2245676、OR9Q1 rs921134及SNCA rs894278,以探讨与嗅觉相关的SNP-SNP之间及基因-环境之间的交互作用对长寿人群认知功能的影响,从而进一步了解长寿群体认知衰老的遗传学优势。
1 材料与方法 1.1 研究对象随机选取广西红水河流域壮族老人905人,分为3组:长寿组(Long-lived Group,LG)283例,年龄≥90岁,平均年龄(93.46±3.46)岁;子女组(Offspring Group,OG)296例,为长寿老人第一代子女,年龄60-79岁,平均年龄(68.46±4.84)岁;当地组(Control Group,CG)326例,家系中无长寿史,年龄60-79岁,平均年龄(69.21±5.50)岁;另设一个外地(贺州)组(Hezhou group,HZ),为贺州市八步区南乡镇的普通壮族老人286例,家系中无长寿史,年龄60-79岁,平均年龄(68.95±5.63)岁。所有受检者均通过问诊和体格检查,排除阿尔茨海默病、帕金森病等认知疾病,以及肝、肾功能障碍,中风,高血压,糖尿病等衰老相关性疾病,且均已签署知情同意书。本研究由广西医科大学伦理委员会批准,批号:20200036。
1.2 方法 1.2.1 样本的采集受试者在空腹12 h后,于次日清晨坐位抽取外周静脉血8 mL,其中6 mL分离血清,用于检测各种血液生化学指标;另外2 mL置于抗凝管中并于-20 ℃中保存,用于抽提全基因组DNA并进行后续的SNP分型检测。采用微量血糖仪(罗氏ACCU-CHEK Active)检测空腹血糖(Fasting Blood Glucose,FBG)。采用标准酶法测定总胆固醇(Total Cholesterol,TC)和甘油三酯(Triglyceride,TG)的水平,高密度脂蛋白胆固醇(High-Density Lipoprotein Cholesterol,HDL-C)和低密度脂蛋白胆固醇(Low-Density Lipoprotein Cholesterol,LDL-C)等则采用酶联免疫一步法测定。全部测定均在广西壮族自治区江滨医院检验科进行。
采用简易精神状态检查(Mini-Mental State Examination, MMSE)量表来进行认知测评。其中时间定向(5个小题)、地点定向(5个小题)、表达(2个小题)、注意力和计算力(5个小题)、记忆力(3个小题)、语言(10个小题)。每个问题1分,答对得1分,答错得0分。MMSE总分30分。根据受教育程度及MMSE分值来划分不同亚组:MMSE≤17分的未受教育者、MMSE≤20分的受小学教育者、MMSE≤24分的受中学及以上教育者纳入CI亚组;其他高于对应分界值者归入认知正常(Cognitive Normal, CN)亚组[12]。
采用嗅觉识别测试(Sniffin Sticks Identification Test,SSIT)中的气味识别阈试验进行嗅觉检测,测试使用16支含有不同气味的嗅棒进行,受试者需根据所给的显示4种气味的图像和名称的卡片找出正确的气味,答对得1分,全部选择正确得16分。
1.2.2 标签SNP的筛选及基因分型通过查阅全基因组关联研究(Genome-Wide Association Studies,GWAS)数据库和近期嗅觉相关SNP文献报道,并从在线资源1000 Genomes Project数据库(https://www.internationalgenome.org/1000-genomes-browsers/index.html)下载基因片段上的SNP信息。利用Haploview 4.2软件的Tagger程序挑选出目的基因上的标签SNP (TagSNP),选出与嗅觉存在相关性且最小等位基因频率(Minor Allele Frequency,MAF)>1%的SNP作为研究位点,最终选取7个SNP,其相关信息如表 1所示。SNP基因分型采用改进的多重高温连接酶检测反应(improved Multiple Ligase Detection Reaction, iMLDR)方法进行,具体方法见Huang等[13]的相关研究。
| 单核苷酸多态性 SNP |
染色体位置 Chromosome position |
基因区域 Region of gene |
等位基因 Allele |
最小等位基因频率 MAF |
| OR7D4 rs61729907 | 19:9214576 | Nonsynon_exo1 | G>A | A=0.199 5 |
| OR7D4 rs8100858 | 19:9216221 | 5′-flanking | C>T | T=0.055 2 |
| OR7E24 rs12608694 | 19:9250626 | 5′-UTR_exon1 | T>C | C=0.382 2 |
| OR7E24 rs2240927 | 19:9251667 | Synon_exon1 | C>T | T=0.439 9 |
| OR9Q1 rs921134 | 11:58027924 | Intron | A>G | G=0.312 5 |
| OR9Q1 rs2245676 | 11:58145221 | Intron | T>G | G=0.305 2 |
| SNCA rs894278 | 4:89813384 | Intron | T>G | G=0.341 3 |
1.2.3 统计学分析
应用IBM SPSS Statistics 26.0软件对上述数据进行统计分析。计量资料用平均数(mean)±标准差(standard)来表示,组间比较采用协方差分析(ANCOVA)。分类变量以频数表示,组间比较采用卡方检验,用χ2表示。
利用卡方检验比较SNP基因型及其等位基因在各组间的分布差异。在共显性、显性、隐性和等位基因4种遗传模型下,采用非条件性Logistic回归分析评估各位点基因型与CI的关联情况。对性别、年龄、体重指数(BMI)、收缩压(SBP)、甘油三酯、低密度脂蛋白胆固醇等协变量进行校正,将混杂因素的影响分离出来,使分析结果能更真实地反映自变量与因变量之间的关系。
采用Haploview 4.2软件计算各SNP基因型的哈迪-温伯格平衡(Hardy-Weinberg Equilibrium,HWE),分析各位点间的连锁不平衡状态,构建单倍型以了解基因SNP之间的内在交互作用。两位点之间的连锁不平衡状态用D′表示,当D′>0.8表明两位点处于强连锁不平衡状态,0.8≥D′>0.5表明两位点处于连锁不平衡状态,而D′≤0.5则说明两位点之间不存在连锁不平衡。基于GMDR 0.9软件,用广义多因子降维分析方法(Generalized Multifactor Dimensionality Reduction, GMDR)分析各位点与各环境因素之间的关系。基于MDR 0.7软件,用多因子降维分析方法构建各位点与环境因素之间的交互作用树状图。由于CI为多病因性疾病,同时受多种环境因素的影响,因此纳入收缩压、舒张压、空腹血糖、体重指数、总胆固醇、甘油三酯、高密度脂蛋白胆固醇、低密度脂蛋白胆固醇、嗅觉功能、腰臀比等环境因素,进一步研究SNP-SNP和基因-环境交互作用对CI的影响。
2 结果与分析 2.1 一般情况及认知水平的比较通过比较长寿组、子女组、当地组以及贺州组的一般临床资料发现,长寿组的身高、体重、体重指数、甘油三酯皆明显低于当地组和贺州组(P<0.05),一定程度上说明长寿老人保持较好的血脂水平;子女组、当地组和贺州组3组之间的年龄无差异(P>0.05),但子女组的身高、体重、甘油三酯皆高于当地组和贺州组,其中身高体重的差异可能与子女组男性占比明显高于当地组和贺州组有关。贺州组的低密度脂蛋白胆固醇明显高于其他3组,提示贺州组有相对较高的心脑血管疾病风险(表 2)。
| 指标 Indicator |
长寿组 LG (n=283) |
子女组 OG (n=296) |
当地组 CG (n=326) |
贺州组 HZ (n=286) |
F(x2) | P |
| Ratio of male to female | 77/206 | 222/74* | 185/141*# | 122/164*#☆ | 144.583 | 0.000 |
| Age/years | 93.46±3.46 | 68.46±4.84* | 69.21±5.50* | 68.95±5.63* | 176.344 | 0.000 |
| SBP/mmHg | 145.24±18.30 | 141.57±17.82* | 147.20±19.43*# | 139.33±19.77☆ | 7.581 | 0.000 |
| DBP/mmHg | 79.83±14.40 | 80.07±11.22 | 81.33±13.89 | 79.28±12.05 | 0.531 | 0.661 |
| PP/mmHg | 65.41±15.96 | 61.50±13.38* | 65.87±15.66*# | 60.05±14.84*☆ | 9.778 | 0.000 |
| FBG/(mol/L) | 5.40±1.46 | 5.49±1.63 | 5.61±0.93 | 5.58±0.81 | 0.799 | 0.494 |
| Height/cm | 143.53±9.38 | 156.62±8.05* | 152.66±8.41*# | 151.80±7.56*# | 25.001 | 0.000 |
| Weight/kg | 39.09±8.07 | 52.60±9.14* | 51.05±9.03*# | 47.19±7.89*#☆ | 24.973 | 0.000 |
| BMI/(kg/m2) | 18.99±3.57 | 21.51±3.34* | 21.73±3.04* | 20.44±2.80*#☆ | 15.415 | 0.000 |
| Waist circumference/cm | 70.07±9.42 | 72.56±8.36 | 73.28±7.80 | 69.63±8.56# | 1.579 | 0.193 |
| Hip circumference/cm | 79.61±6.73 | 84.45±5.55 | 86.18±5.74*# | 83.33 ± 5.24☆ | 6.622 | 0.000 |
| Creatinine/(μmol/L) | 84.47±28.22 | 83.24±37.43 | 75.51±18.13* | 69.93±16.09*☆ | 11.967 | 0.000 |
| TC/(mmol/L) | 4.79±0.94 | 5.00±0.90 | 4.99±0.96 | 5.31±0.99*#☆ | 10.862 | 0.000 |
| TG/(mmol/L) | 1.16±0.54 | 1.44±1.42* | 1.18±0.70# | 1.12±0.83# | 6.570 | 0.000 |
| HDL-C/(mmol/L) | 1.39±0.34 | 1.49±0.37 | 1.51±0.43 | 1.52±0.34 | 0.598 | 0.616 |
| LDL-C/(mmol/L) | 2.94±0.82 | 3.00±0.88 | 2.95±0.82 | 3.31±0.87*#☆ | 14.126 | 0.000 |
| Note: except for gender, all other data are presented as mean±standard.Statistical significance (P<0.05) compared with the LG is indicated by *, compared with the OG by #, and compared with the CG by ☆. | ||||||
各组认知功能测试和嗅觉功能测试结果如表 3所示。由于长寿组年龄较大、受教育程度较低,其MMSE评分(18.25±3.28)和嗅觉评分(8.40±2.19)明显低于其他3组(P<0.05)。子女组、当地组和贺州组年龄无明显差异,且受教育程度大体相近,因此子女组、当地组和贺州组之间的比较可以消除年龄及受教育程度影响,子女组的MMSE评分和嗅觉评分皆高于当地组和贺州组(P<0.05);当地组的MMSE评分和嗅觉评分高于贺州组(P < 0.05)。
| 项目 Item |
认知功能测试 Cognitive function test |
嗅觉功能测试 Olfactory function test |
|||||||
| 时间定向 Time orientation |
位置定向 Location orientation |
表达 Registration |
注意力和计算力 Attention and calculation |
记忆力 Recall |
语言 Language |
总计 Total |
嗅觉 Olfactory |
||
| LG(n=283) | 2.77±0.85 | 3.70±1.07 | 2.90±0.41 | 1.45±1.15 | 1.42±1.19 | 6.01±0.84 | 18.25±3.28 | 8.40±2.19 | |
| OG(n=296) | 3.86±1.09 | 4.93±0.29* | 2.99±0.10* | 3.60±1.57* | 2.70±0.60* | 7.46±1.04* | 25.54±3.10* | 11.21±1.97* | |
| CG(n=326) | 3.69±1.33 | 4.73±0.61*# | 3.01±0.11* | 2.78±1.70# | 2.71±0.74* | 6.63±0.87# | 23.54±3.71*# | 9.82±2.39*# | |
| HZ(n=286) | 3.24±1.58#☆ | 4.44±1.00#☆ | 3.00±0.00* | 2.70±1.94# | 2.67±0.73* | 6.75±089# | 22.81±4.50#☆ | 9.12±2.78*#☆ | |
| F(x2) | 11.379 | 20.035 | 2.744 | 18.298 | 9.935 | 48.417 | 30.367 | 42.002 | |
| P | 0.000 | 0.000 | 0.042 | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | |
| Note: all data are presented as mean±standard.Statistical significance (P<0.05) compared with the LG is indicated by *, compared with the OG by #, and compared with the CG by ☆. | |||||||||
2.2 基因型和等位基因频率比较及HWE检验
对OR7D4、OR7E24、OR9Q1、SNCA中7个SNP基因型及其等位基因在各组人群中的频率分布情况进行比较(表 4)。各人群HWE检验显示,基因型频率分布均符合哈迪-温伯格平衡(PHWE>0.05)。其中,OR7E24 rs12608694、OR7E24 rs2240927、SNCA rs894278和OR7D4 rs8100858的基因型频率和等位基因频率在4个组间的分布无差异(P>0.05)。而OR9Q1 rs2245676和OR7D4 rs61729907的基因型和等位基因频率分布在长寿组和贺州组之间有明显差异(P<0.05)。OR9Q1 rs921134在子女组与贺州组之间的等位基因频率分布也存在差异性(P<0.05)。由结果可知,OR9Q1 rs2245676 T等位基因和OR7D4 rs61729907 G等位基因在长寿组中的占比最高,尤其与贺州组存在明显差异(P<0.05),故推测它们可能是长寿群体特有的遗传变异。
| 单核苷酸多态性 SNP |
组别(样本数) Group (Sample size) |
基因型 Genotype |
基因型数(占比/%) Number of genotype (Proportion/%) |
等位基因 Allele |
等位基因数(占比/%) Number of allele (Proportion/%) |
PHWE |
| OR7E24 rs12608694 | LG(283) | TT | 90(31.8) | T | 325(57.4) | 0.513 |
| TC/CC | 193(68.2) | C | 241(42.6) | |||
| OG(296) | TT | 90(30.4) | T | 335(56.6) | 0.324 | |
| TC/CC | 206(69.6) | C | 257(43.4) | |||
| CG(326) | TT | 93(28.6) | T | 355(54.4) | 0.450 | |
| TC/CC | 233(71.4) | C | 297(45.6) | |||
| HZ(286) | TT | 108(37.8) | T | 348(60.8) | 0.661 | |
| TC/CC | 178(62.2) | C | 224(39.2) | |||
| χ2 | 6.523 | 5.214 | ||||
| P | 0.089 | 0.157 | ||||
| OR7E24 rs2240927 | LG(283) | CC | 43(15.2) | T | 342(60.4) | 0.862 |
| CT/TT | 240(84.8) | C | 224(39.6) | |||
| OG(296) | CC | 39(13.2) | T | 356(60.1) | 0.071 | |
| CT/TT | 257(86.8) | C | 236(39.9) | |||
| CG(326) | CC | 53(16.3) | T | 388(59.5) | 1.000 | |
| CT/TT | 273(83.7) | C | 264(40.5) | |||
| HZ(286) | CC | 55(19.2) | T | 315(55.1) | 0.614 | |
| CT/TT | 231(80.8) | C | 257(44.9) | |||
| χ2 | 4.139 | 4.473 | ||||
| P | 0.247 | 0.215 | ||||
| OR9Q1 rs2245676 | LG* (283) | TT | 187(66.1) | T | 458(80.9) | 0.610 |
| TG/GG | 96(33.9) | G | 108(19.1) | |||
| OG(294) | TT | 192(65.3) | T | 469(79.8) | 0.106 | |
| TG/GG | 102(34.7) | G | 119(20.2) | |||
| CG(326) | TT | 197(60.4) | T | 506(77.6) | 0.926 | |
| TG/GG | 129(39.6) | G | 146(22.4) | |||
| HZ(285) | TT | 158(55.4) | T | 421(73.9) | 0.510 | |
| TG/GG | 127(44.6) | G | 149(26.1) | |||
| χ2 | 8.868 | 9.635 | ||||
| P | 0.031 | 0.022 | ||||
| OR9Q1 rs921134 | LG(283) | AA | 18(6.4) | G | 425(75.1) | 0.981 |
| AG/GG | 265(93.6) | A | 141(24.9) | |||
| OG▲(296) | AA | 15(5.1) | G | 475(80.2) | 0.269 | |
| AG/GG | 281(94.9) | A | 117(19.8) | |||
| CG(326) | AA | 24(7.4) | G | 486(74.5) | 0.468 | |
| AG/GG | 302(92.6) | A | 166(25.5) | |||
| HZ(286) | AA | 22(7.7) | G | 420(73.4) | 0.664 | |
| AG/GG | 264(92.3) | A | 152(26.6) | |||
| χ2 | 2.003 | 8.834 | ||||
| P | 0.572 | 0.032 | ||||
| SNCA rs894278 | LG(282) | TT | 138(48.9) | T | 388(68.8) | 0.250 |
| TG/GG | 144(51.1) | G | 176(31.2) | |||
| OG(296) | TT | 123(41.6) | T | 384(64.9) | 0.816 | |
| TG/GG | 173(58.4) | G | 208(35.1) | |||
| CG(326) | TT | 150(46.0) | T | 440(67.5) | 0.769 | |
| TG/GG | 176(54.0) | G | 212(32.5) | |||
| HZ(286) | TT | 118(41.3) | T | 367(64.2) | 1.000 | |
| TG/GG | 168(58.7) | G | 205(35.8) | |||
| χ2 | 4.806 | 3.698 | ||||
| P | 0.187 | 0.296 | ||||
| OR7D4 rs61729907 | LG* (274) | GG | 253(92.3) | G | 525(95.8) | 0.144 |
| GA/AA | 21(7.7) | A | 23(4.2) | |||
| OG(291) | GG | 252(86.6) | G | 542(93.1) | 1.000 | |
| GA/AA | 39(13.4) | A | 40(6.9) | |||
| CG△(322) | GG | 296(91.9) | G | 616(95.7) | 0.223 | |
| GA/AA | 26(8.1) | A | 28(4.3) | |||
| HZ(274) | GG | 232(84.7) | G | 501(91.4) | 0.071 | |
| GA/AA | 42(15.3) | A | 47(8.6) | |||
| χ2 | 12.86 | 13.62 | ||||
| P | 0.005 | 0.003 | ||||
| OR7D4 rs8100858 | LG(282) | CC | 267(94.7) | C | 549(97.3) | 1.000 |
| CT | 15(5.3) | T | 15(2.7) | |||
| OG(296) | CC | 281(94.9) | C | 577(97.5) | 1.000 | |
| CT | 15(5.1) | T | 15(2.5) | |||
| CG(325) | CC | 307(94.5) | C | 632(96.9) | 0.519 | |
| CT | 18(5.5) | T | 20(3.1) | |||
| HZ(285) | CC | 262(91.9) | C | 547(96.0) | 1.000 | |
| CT | 23(8.1) | T | 23(4.0) | |||
| χ2 | 2.986 | 2.676 | ||||
| P | 0.394 | 0.444 | ||||
| Note: * indicates that there are differences in genotype and allelic frequency distribution between the LG and the HZ (P<0.05); △ indicates that there are differences in genotype and allelic frequency distribution between the CG and the HZ (P<0.05);▲ indicates that there are differences in allele distribution groups between the OG and the HZ (P<0.05); PHWE indicates Hardy-Weinberg equilibrium test. | ||||||
2.3 SNP与CI风险的关联性分析
进一步对各位点建立4种遗传模型,采用非条件性Logistic回归分析研究不同组内各SNP与CI风险的相关性(表 5至表 7)。当优势比(OR)值>1且P < 0.05时,表示该基因型/等位基因会增加CI的风险;当OR值<1且P < 0.05时,表示该基因型/等位基因会降低CI的风险。在长寿组和当地组中,携带OR7E24 rs2240927 T等位基因的个体在多种遗传模型下显示罹患CI的风险较低,推测OR7E24 rs2240927 T等位基因及TT基因型影响受试人群寿命。在子女组中,携带OR9Q1 rs2245676 G等位基因的个体明显增加CI的风险(OR=2.191,95% CI=1.255-3.825,P=0.006),而携带OR9Q1 rs921134 G等位基因的个体明显降低CI的风险(OR=0.520,95% CI=0.294-0.919,P=0.024)。其他4个SNP与CI无关联。OR7E24 rs2240927、OR9Q1 rs2245676、OR9Q1 rs921134在其他组中的分析不具有统计学意义(P>0.05),故不进行展示。
| 组别 Group |
遗传模型 Genetic model |
基因分型 Genotyping |
认知正常 (占比/%) Cognitive normal (Proportion/%) |
认知障碍 (占比/%) Cognitive impairment (Proportion/%) |
优势比 (95%置信区间) OR(95% CI) |
P | PHWE |
| LG | Allelic model | C | 98(43.8) | 126(56.2) | 1 | PA=0.953 | |
| T | 188(55.0) | 154(45.0) | 0.637(0.454-0.894) | 0.009 | PB=0.804 | ||
| Codominant model | CC | 16(37.2) | 27(62.8) | 1 | |||
| CT | 66(47.8) | 72(52.2) | 0.712(0.337-1.506) | 0.374 | |||
| TT | 61(59.8) | 41(40.2) | 0.365(0.167-0.800) | 0.012 | |||
| Dominant model | CC | 16(37.2) | 27(62.8) | 1 | |||
| CT+TT | 127(52.9) | 113(47.1) | 0.538(0.265-1.095) | 0.087 | |||
| Recessive model | CC+CT | 82(45.1) | 99(54.9) | 1 | |||
| TT | 61(59.8) | 41(40.2) | 0.473(0.277-0.807) | 0.006 | |||
| CG | Allelic model | C | 200(75.8) | 64(24.2) | 1 | PA=0.717 | |
| T | 344(88.7) | 44(11.3) | 0.574(0.359-0.918) | 0.021 | PB=0.702 | ||
| Codominant model | CC | 33(62.3) | 20(37.7) | 1 | |||
| CT | 134(84.8) | 24(15.2) | 0.463(0.204-1.051) | 0.066 | |||
| TT | 105(91.3) | 10(8.7) | 0.328(0.130-0.827) | 0.018 | |||
| Dominant model | CC | 33(62.3) | 20(37.7) | 1 | |||
| CT+TT | 239(87.5) | 34(12.5) | 0.404(0.188-0.866) | 0.020 | |||
| Recessive model | CC+CT | 167(79.1) | 44(20.9) | 1 | |||
| TT | 105(91.3) | 10(8.7) | 0.555(0.259-1.193) | 0.131 | |||
| Note: OR and P value are the value after adjustment for covariables of gender, age, BMI, SBP, TG and LDL-C.PA is the HWE test value of the cognitive normal group; PB is the HWE test value of the cognitive impairment group. | |||||||
| 组别 Group |
遗传模型 Genetic model |
基因分型 Genotyping |
认知正常(占比/%) Cognitive normal (Proportion/%) |
认知障碍(占比/%) Cognitive impairment (Proportion/%) |
优势比 (95%置信区间) OR(95% CI) |
P | PHWE |
| OG | Allelic model | T | 425(90.6) | 44(9.4) | 1 | PA=0.150 | |
| G | 97(81.5) | 22(18.5) | 2.191(1.255-3.825) | 0.006 | PB=0.952 | ||
| Codominant model | TT | 177(92.2) | 15(7.8) | 1 | |||
| TG | 71(83.5) | 14(16.5) | 2.704(1.175-6.224) | 0.019 | |||
| GG | 13(76.5) | 4(23.5) | 2.681(0.739-9.724) | 0.134 | |||
| Dominant model | TT | 177(92.2) | 15(7.8) | 1 | |||
| TG+GG | 84(82.4) | 18(17.6) | 2.698(1.252-5.814) | 0.011 | |||
| Recessive model | TT+TG | 248(89.5) | 29(10.5) | 1 | |||
| GG | 13(76.5) | 4(23.5) | 2.234(0.628-7.947) | 0.214 | |||
| Note: OR and P value are the value after adjustment for covariables of gender, age, BMI, SBP, TG and LDL-C.PA is the HWE test value of the cognitive normal group; PB is the HWE test value of the cognitive impairment group. | |||||||
| 组别 Group |
遗传模型 Genetic model |
基因分型 Genotyping |
认知正常(占比/%) Cognitive normal (Proportion/%) |
认知障碍(占比/%) Cognitive impairment (Proportion/%) |
优势比 (95%置信区间) OR(95% CI) |
P | PHWE |
| OG | Allelic model | A | 97(82.9) | 20(17.1) | 1 | PA=0.935 | |
| G | 429(90.3) | 46(9.7) | 0.520(0.294-0.919) | 0.024 | PB=0.235 | ||
| Codominant model | AA | 9(60.0) | 6(40.0) | 1 | |||
| AG | 79(90.8) | 8(9.2) | 0.106(0.023-0.490) | 0.004 | |||
| GG | 175(90.2) | 19(9.8) | 0.127(0.033-0.492) | 0.003 | |||
| Dominant model | AA | 9(60.0) | 6(40.0) | 1 | |||
| AG+GG | 254(90.4) | 27(9.6) | 0.123(0.032-0.468) | 0.002 | |||
| Recessive model | AA+AG | 88(86.3) | 14(13.7) | 1 | |||
| GG | 175(90.2) | 19(9.8) | 0.687(0.315-1.497) | 0.345 | |||
| Note: OR and P value are the value after adjustment for covariables of gender, age, BMI, SBP, TG and LDL-C.PA is the HWE test value of the cognitive normal group; PB is the HWE test value of the cognitive impairment group. | |||||||
2.4 SNP连锁不平衡分析
为了解各基因SNP间的内在交互作用,对OR7D4、OR7E24、OR9Q1的SNP进行连锁不平衡分析(图 1)。本研究中SNCA基因仅rs894278一个位点,无法构成单倍型,故不纳入分析。结果显示,在长寿组中的OR7E24 rs12608694和OR7E24 rs2240927(D′=0.97)之间存在强连锁不平衡状态。
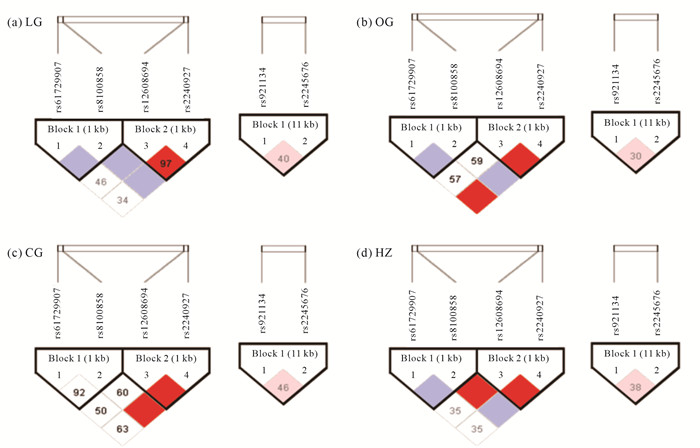
|
| The unit of the value D′ on the color block is 10-2.The darker the red, meaning the linkage disequilibrium relationship between the two loci is more closely related.White and gray: there is no linkage disequilibrium relationship between the two loci. 图 1 各组SNP的基因连锁不平衡情况 Fig.1 Linkage disequilibrium of SNP in each group |
2.5 单倍型与CI风险的关联性分析
进一步对各SNP构成的单倍型进行分析,了解内在交互作用对认知功能的影响(表 8)。经性别、年龄等协变量校正后发现,在长寿组和当地组中,携带OR7E24 (rs12608694、rs2240927) T-C单倍型的个体患CI的风险更高,尤其在长寿组中,携带OR7E24 (rs12608694、rs2240927) T-C单倍型的个体患CI的风险显著增加(OR=1.659,95% CI=1.155-2.389,P=0.006)。而当地组中OR7E24(rs12608694、rs2240927) C-T单倍型降低了CI的发病风险。在子女组中,携带OR9Q1 (rs921134、rs2245676) G-T单倍型会降低CI的发病风险(OR=0.460,95% CI=0.267-0.797,P=0.005),而OR9Q1 (rs921134、rs2245676) A-G的携带者患CI的风险更高(OR=2.198,95% CI=1.011-4.520,P=0.038)。OR7D4 (rs61729907、rs8100858) G-C单倍型会增加长寿组、当地组患CI的风险。
| 基因 Gene |
组别 Group |
单倍型 Haplotype |
认知正常 (占比/%) Cognitive normal (Proportion/%) |
认知障碍 (占比/%) Cognitive impairment (Proportion/%) |
优势比 (95%置信区间) OR(95% CI) |
P |
| OR7E24 | LG | T-C | 125(44.8) | 97(34.0) | 1.659(1.155-2.389) | 0.006 |
| C-T | 109(39.1) | 130(45.6) | 0.729(0.507-1.045) | 0.086 | ||
| T-T | 45(16.1) | 58(20.4) | 0.738(0.464-1.168) | 0.196 | ||
| OG | T-T | 9(13.6) | 90(17.1) | 0.818(0.359-1.683) | 0.605 | |
| T-C | 30(45.5) | 206(39.2) | 1.139(0.663-1.946) | 0.633 | ||
| C-T | 27(40.9) | 230(43.7) | 0.974(0.561-1.670) | 0.925 | ||
| CG | T-C | 64(59.2) | 200(36.8) | 2.697(1.711-4.297) | 0.001 | |
| C-T | 33(30.6) | 264(48.5) | 0.463(0.283-0.741) | 0.002 | ||
| T-T | 11(10.2) | 80(14.7) | 0.565(0.253-1.131) | 0.131 | ||
| HZ | T-C | 47(42.0) | 210(45.7) | 0.896(0.574-1.393) | 0.627 | |
| C-T | 48(42.9) | 176(38.3) | 1.116(0.714-1.735) | 0.627 | ||
| T-T | 17(15.2) | 74(16.1) | 1.002(0.533-1.810) | 0.994 | ||
| OR9Q1 | LG | G-T | 187(66.8) | 196(68.5) | 0.906(0.618-1.325) | 0.610 |
| G-G | 23(8.2) | 19(6.6) | 1.174(0.592-2.361) | 0.646 | ||
| A-G | 33(11.8) | 33(11.5) | 1.115(0.638-1.955) | 0.702 | ||
| A-T | 37(13.2) | 38(13.3) | 0.997(0.588-1.688) | 0.992 | ||
| OG | G-T | 36(54.5) | 379(72.1) | 0.460(0.267-0.797) | 0.005 | |
| A-G | 12(18.2) | 47(8.9) | 2.198(1.011-4.520) | 0.038 | ||
| G-G | 10(15.1) | 50(9.5) | 1.823(0.797-3.876) | 0.133 | ||
| A-T | 8(12.1) | 50(9.5) | 1.331(0.547-2.910) | 0.497 | ||
| CG | A-T | 14(13.0) | 57(10.5) | 1.480(0.753-2.754) | 0.233 | |
| A-G | 13(12.0) | 82(15.1) | 0.747(0.359-1.430) | 0.404 | ||
| G-G | 7(6.5) | 44(8.1) | 0.944(0.374-2.075) | 0.894 | ||
| G-T | 74(68.5) | 361(66.4) | 0.974(0.611-1.576) | 0.912 | ||
| HZ | A-G | 11(9.8) | 78(17.0) | 0.531(0.252-1.033) | 0.0766 | |
| A-T | 16(14.3) | 47(10.2) | 1.306(0.672-2.435) | 0.414 | ||
| G-T | 73(65.2) | 287(62.4) | 1.117(0.712-1.772) | 0.632 | ||
| G-G | 12(10.7) | 48(10.4) | 1.184(0.560-2.356) | 0.643 | ||
| OR7D4 | LG | G-C | 269(96.1) | 259(90.6) | 2.343(1.125-5.173) | 0.027 |
| A-C | 7(2.5) | 16(5.6) | 0.455(0.166-1.138) | 0.103 | ||
| G-T | 4(1.4) | 11(3.8) | 0.410(0.108-1.302) | 0.150 | ||
| OG | A-C | 8(12.1) | 32(6.1) | 1.936(0.752-4.534) | 0.145 | |
| G-C | 56(84.8) | 481(91.4) | 0.596(0.281-1.364) | 0.195 | ||
| G-T | 2(3.0) | 13(2.5) | 1.064(0.161-4.112) | 0.937 | ||
| CG | G-C | 104(96.2) | 500(90.1) | 4.383(1.295-27.413) | 0.046 | |
| G-T | 2(1.9) | 18(3.3) | 0.619(0.095-2.312) | 0.536 | ||
| A-C | 2(1.9) | 26(4.8) | 1.215(NA) | 0.983 | ||
| HZ | G-C | 94(83.9) | 408(88.7) | 0.684(0.374-1.294) | 0.229 | |
| G-T | 6(5.3) | 17(3.7) | 1.575(0.522-4.270) | 0.390 | ||
| A-C | 12(10.7) | 35(7.6) | 1.362(0.633-2.771) | 0.408 | ||
| Note: haplotypes composition method: OR7E24 (rs12608697-rs2240927); OR9Q1 (rs921134-rs2245676); OR7D4 (rs61729907-rs8100858).The meaning of the data in brackets is the percentage of the total number of haplotypes. | ||||||
2.6 长寿组SNP-SNP及基因-环境间的交互作用对认知水平的影响
使用GMDR来评估长寿组SNP-SNP、基因-环境间的交互作用对认知水平的影响(表 9)。结果发现长寿组中SNP-SNP间存在二阶交互作用(P<0.05),其中OR7E24 rs2240927、SNCA rs894278、OR7E24 rs12608694的三基因座模型测试准确度为59.70%,交叉验证一致性为10/10。SNP-环境交互中可见OR7D4 rs61729907、olfactory(嗅觉)、WHR三基因座模型,交叉验证一致性为7/10,测试准确度为65.31%。单倍型与环境因素之间存在二、四基因座的交互模型,OR7D4 (rs61729907、rs8100858) G-C、嗅觉两因素一阶交互效果最优,交叉验证一致性为10/10,测试准确度为67.37%。
| 组合方式 Combination mode |
训练模型准确度/% Training model accuracy/% |
验证模型准确度/% Testing model accuracy/% |
交叉验证一致性 CV consistency |
P | |
| SNP-SNP | rs2240927 & rs894278 | 60.45 | 55.63 | 9/10 | 0.171 9 |
| rs2240927 & rs894278 & rs12608694 | 69.94 | 59.70 | 10/10 | 0.010 7 | |
| SNP-Environment | rs61729907 & olfactory & WHR | 70.91 | 65.31 | 7/10 | 0.010 7 |
| Haplotype-Environment | OR7D4 G-C & olfactory | 67.63 | 67.37 | 10/10 | 0.001 0 |
| OR7D4 G-C & olfactory & TG & HDL-C | 70.45 | 64.98 | 6/10 | 0.001 0 | |
| Note: the values of each variable in the interaction are as follows: olfactory test score>10 points; WHR, for men ≥0.90, for women ≥0.85;TG≥1.7 mmol/L; HDL-C<1.29 mmol/L (for women), <1.04 mmol/L (for men). | |||||
进一步运用多因子降维分析方法对各组合方式的最佳交互模型构建交互作用的树状图,以此来判断交互作用的强弱情况。如图 2所示,在长寿组SNP-SNP交互作用中,SNCA rs894278、OR7E24 rs12608694具有最强的协同交互作用;OR7D4 rs61729907、嗅觉的强拮抗作用可以在SNP-环境中被发现;从单倍型-环境的树状图可以看出OR7D4 (rs61729907、rs8100858) G-C、嗅觉之间具有强拮抗作用。
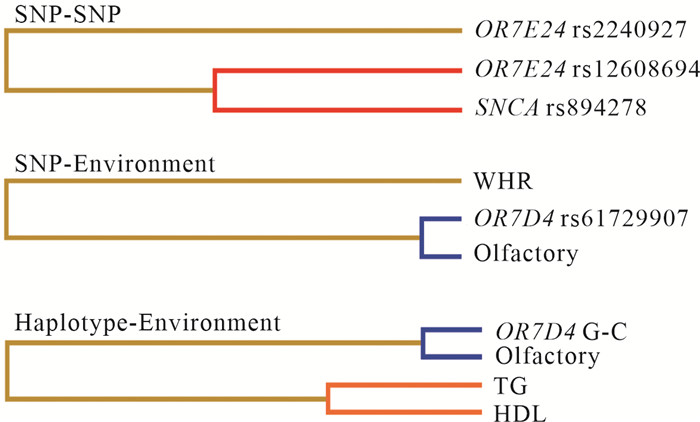
|
| The stronger the interaction in the dendrogram, the closer the distance, the farther the interaction, the weaker the interaction.Red means the two have a strong synergistic interaction, orange and brown means the weaker synergistic interaction; blue means the two have a strong antagonistic effect. 图 2 长寿组SNP-SNP、基因-环境交互作用树状图 Fig.2 Dendrogram of the interaction between SNP-SNP and gene-environment with the LG |
3 讨论
嗅觉功能障碍是阿尔茨海默病、帕金森病等神经退行性疾病的早期迹象之一,并且嗅觉能力会随着年龄的增长而下降[10]。嗅觉和认知的联系与外周嗅觉系统神经元同认知相关的皮质区域直接联系有关,嗅觉受损往往伴随着大脑区域体积的损失[14-15]。有研究发现,进行嗅觉功能恢复还有助于改善记忆[16-17]。OR7D4、OR7E24、OR9Q1同属于嗅觉受体家族的基因,OR7D4 rs61729907与雄烯酮嗅觉敏感性的差异有关,同时发现位于OR7D4上的rs2878329与肥胖、认知饮食限制和饥饿水平降低存在相关性[18-21]。嗅觉可以通过直接影响摄食行为改变饮食模式,从而有可能造成包括CI在内的一系列代谢性疾病的发生[22-23]。OR9Q1上的SNP则与气味诱导的过敏性鼻炎之间存在联系[24]。Luo等[25]还发现携带SNCA rs894278 G等位基因个体的嗅觉功能障碍风险降低。但是迄今为止,这类嗅觉受体基因上的SNP与认知功能的相关性研究鲜有报道。本研究对嗅觉受体基因OR7D4、OR7E24、OR9Q1和SNCA上的SNP进行基因分型,分析嗅觉相关的SNP与认知功能的相关性,以进一步探索嗅觉影响认知功能的机制,以期为CI的防治提供参考。
根据美国国家生物技术信息中心公布的数据,中国北京汉族人群OR9Q1 rs2245676 G、OR9Q1 rs921134 A、OR7D4 rs61729907 A、OR7D4 rs8100858 T的分布频率明显高于本次研究的壮族人群,OR7E24 rs12608694 C的分布频率明显低于本研究人群。另外,7个SNP的等位基因频率在本研究人群与欧洲人群中的分布情况均存在明显差异,其中欧洲人群的OR7D4 rs61729907 A、OR7D4 rs8100858 T、OR9Q1 rs2245676 G的分布频率要明显高于本研究人群;OR7E24 rs12608694 C、OR7E24 rs2240927 T、OR9Q1 rs921134 G、SNCA rs894278 G的分布频率要明显低于本研究人群。以上研究结果也说明此类差异具有地域性和群体特性。OR7E24 rs2240927、OR9Q1 rs2245676、OR9Q1 rs921134与红水河流域的长寿组、子女组、当地组的CI发病风险有关,这种相关性尤其体现在长寿组及子女组中。此外,单倍型分析的结果显示,OR7D4 (rs61729907、rs8100858) G-C、OR7E24 (rs12608694、rs2240927) T-C会增加长寿人群的CI发病风险;子女组中携带OR9Q1 (rs921134、rs2245676) G-T会降低CI发病风险,而OR9Q1 (rs921134、rs2245676) A-G会增高风险。由此可见,嗅觉相关SNP与红水河流域老年人群的认知功能存在相关性,并且这种相关性在长寿组及子女组中更为显著。而基因型频率分布情况以及产生的效应在红水河流域长寿家系(长寿组及子女组)与当地组之间,以及长寿家系(长寿组及子女组)与贺州组之间都存在差异。
除遗传学因素影响外,认知功能障碍也受环境因素的影响,例如身高、体重、血脂水平、空腹血糖水平等。Vitali等[26]研究发现高密度脂蛋白(HDL)水平与CI以及各种神经退行性疾病相关。广西红水河流域地区是著名的“长寿之乡”,并且鲜与外族通婚,遗传背景单一,是研究基因-环境交互的理想之地。本研究在红水河流域长寿人群基因-环境交互作用的分析中发现,OR7E24 rs2240927、SNCA rs894278、OR7E24 rs12608694之间对CI的影响存在协同的交互作用;嗅觉、WHR、OR7D4 rs61729907以及OR7D4 (rs61729907、rs8100858) G-C、嗅觉、TG、HDL-C之间对CI的影响存在基因-环境交互作用。其中,嗅觉正常(olfactory>10)与OR7D4 (rs61729907、rs8100858) G-C之间存在拮抗交互作用,嗅觉正常是CI的保护因素,而OR7D4 (rs61729907、rs8100858) G-C可以增加长寿人群患CI的风险。另外TG和HDL-C之间显示为协同交互作用,表明正常的血脂水平对降低CI的发病风险具有重要意义。在红水河流域长寿人群中,SNP-SNP、基因-环境交互作用影响CI的发生,且主要环境因素为血脂水平和嗅觉功能,保持正常的嗅觉功能和血脂水平会降低长寿人群CI发生的风险。
4 结论本研究通过对广西红水河流域和贺州市八步区南乡镇老年人群中的嗅觉受体基因OR7D4、OR7E24、OR9Q1和SNCA上的7个SNP进行基因分型,并探讨SNP-SNP以及基因-环境交互作用对认知衰老的影响。嗅觉相关SNP的基因型和等位基因在广西红水河流域长寿人群与贺州组之间的频率分布存在差异,并且与广西红水河流域长寿人群的认知功能障碍相关,且与CI发病的关联性主要见于广西红水河流域长寿组和子女组。防止嗅觉功能损害,减少认知功能障碍的发生,可能有助于减缓衰老进程,促进健康长寿。但目前基因-环境间的交互作用影响认知衰老发生的生物学机制仍不明确,未来有待开展更多的研究加以探索。
| [1] |
赵明, 戚立权, 董明英. 中老年人无认知障碍预期寿命研究[J]. 中国人口科学, 2023, 37(2): 52-67. |
| [2] |
BRAUN T, DOERR J M, PETERS L, et al. Age-related changes in oral sensitivity, taste and smell[J]. Scientific Reports, 2022, 12(1): 1533. DOI:10.1038/s41598-022-05201-2 |
| [3] |
WANG J, SUN X, YANG Q X. Early aging effect on the function of the human central olfactory system[J]. The Journals of Gerontology Series A, Biological Sciences and Medical Sciences, 2017, 72(8): 1007-1014. |
| [4] |
DONG Y, WANG Y X, LIU K K, et al. Olfactory impairment among rural-dwelling Chinese older adults: prevalence and associations with demographic, lifestyle, and clinical factors[J]. Frontiers in Aging Neuroscience, 2021, 13: 621619. DOI:10.3389/fnagi.2021.621619 |
| [5] |
LIU D M, LU J M, WEI L P, et al. Olfactory deficit: a potential functional marker across the Alzheimer's disease continuum[J]. Frontiers in Neuroscience, 2024, 18: 1309482. DOI:10.3389/fnins.2024.1309482 |
| [6] |
CARLSON H, LEITÃO J, DELPLANQUE S, et al. Sustained effects of pleasant and unpleasant smells on resting state brain activity[J]. Cortex: a Journal Devoted to the Study of the Nervous System and Behavior, 2020, 132: 386-403. DOI:10.1016/j.cortex.2020.06.017 |
| [7] |
BATHINI P, BRAI E, AUBER L A. Olfactory dysfunction in the pathophysiological continuum of dementia[J]. Ageing Research Reviews, 2019, 55: 100956. DOI:10.1016/j.arr.2019.100956 |
| [8] |
BRAI E, HUMMEL T, ALBERI L. Smell, an underrated early biomarker for brain aging[J]. Frontiers in Neuroscience, 2020, 14: 792. DOI:10.3389/fnins.2020.00792 |
| [9] |
SHADYAB A H, LACROIX A Z. Genetic factors associated with longevity: a review of recent findings[J]. Ageing Research Reviews, 2015, 19: 1-7. DOI:10.1016/j.arr.2014.10.005 |
| [10] |
FATUZZO I, NICCOLINI G F, ZOCCALI F, et al. Neurons, nose, and neurodegenerative diseases: olfactory function and cognitive impairment[J]. International Journal of Molecular Sciences, 2023, 24(3): 2117. DOI:10.3390/ijms24032117 |
| [11] |
黎海燕. 广西巴马地区壮族长寿群体认知状况调查及认知相关基因多态性的相关性研究[D]. 南宁: 广西医科大学, 2016.
|
| [12] |
LI H Z, JIA J P, YANG Z Q. Mini-mental state examination in elderly Chinese: a population-based normative study[J]. Journal of Alzheimer's Disease, 2016, 53(2): 487-496. DOI:10.3233/JAD-160119 |
| [13] |
HUANG L, PENG J H, LIANG Q H, et al. Insulin-like factor-2 receptor rs9456497 G genotype overrepresents in males of average population and its correlation with cardiovascular risks[J]. Archives of Gerontology and Geriatrics, 2018, 76: 202-209. DOI:10.1016/j.archger.2018.03.004 |
| [14] |
LEON M, WOO C. Environmental enrichment and successful aging[J]. Frontiers in Behavioral Neuroscience, 2018, 12: 155. DOI:10.3389/fnbeh.2018.00155 |
| [15] |
YAO L, PINTO J M, YI X, et al. Gray matter volume reduction of olfactory cortices in patients with idiopathic olfactory loss[J]. Chemical Senses, 2014, 39(9): 755-760. DOI:10.1093/chemse/bju047 |
| [16] |
JOHNSON A J. Cognitive facilitation following intentional odor exposure[J]. Sensors, 2011, 11(5): 5469-5488. DOI:10.3390/s110505469 |
| [17] |
TSUSHIMA Y, NISHINO Y, ANDO H. Olfactory stimulation modulates visual perception without training[J]. Frontiers in Neuroscience, 2021, 15: 642584. DOI:10.3389/fnins.2021.642584 |
| [18] |
CHOQUETTE A C, BOUCHARD L, DRAPEAU V, et al. Association between olfactory receptor genes, eating behavior traits and adiposity: results from the Quebec family study[J]. Physiology & Behavior, 2012, 105(3): 772-776. |
| [19] |
KELLER A, ZHUANG H, CHI Q, et al. Genetic variation in a human odorant receptor alters odour perception[J]. Nature, 2007, 449(7161): 468-472. DOI:10.1038/nature06162 |
| [20] |
LUNDE K, EGELANDSDAL B, SKUTERUD E, et al. Genetic variation of an odorant receptor OR7D4 and sensory perception of cooked meat containing androstenone[J]. PLoS One, 2012, 7(5): e35259. DOI:10.1371/journal.pone.0035259 |
| [21] |
CHOI Y, YOON M. The expression of androstenone receptor (OR7D4) in vomeronasal organ and olfactory epithelium of horses[J]. Domestic Animal Endocrinology, 2021, 74: 106535. DOI:10.1016/j.domaniend.2020.106535 |
| [22] |
ROLLS E T. Taste and smell processing in the brain[J]. Handbook of Clinical Neurology, 2019, 164: 97-118. |
| [23] |
BRATMAN G N, BEMBIBRE C, DAILY G C, et al. Nature and human well-being: the olfactory pathway[J]. Science Advances, 2024, 10(20): eadn3028. DOI:10.1126/sciadv.adn3028 |
| [24] |
BERNSTEIN J A, ZHANG G, JIN L, et al. Olfactory receptor gene polymorphisms and nonallergic vasomotor rhinitis[J]. The Journal of Asthma, 2008, 45(4): 287-292. DOI:10.1080/02770900701867579 |
| [25] |
LUO N D, LI Y Y, NIU M Y, et al. Variants in the SNCA locus are associated with the progression of Parkinson's disease[J]. Frontiers in Aging Neuroscience, 2019, 11: 110. DOI:10.3389/fnagi.2019.00110 |
| [26] |
VITALI C, WELLINGTON C L, CALABRESI L. HDL and cholesterol handling in the brain[J]. Cardiovascular Research, 2014, 103(3): 405-413. DOI:10.1093/cvr/cvu148 |



